Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.28 no.4 Lima oct./dic. 2017
http://dx.doi.org/10.15381/rivep.v28i4.13885
ARTÍCULOS PRIMARIOS
Aislamiento y detección molecular de cepas emergentes del virus de la diarrea epidémica porcina en Lima, Perú
Isolation and molecular detection of emerging porcine epidemic diarrhea virus strains in Lima, Peru
Gina Castro-Sanguinetti1, Mercy Ramírez V.1,2, Juan More B.1, Alberto Manchego S.1, Hermelinda Rivera G.1
1 Laboratorio de Microbiología y Parasitología Veterinaria, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima, Perú
2 E-mail: mramirezv@unmsm.edu.pe
RESUMEN
En 2013 se observaron cuadros clínicos sugerentes a PEDv en granjas porcinas de Lima y Arequipa llegando hasta el 100% de mortalidad en lechones y con resultados negativos a otros agentes virales como peste porcina clásica (PPCv) y gastroenteritis transmisible (TGEv). Por esta razón, el objetivo del trabajo fue aislar y detectar molecularmente cepas del PEDv en granjas porcinas de Lima. Se colectaron 37 muestras de heces y contenido intestinal de lechones entre 2 y 21 días de edad con cuadros clínicos sugerentes a PEDV procedentes de granjas porcinas del departamento de Lima, Perú. El 94.3% (35/37) resultaron positivos al test de inmunocromatografía (IHC). El 97.1% (34/35) de estos fueron confirmados por RT-PCR en tiempo real que amplifica un segmento de 101 pb del gen ORF3 del PEDv. El control y las muestras positivas mostraron un ciclo umbral (Ct) entre 10 y 21 ciclos con una temperatura de disociación (Tm) de 77.7 °C. No hubo amplificación de TGEv ni PPCv utilizados como controles negativos. Todas las muestras positivas fueron procesadas para el aislamiento en la línea celular VERO-21 suplementado con 20 µg/ml tripsina. El 23.5% (8/34) de las células mostraron formas redondeadas y lisis en el cultivo celular entre el primer y segundo pasaje; sin embargo, solo la mitad (4/8) fueron confirmadas por RT-PCR en tiempo real. Este trabajo representa el primer aislamiento del PEDv en el Perú utilizando IHC y confirmado por RT-PCR en tiempo real.
Palabras clave: virus de la diarrea epidémica porcina; PEDv; aislamiento viral; RT-PCR en tiempo real; cultivo celular
ABSTRACT
In 2013, clinically suggestive cases of PEDv were reported in pig farms in Lima and Arequipa, reaching up to 100% mortality in piglets and negative results to other viral agents such as classical swine fever (CPPv) and transmissible gastroenteritis (TGEv). For this reason, the aim of this study was to isolate and molecularly detect PEDv strains in Lima pig farms. A total of 37 stool samples and intestinal contents of piglets between 2 and 21 days of age with clinical signs suggestive of PEDv from pig farms of the department of Lima, Peru were collected. Results showed that 94.3% (35/37) were positive by the immunochromatography (IHC) test; 97.1% (34/35) of them were confirmed by RT-PCR real-time that amplified a 101-bp segment of the ORF3 gene of the PEDv. The control and positive samples showed a threshold cycle (Ct) between 10 and 21 cycles with a melting temperature (Tm) of 77.7 °C. There was no amplification of TGEv or PPCv used as negative controls. All positive samples were processed for isolation in the VERO-21 cell line supplemented with 20 µg/ml trypsin. The 23.5% (8/34) of the inoculated cells showed rounded shapes and lysis in cell culture between the first and second passage; however, only half (4/8) were confirmed by RT-PCR real-time. This work represents the first isolation of PEDv in Peru using IHC and confirmed by RT-PCR real-time.
Keywords: porcine epidemic diarrhea virus; PEDv; virus isolation; RT-PCR real-time; cell culture
INTRODUCCIÓN
El virus de la diarrea epidémica porcina (PEDv, por sus siglas en inglés) es un miembro del género Alphacoronavirus de la familia Coronaviridae, que posee un genoma ARN de una sola cadena, polaridad positiva y con envoltura (Song y Park, 2012; Zhao et al., 2014; Hu et al., 2015). Este virus causa la diarrea epidémica porcina (PED), enfermedad gastroentérica, aguda y contagiosa, caracterizada por diarrea acuosa, vómitos, deshidratación y pérdida de peso, con alta mortalidad en lechones lactantes menores de tres semanas (Kim y Chae, 2000). Otro virus porcino que incluye este género es gastroenteritis transmisible (TGEv), cursa con cuadros clínicos similares a PED como resultado de la replicación viral en los enterocitos diferenciados de la vellosidad intestinal, principalmente yeyuno e íleon, y que conduce a atrofia de las vellosidades y a la diarrea por mala absorción (Jung et al., 2014; Lin et al., 2015).
El genoma del PEDv es de 28 Kb aproximadamente y posee siete marcos de lecturas abiertas (ORFs, ‘open reading frame’) que codifican cuatro proteínas estructurales: espícula (Spike, S), envoltura (E), membrana (M) y la nucleocápsida (N), además de tres proteínas no estructurales (replicasa 1a y 1b) y el ORF3 (Song y Park, 2012). El gen S es el mayor gen estructural del genoma del PEDv, responsable de la unión al receptor celular, la aminopeptidasa N (APN) e inductor de la formación de anticuerpos neutralizantes y de la fusión celular (Li et al., 2012; Jung y Saif, 2015). El gen S es utilizado también como blanco para determinar la variabilidad genética del virus.
El únicohospederonatural conocidoes el cerdo doméstico (Sus scrofa domesticus). Los animales son afectados en cualquier edad; sin embargo, los lechones lactantes (menores de 10 días) son los más susceptibles, ocasionando 90-100% de mortalidad. Estos se infectan por la vía oral con heces de animales infectados en estadios temprano de la enfermedad. Los fómites juegan un rol importante para la diseminación del virus entre granjas (OIE, 2014).
La línea celular VERO (células epiteliales del riñón del mono verde africano -Chlorocebus sp) se utiliza para el aislamiento del virus bajo condiciones de laboratorio. Sin embargo, la propagación exitosa del virus depende de la adecuada suplementación de una proteasa exógena como tripsina en el medio de cultivo celular que permita cortar a la proteína S en dos subunidades y hacerla infectiva (Shirato et al., 2011). Los efectos citopáticos que se describen en la literatura son vacuolización, formación de sincitios y lisis de la célula (Song y Park, 2012; Suzuki et al., 2015).
Las técnicas de diagnóstico que se utilizan son la inmunofluorescencia directa, diversos tipos de ELISA (Sozzi et al., 2010) e hibridación in situ, siendo la prueba confirmatoria el aislamiento viral utilizando la línea celular VERO 21 (‘African Green Monkey Kidney cell line’) (Pan et al., 2012) y la técnica de RT-PCR en tiempo real capaz de detectar pequeñas cantidades de material genético, tanto como 101-102 DICT50/ ml (Kim et al., 2001; Song et al., 2006). Los cebadores son diseñados teniendo como blanco a los genes S, N y ORF3 del PEDV (Susuki et al., 2015).
En el Perú, entre 2013 y comienzos de 2014 se observaron brotes de diarreas agudas severas, vómitos y deshidratación en lechones, marranas y cerdos de engorde, ocasionando el 90-100% de mortalidad en los lechones. En octubre de 2013, el Servicio Nacional de Sanidad Agraria (SENASA), a través de su boletín epidemiológico (SENASA, 2013), reporta la presencia del PEDv en el país en un trabajo conjunto con el Laboratorio de Virología de la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos a través de la prueba molecular RT-PCR en tiempo real, pero sin estudios de aislamiento viral. Por esta razón, el objetivo del presente trabajo fue aislar e identificar molecularmente cepas emergentes del PEDv en granjas porcinas de Lima.
El estudio se llevó a cabo en las instalaciones del Laboratorio de Virología de la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos (FMVUNMSM) en Lima, Perú.
Se utilizaron 37 muestras de contenido intestinal y heces diarreicas de lechones menores de tres semanas de edad, cuya historia clínica describió cuadros entéricos compatibles con los dos coronavirus más comunes, PED y TGE (Figura 1). Todas las muestras fueron obtenidas de animales recientemente muertos o sacrificados en sus respectivas granjas y remitidas al laboratorio entre 2013 y 2014. Las nuestras provinieron de seis granjas tecnificadas ubicadas en Chilca (n=8), Chincha (n=1), Pachacamac (n=11), Ventanilla (n=9), San Juan de Miraflores (n=5) y Huacho (n=3) del departamento de Lima. Las muestras fueron evaluadas mediante la técnica de inmunocromatografía (IHC) con un kit comercial (Anigen Rapid TGE/PED Ag Test Kit, Bionote, Corea), que permite diferenciar ambos agentes virales.
Las muestras de contenido intestinal (yeyuno e íleon) y de heces diarreicas (~5 ml) fueron diluidas en PBS pH 7.4 estéril, para lograr una suspensión aproximada del 10%. Posteriormente se homogenizaron en vórtex y centrifugaron a 1500 g por 7 min; se colectó el sobrenadante y se filtró mediante filtros de jeringa estériles de 0.45 µm. Las muestras fueron almacenadas a -70 °C.
La confirmación de la presencia de PEDv se llevó a cabo mediante un RT-PCR dúplex en tiempo real, para la detección en simultáneo de PEDv y TGEv. La extracción y purificación de ARN se llevó a cabo de acuerdo con las especificaciones del kit comercial ‘QIAamp Viral RNA Mini Kit’ (Qiagen, EEUU). Los productos obtenidos fueron almacenados a -70 °C hasta su utilización.
La técnica de RT-PCR en tiempo real se llevó a cabo en dos pasos, utilizando el kit comercial ‘GoTaq® 2-Step RT-qPCR System’ (Promega, EEUU), y utilizando el termociclador MJ Research PTC-200 adosado al lector de fluorescencia Chrome 4 (BioRad, EEUU), siguiendo las indicaciones del fabricante. Se utilizaron cebadores específicos descritos por Wang et al. (2014) y Kwon et al. (2013). Los cebadores para PEDv reconocen un segmento conservado de 101 bases nucleotídicas del gen que codifica el ORF-3 (Forward 5’-GCACTTATTGGCAGGCTTTGT-3’; Reverse 5’-CCATTGAGAAAAGAAAGTGTCGTAG-3’). Los cebadores utilizados para TGEv reconocen un segmento conservado de 109 bases nucleotídicas del gen que codifica la proteína de la espícula (proteína S) (Forward 5’GCTTGATGAATTGAGTGCTGATG-3’; Reverse 5’CCTAACC-TCGGCTTGTCTGG-3’). Como control positivo de PEDv se utilizó una secuencia de ADN complementario (ADNc) cedida por el SENASA del Perú, proveniente de USDA-APHIS. Asimismo, para TGEv se utilizó la cepa Holland (SVANOVA, Suecia). Como control negativo se utilizó una cepa de virus de la peste porcina clásica (PPC), Cepa Allfort/187, genotipo 1, donado por el Dr. J. Pasick, Canadian Food InspectionAgency. Las muestras positivas se mantuvieron congeladas hasta su procesamiento para el aislamiento viral.
Para el aislamiento viral se inoculó en la línea celular VERO-21 en placas de 24 pozos con medio esencial mínimo (MEM) suplementado con 10% de suero fetal bovino (FBS) y 1% de antibiótico. Para el aislamiento se preparó medio de mantenimiento que consistió en medio MEM suplementado con 3% de suero fetal bovino, antibiótico y tripsina en concentración de 20 µg/ml. Para la inoculación se retiró el medio de crecimiento, se realizó dos lavados con suero fisiológico/PBS/ medio MEM, y se inocularon 200 µl de la muestra filtrada en la monocapa (en duplicado) y medio de mantenimiento.
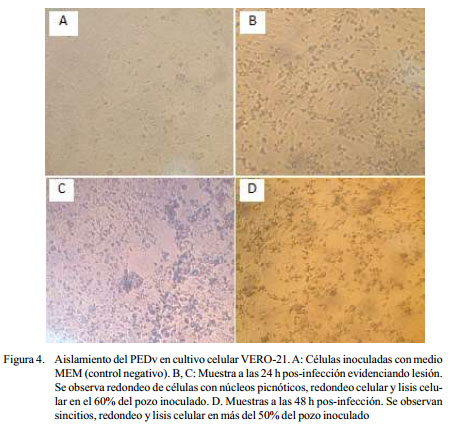
Los cultivos inoculados fueron colocados en estufa a 37 °C, ambientado a 5% de CO2 e incubados durante 5 a 7 días (Pan et al., 2012; Wang et al., 2015), observándose diariamente buscando evidencia de formación de sincitios, fusión celular, formas redondeadas y lisis celular en más del 50% del pozo inoculado (Chen et al., 2014; Wang et al., 2015). Las muestras de cada pocillo se colectaron al observarse dichos cambios, se centrifugaron a 3000 g por 10 min y el sobrenadante fue conservado para la realización del siguiente pasaje. De no existir cambios, se realizó hasta cinco pasajes antes de considerar negativa a una muestra. Los aislados positivos y negativos fueron congelados para su confirmación por RT-PCR en tiempo real con el protocolo descrito anteriormente.
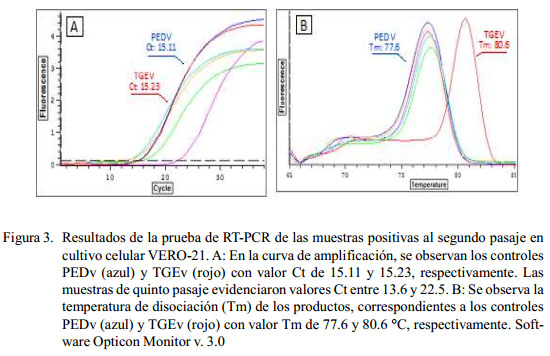
La prueba de IHC permitió detectar al PEDv en el 94.3% (35/37) de las muestras de campo, siendo todas negativas a TGEv (Figura 2). Asimismo, el resultado del RTPCR en tiempo real indicó que el 97.1% (34/ 35) de estas muestras fueron positivas. Los controles positivos de PEDv y TGEv presentaron un valor de ciclo umbral (Ct) de alrededor de 15, con un valor de temperatura de disociación (Tm) de 77.6 °C para PEDv y 80.1 °C para TGEv. No se observó amplificación del virus de PPC utilizado como control negativo. Las muestras de campo positivas a RT-PCR en tiempo real presentaron valores de amplificación Ct de 10 a 21 y Tm de 77.3 a 77.9, evidenciando la alta carga viral y la amplificación de un producto específico (Figura 3).
Los resultados obtenidos en el aislamiento viral evidenciaron la presencia de lesiones en el 23.0% (8/34) de las muestras de PEDv positivas por RT-PCR en tiempo real. No se observaron lesiones en el resto de muestras inoculadas. Dichos cambios se describieron como células redondeadas y lisis celular, principalmente; y fueron observados desde el primer hasta el segundo pasaje (Figura 4). La extensión de los cambios celulares abarcó más del 50% del pozo inoculado.
La confirmación por RT-PCR en tiempo real se obtuvo en el 50% (4/8) de las muestras aisladas con valores en el Ct entre 13.6 y 22.5. De igual manera, todas las muestras que hasta el 5o pasaje no evidenciaron efecto citopático (ECP) resultaron negativas al RTPCR en tiempo real.
Las cuatro muestras aisladas en VERO 21 y confirmadas por RT-PCR en tiempo real, correspondieron a las granjas ubicadas en Chilca (n=1), Pachacamac (n=2) y Ventanilla (n=1).
DISCUSIÓN
La presencia del virus PED se reporta por primera vez en mayo de 2013 en granjas porcinas de Iowa y Minnesota, EEUU, extendiéndose para fines de ese año a 18 estados del país (Kehrli et al., 2014). La enfermedad fue reportada por primera vez en el Perú en setiembre de 2013 por SENASA y la FMV-UNMSM (SENASA, 2013), mediante la prueba de RT-PCR en tiempo real, pero sin el aislamiento viral, prueba reconocida como ‘Gold standard’ para el reporte de una enfermedad cuando es considerada exótica para un país o región.
La primera evidencia clínica de la enfermedad de PED en el país se hizo a través de la prueba cualitativa de IHC (Song y Park, 2012), prueba que permite diferenciar a PEDv de TGEv, ya que ambos virus presentan signos clínicos y causan lesiones macroscópicas y microscópicas similares (Jung et al., 2014; Madson et al., 2014). Esta prueba se hizo ya que se había reportado un brote de TGEv en lechones en 2012 (M. Ramírez, Lima, comunicación personal). El 94.3% (35/37) de las muestras resultaron positivas a PEDv por IHC, siendo probable que las dos muestras negativas a IHC tengan otro origen, ya que los signos clínicos gastroentéricos no son específicos a PEDv (OIE, 2104).
Siendo la PED una enfermedad exótica para el país era necesario la confirmación molecular de las 35 muestras positivas a IHC. La prueba de RT-PCR dúplex en tiempo real permitió diferenciar los virus causantes de TGE y PED en una sola corrida, donde el 97.1% (34/35) de las muestras positivas a IHC resultaron positivas a PED. La alta sensibilidad y especificidad de técnicas moleculares como el PCR, permite detectar hasta el 100% de animales infectados si se trabaja con las muestras adecuadas y con protocolos adecuados (Kim et al., 2001; Kim y Chae, 2002), por lo que constituye la técnica más idónea para la detección viral frente a otras pruebas convencionales (Song et al., 2006; Lin et al., 2015).
De las ocho muestras que presentaron lesión en el cultivo celular, solo cuatro fueron confirmados como PEDv mediante la prueba de RT-PCR en tiempo real (Figura 3). Los cuatro aislados negativos a PEDv serían otros agentes virales que encontraron receptores para su ingreso y replicación en las células VERO ocasionando lesiones similares a PEDv. Existen múltiples factores que afectan los resultados del aislamiento del PEDv en cultivo celular, como son el tipo de espécimen, título viral, sustancias inhibidoras en el contenido intestinal, así como factores específicos de las cepas virales, tipos de líneas celulares y las condiciones de cultivo que tiene que ver con la concentración de tripsina (Wang et al., 2015). Diversos estudios describen la dificultad de la técnica de aislamiento de PEDv en cultivo celular a partir de muestras intestinales o de heces (Chen et al., 2014; Wang et al., 2015; Lv et al., 2016). Oka et al. (2014) reportaron el aislamiento con éxito de 9 de las 88 muestras positivas por pasajes seriados en células VERO suplementadas con tripsina. Estas cepas PEDv mostraron tres perfiles genéticos en la región parcial de la proteína S en base al análisis de la secuencia genómica.
En el presente estudio, la lesión o ECP fue observado en los dos primeros pasajes, similar a lo reportado por Hofmann y Wyler (1988) y a diferencia de lo descrito por Pan et al. (2012), donde el ECP fue observado en el sétimo pasaje a pesar de que el virus era detectado por RT-PCR en los pasajes previos. Estas discrepancias se atribuyen a la relativa susceptibilidad de las células de la línea VERO a las diferentes cepas de PEDv (Pan et al., 2012).
Otra dificultad en el aislamiento del PEDv está en la concentración de tripsina en el medio de cultivo celular. El protocolo utilizado fue una adaptación de los descritos por Pan et al. (2012) y Oka et al. (2014); sin embargo, hasta el momento no se ha podido lograr una tasa de replicación mayor que permita obtener altas concentración de viriones. Cabe mencionar que diversos autores coinciden en la dificultad que representa adaptar esta técnica en el laboratorio (Chen et al., 2014; Oka et al., 2014; Wang et al., 2015); no obstante, se continúa realizando los ensayos correspondientes a fin de obtener un protocolo más eficiente que permita el aislamiento de mayor número de cepas del PEDv en el Perú.
CONCLUSIONES
-
Se aislaron cuatro cepas de campo del virus de la diarrea epidémica porcina (PED) de lechones infectados provenientes de granjas de Lima.
-
El aislamiento de cepas de campo del PEDv permitió obtener replicación en el 12.9% de las muestras inoculadas (4/31) en células VERO-21 evidenciadas hasta el segundo pasaje.
-
Las cepas del PEDv aisladas presentaron un Ct de 15.11 y un Tm de 77.6.
Agradecimientos
Este estudio fue financiado con fondos del Vicerrectorado de Investigación y Postgrado de la Universidad Nacional Mayor de San Marcos (Proyecto N.° 140801011).
LITERATURA CITADA
1. Chen Q, Li G, Stasko J, Thomas JT, Stensland WR, Pillatzki AE, Gauger PC, Schwartz KJ, et al. 2014. Isolation and characterization of porcine epidemic diarrhea viruses associated with the 2013 disease outbreak among swine in the United States. J Clin Microbiol 52: 234-243. doi: 10.1128/JCM.02820-13 [ Links ]
2. Hofmann M, Wyler R. 1988. Propagation of the virus of porcine epidemic diarrhea in cell culture. J Clin Microbiol 26: 2235-2239. [ Links ]
3. HuX, LiN, TianZ, YinX, QuL, Qu J. 2015. Molecular characterization and phylogenetic analysis of transmissible gastroenteritis virus HX strain isolated from China. BMC Vet Res 11:72. doi: 10.1186/s12917-015-0387-8 [ Links ]
4. Jung K, Saif LJ. 2015. Porcine epidemicdiarrhea virus infection: etiology, epidemiology, pathogenesis and immunoprophylaxis. Vet J 204: 134-143. doi: 10.1016/j.tvjl.2015.02.017 [ Links ]
5. Jung K, Wang Q, Scheuer KA, Lu Z, Zhang Y, Saif LJ. 2014. Pathology of US porcine epidemic diarrhea virus strain PC21A in gnotobiotic pigs. Emerg Infect Dis 20: 668-671. doi: 10.3201/ eid2004.131685 [ Links ]
6. Kehrli M, Stasko J, Lager K. 2014. Status report on porcine epidemic diarrhea virus in the United States. Animal Frontiers 4(1): 44-45. doi: 10.2527/ af 2014-0006 [ Links ]
7. Kim O, Chae C. 2000. In situ hybridization for the detection and localization of porcine epidemic diarrhea virus in the intestinal tissues from naturally infected piglets. Vet Pathol 37: 62-67. doi: 10.1354/vp.37-1-62 [ Links ]
8. Kim O, Chae C. 2002. Comparison of reverse transcription polymerase chain reaction, immunohistochemistry, and in situ hybridization for the detection of porcine epidemic diarrhea virus in pigs. Can J Vet Res 66: 112-116. [ Links ]
9. Kim SY, Song DS, Park BK. 2001. Differential detection of transmissible gastroenteritis virus and porcine epidemic diarrhea virus by duplex RT-PCR. J Vet Diagn Invest 13: 516-520. doi: 10.1177/ 104063870101300611 [ Links ]
10. Kwon HJ, Ryu YB, Kim YM, Song N, Kim CY, Rho MC, Jeong JH, et al. 2013. In vitro antiviral activity of phlorotannins isolated from Ecklonia cava against porcine epidemic diarrhea coronavirus infection and hemagglutination. Bioorg Med Chem 21: 4706-4713. doi: 10.1016/j.bmc.2013.04.085 [ Links ]
11. Li W, Li H, Liu Y, Pan Y, Deng F, Song Y, Tang X, He Q. 2012. New variants of porcine epidemic diarrhea virus, China, 2011. Emerg Infect Dis 8: 1350-1353. doi: 10.3201/eid1808.120002 [ Links ]
12. Lin CM, Gao X, Oka T, Vlasova AN, Esseili MA, Wang Q, Saif LJ. 2015b. Antigenic relationships among porcine epidemic diarrhea virus and transmissible gastroenteritis virus strains. J Virol 89: 3332-3342. doi: 10.1128/JVI.03196-14 [ Links ]
13. Lv C, Xiao Y, Li XD, Tian K. 2016. Porcine epidemic diarrhea virus: current insights. Virus Adapt Treat 8: 1-12. doi: 10.2147/VAAT.S107275 [ Links ]
14. Madson DM, Magstadt DR, Arruda PH, Hoang H, Sun D, Bower LP, Bhandari M, et al. 2014. Pathogenesis of porcine epidemic diarrhea virus isolate (US/Iowa/18984/2013) in 3-week-old weaned pigs. Vet Microbiol 174: 60-68. doi: 10.1016/j.vetmic.2014.09.002 [ Links ]
15. [OIE] Organización Mundial de Sanidad Animal. 2014. Infección por el virus de la diarrea epidémica porcina. Ficha técnica de la OIE. [Internet]. Disponible en: http://www.oie.int/fileadmin/ Home/esp/Our_scientific_expertise/ docs/pdf/E_factsheet_PEDV.pdf [ Links ]
16. Oka T, Saif LJ, Marthaler D, Esseili MA, Meulia T, Lin CM, Vlasova AN, et al. 2014. Cell culture isolation and sequence analysis of genetically diverse US porcine epidemic diarrhea virus strains including a novel strain with a large deletion in the spike gene. Vet Microbiol 173: 258-269. doi: 10.1016/ j.vetmic.2014.08.012 [ Links ]
17. Pan Y, Tian X, Li W, Zhou Q, Wang D, Bi Y, Chen F, Song Y. 2012. Isolation and characterization of a variant porcine epidemic diarrhea virus in China. Virology J 9: 195. doi: 10.1186/1743422X-9-195 [ Links ]
18. [SENASA] Servicio Nacional de Sanidad Agraria. 2013. Dirección de SanidadAnimal. Ministerio de Agricultura y Riego. República del Perú. Boletín Epidemiológico. Semana Epidemiológica N.° 40-44. [Internet]. Disponible en: https://www.senasa.gob.pe/senasa/wpcontent/uploads/2014/11/ BOLET%C3%8DN-OCTUBRE 2013.pdf [ Links ]
19. Shirato K, Matsuyama S, Ujike M, Taguchi F. 2011. Role of proteases in the release of porcine epidemic diarrhea virus from infected cells. J Virol 85: 78727880. doi: 10.1128/JVI.00464-11 [ Links ]
20. Song DS, Kang BK, Oh JS, Ha GW, Yang JS, Moon HJ, Jang YS, Park BK. 2006. Multiplex reverse transcription-PCR for rapid differential detection of porcine epidemic diarrhea virus, transmissible gastroenteritis virus, and porcine group A rotavirus. J Vet Diagn Invest 18: 278-281. doi: 10.1177/ 104063870601800309 [ Links ]
21. Song D, Park B. 2012. Porcine epidemic diarrhea virus: a comprehensive review of molecular epidemiology, diagnosis, and vaccines.Virus Genes 44: 167-175. doi: 10.1007/s11262-012-0713-1 [ Links ]
22. Sozzi E, Luppi A, Lelli D, Moreno A, Canelli E, Brocchi E, Lavazza A, Cordioli P. 2010. Comparison of enzyme-linked immunosorbent assayand RT-PCR for the detection of porcine epidemic diarrhoea virus. Res Vet Sci 88:166:168. doi: 10.1016/j.rvsc.2009.05.009 [ Links ]
23. Suzuki T, Murukami S, Takahashi O, Kodera A, Masuda T, Itoh S, Miyazaki A, et al. 2015. Molecular characterization of pig epidemic diarrhea viruses isolated in Japan from 2013 to 2014. Infect Genet Evol 36: 363-368. doi: 10.1016/j.meegid.2015.10.017 [ Links ]
24. Wang L, Byrum B, Zhang Y. 2014. New variant of porcine epidemic diarrhea virus, United States, 2014. Emerg Infect Dis 20: 917-919. doi: 10.3201/eid2005.140195 [ Links ]
25. WangY,GaoX,YaoY,ZhangY,Lv C , Sun Z ,WangY, et al. 2015. The dynamics of Chinese variant porcine epidemic diarrhea virus production in Vero cells and intestines of 2-day old piglets. Virus Res 208: 82-88. doi: 10.1016/j.virusres.2015.06.009 [ Links ]
26. Zhao S, Gao J, Zhu L, Yang Q. 2014. Transmissible gastroenteritis virus and porcine epidemic diarrhoea virus infection induces dramatic changes in the tight junctions and microfilaments of polarized IPEC-J2 cells. Virus Res 192: 34-45. doi: 10.1016/j.viruses.2014.08.014 [ Links ]
Recibido: 1 de abril de 2017
Aceptado para publicación: 27 de julio de 2017