Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.29 no.1 Lima ene./mar. 2018
http://dx.doi.org/10.15381/rivep.v29i1.14198
ARTÍCULOS PRIMARIOS
Variabilidad genética de las poblaciones de palomas domésticas (Columba livia) en Sincelejo, Sucre
Genetic variability of domestic pigeon (Columba livia) populations in Sincelejo, Sucre
Enrique Pardo1, Jorge Bracamontes1, Mauricio Begambre1
1 Departamento de Biología, Facultad de Ciencias Básicas, Universidad de Córdoba, Colombia
2 E-mail: epardop@correo.unicordoba.edu.co
RESUMEN
El objetivo de la presente investigación fue determinar la variabilidad genética de las poblaciones de palomas domésticas (Columba livia), utilizando genes que codifican la coloración y diseño del plumaje en Sincelejo, Sucre (Colombia). Las poblaciones, distribuidas en veinticuatro colonias, se estudiaron entre julio de 2016 y enero de 2017 e incluyeron 1402 individuos. Se evaluaron los marcadores: Patrón del plumaje, Coloración primaria, Spread y Grizzle. Los parámetros genéticos frecuencia alélica, diversidad genética y estructura poblacional fueron calculados a través del programa PopGene 1.31, la distancia genética se determinó mediante el programa FSTAT v. 2.9.3.2 y el dendrograma se realizó utilizando el programa MEGA 7. Las frecuencias alélicas obtenidas oscilaron entre 0.2290 para el alelo Checker (C), 0.1458 para el marcador T-Pattern (CT), 0.1317 para el gen Grizzle (G), 0.0882 para el marcador Ash-Red (BA) y 0.0282 para el alelo Spread (S). El coeficiente de diferenciación genética fue de 0.0148 y el número de migrantes detectados por generación fue de 33 individuos. El dendograma mostró tres grupos: uno ubicado en la zona céntrica de Sincelejo y dos grupos en la periferia. Los resultados muestran poca diferenciación genética entre las poblaciones de palomas estudiadas.
Palabras clave: Columba livia; genes del plumaje; frecuencias alélicas; flujo génico; variabilidad genética; Sincelejo
ABSTRACT
The aim of this research was to determine the genetic variability of domestic pigeon populations (Columba livia), using genes that encode plumage color in Sincelejo, Sucre (Colombia). The populations, distributed in twenty-four colonies, were studied between July 2016 and January 2017, and included 1402 individuals. The markers were: Plumage pattern, Primary coloration, Spread and Grizzle. The genetic parameters allele frequencies, genetic diversity and population structure were calculated through the program PopGene 1.31, genetic distance was determined by the program FSTAT v. 2.9.3.2, and the dendrogram was carried out using the program Mega 7. The allelic frequencies obtained varied from 0.2290 for the allele Checker (C), 0.1458 for the marker T-Pattern (CT), 0.1317 for the gene Grizzle (G), 0.0882 for the marker Ash-Red (BA) and 0.0282 for the allele Spread (S). The coefficient of genetic differentiation was 0.0148 and the number of migrants detected per generation was 33 individuals. The dendrogram showed three groups: one located in the central zone of Sincelejo and two groups in the periphery. The results show little genetic differentiation among the studied pigeon populations.
Key words: Columba livia; plumage genes; allelic frequencies; gene flow; genetic variability; Sincelejo
INTRODUCCIÓN
Columba livia (Gmelin, 1789) tiene su origen en la paloma endémica de Eurasia y África, y actualmente se encuentra distribuida a nivel mundial (Ferman et al., 2010). La paloma doméstica es de tamaño mediano (3035 cm) con cola mediana, pico negruzco, iridiscencias moradas y verdes en el cuello, patas rosadas y ojos azabache (Gómez de Silva et al., 2005). No presenta dimorfismo sexual, pero muestra plumaje variable entre individuos. El patrón silvestre es azul claro con dos franjas de color negro en las alas; sin embargo, los colores en la mayor parte de los individuos varían desde blanco y blanquecino con manchas irregulares rojizas hasta negro con plumas primarias y cola blanca. Su peso oscila entre 180 y 355 g (Del Hoyo et al., 1997).
Según Olalla et al. (2009), la paloma doméstica en su medio natural anida en acantilados costeros o en tierras altas. En ambientes urbanos se agrupa en parvadas de hasta cientos de individuos. Normalmente se alimentan, mueven y vuelan juntas. Se les encuentra en techos, azoteas, tuberías de desagüe, buhardillas y domos, donde construyen sus nidos a base de ramas secas y hierbas que colocan sobre una base simple.
Las palomas domésticas (Columba livia) se caracterizan por una amplia variación en su plumaje, lográndose diferenciar hasta 60 factores hereditarios que codifican el patrón y la coloración (Haag-Wackernagel et al., 2006; Hetmañski y Jarosiewicz, 2008; Èanády y Mošanský, 2013, 2016), donde estos marcadores fenotípicos constituyen una buena herramienta en estudios de genética poblacional debido al bajo costo, fácil manipulación e identificación, así como rápida obtención de resultados.
En Colombia se han desarrollado estudios de la diversidad genética de palomas en Bogotá (Pardo et al., 2014), Montería (Begambre, 2014) y Lorica (Causil, et al., 2016). En Sincelejo solo existe reportes de la población estimada en el Mercado Público (Villalba et al., 2015). Es así que debido al desconocimiento de la variabilidad genética de las palomas de Sincelejo, el presente estudio tuvo como objetivo determinar la variabilidad genética de la paloma doméstica (Columba livia) en Sincelejo, Sucre, Colombia, mediante genes que codifican la coloración y diseño del plumaje.
MATERIALES Y MÉTODOS
El estudio se realizó en el área urbana de Sincelejo, Sucre (9°172 583 N; 75°232 453 O), con temperatura promedio de 27 °C y altura promedio de 213 msnm. Se realizaron muestreos aleatorios en 24 barrios entre julio de 2016 y enero de 2017 mediante observación directa y registros fotográficos en el horario de 06:00 a 08:00 horas, donde las palomas forman las bandadas en los sitios de forrajeo (Hetmañski y Jarosiewicz, 2008). Cada ruta se utilizó una sola vez a fin de evitar el re-muestreo. Luego se determinó para cada individuo los marcadores morfológicos conforme a Haag-Wackernagel et al. (2006).
Los cuatro marcadores morfológicos evaluados en el estudio se caracterizan por ser heredables y por presentar una distribución mendeliana (Haag-Wackernagel et al., 2006). Estos son:
-
Patrones primarios: Los patrones primarios presentan herencia autosómica. Exhiben una línea de dominancia CT>C>C+, donde C+: barradas, C: Checker (plumas negras en las alas cubriendo menos del 50% de las mismas) y CT: T-pattern (plumas negras del ala cubriendo más del 50% de las mismas).
-
Coloración primaria: Los genes que expresan la coloración básica están situados en el cromosoma sexual Z, pero la herencia no está ligada al sexo debido a la dominancia graduada. Por tanto, se analizó como un carácter autosómico dominante en la cual BA>B+, donde B+: corresponde a palomas azules y oscuras y BA: palomas rojas (Ash-Red).
-
Grizzle: El patrón Grizzle (G) es un sistema autosómico que expresa el alelo G. Implica una mezcla de blanco y negro en la misma pluma. Observable principalmente en la cabeza.
-
Spread: El patrón Spread (S), sistema autosómico que suprime los patrones primarios. Las palomas presentan una coloración negra o gris oscuro. En la cola no presenta la banda terminal y la coloración es homogénea.
La estimación de las frecuencias alélicas de cada marcador, así como las medidas de diversidad genética establecidas por Nei correspondientes a la heterocigosidad esperada (He), heterocigosidad observada (Ho), heterocigosidad media esperada de las subpoblaciones (H ), heterocigosidad esperada de la población total (H ), heterocigosidad total distribuida entre las poblaciones (DST), la diversidad génica relativa (GST) y el flujo génico (Nm) se estimaron a través del programa PopGene 1.31 (Yeh et al., 1999). La distancia genética entre las poblaciones se evaluó mediante el programa FSTAT v. 2.9.3.2 (Goudet, 1995).
La elaboración del dendrograma se realizó a través del método Neighbor-Joining mediante el programa MEGA 7 (Kumar et al., 2016).
RESULTADOS
Se muestrearon 24 poblaciones con un total de 1402 palomas, donde 1055 individuos fueron de las colonias del centro (Parroquia, Mercado Nuevo, Mercado Viejo, Comando de la Policía, España, Plaza Majagual, La Narcisa, Las Mercedes, Mochila, San Vicente, Berbel, La Manga) y 347 individuos en las poblaciones de la periferia (El Progreso, Uribe Uribe, Las Américas, Nuevo Majagual, Villa Mady, El Cortijo, El Bosque, Botero, El Zumbado, La Campina, Las Colinas y 7 de Agosto).
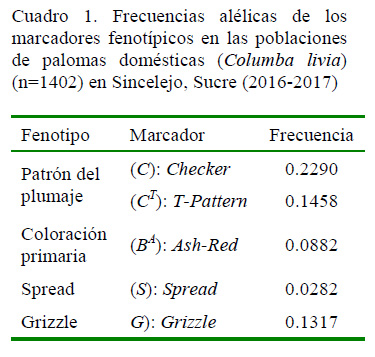
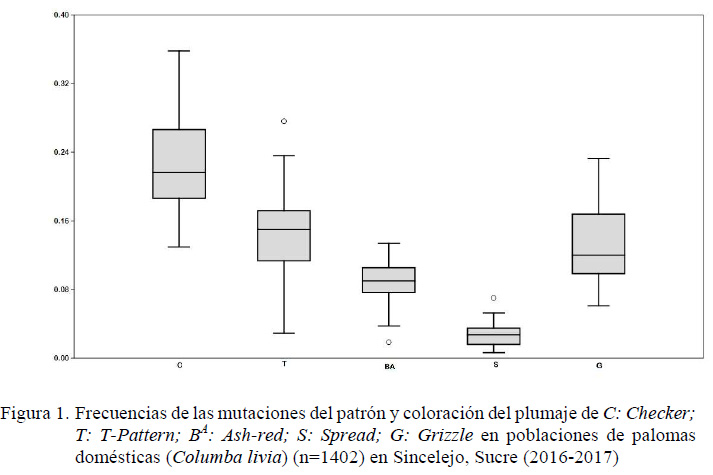
El alelo Checker (C) presentó las mayores frecuencias (0.229), seguida por el alelo T-Pattern (CT). Las frecuencias más bajas corresponden al alelo Spread (S) con un promedio de 0.028 (Cuadro 1). Además, se observó que el alelo Spread (S) presentaba una baja diferenciación de las frecuencias alélicas entre las colonias, mientras que el alelo Checker (C) tenía la mayor variación de las frecuencias (Figura 1).
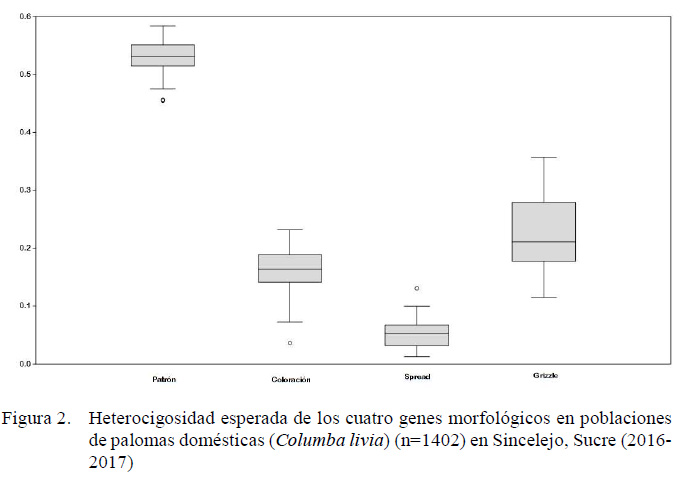
El marcador que determina Patrón del plumaje obtuvo la heterocigosidad esperada más alta con un promedio de He= 0.535, mientras que el marcador Spread presentó los valores más bajos (0.054), con un promedio general de He= 0.241. Asimismo, los valores de heterocigosidad esperada del marcador Grizzle fueron los más variables entre las colonias (Figura 2).
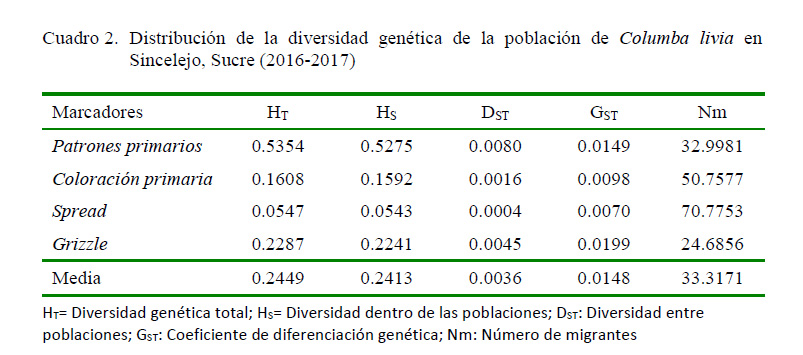
El coeficiente de diferenciación genética en la población total (Gst = 0.0148) muestra que el 1.48% de la diversidad genética se encuentra entre las poblaciones y un 98.52% dentro de las poblaciones, señalando que las 24 poblaciones comparten una gran proporción de la diversidad total (Hartl y Clark, 1997), generando un elevado flujo genético entre las subpoblaciones (Nm = 33.317; Cuadro 3).
En el presente estudio el numero promedio de migrantes por generación entre poblaciones fue de 33 individuos, valor superior al estimado por Piñero et al. (2008) de cuatro, donde a dichas poblaciones las describe como una gran población panmíctica. Este valor refleja las reducidas distancias genéticas entre las colonias, con valores de distancias genéticas que oscilan entre DNEI = 0.0001 (El Zumbado y Botero) y DNEI = 0.0206 (Centro Parroquia y El Progreso) con un promedio de DNEI = 0.0070 (Cuadro 3).
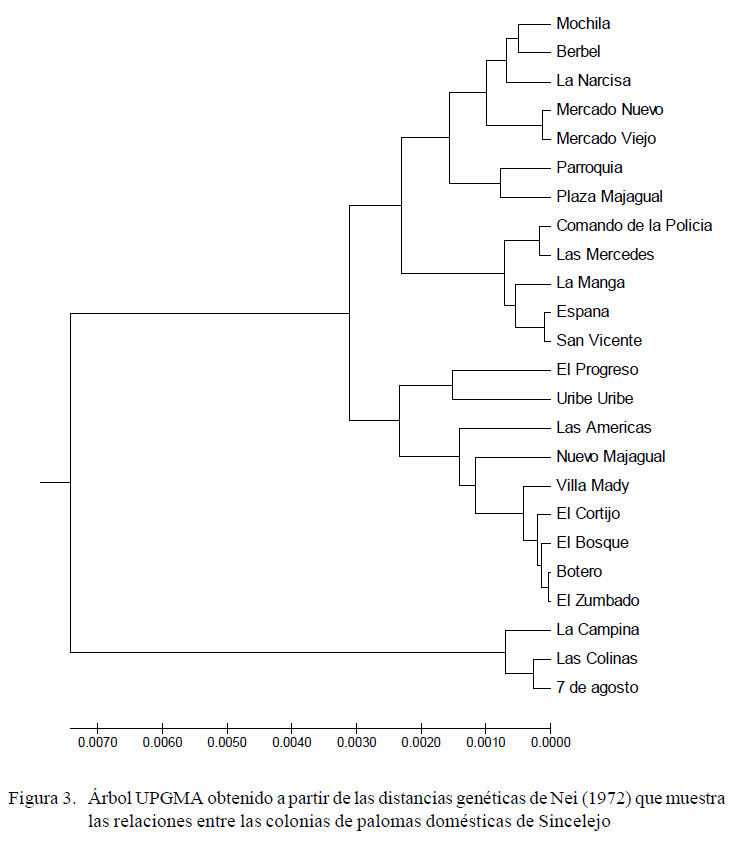
El dendrograma (Figura 3) mostró la conformación de tres grupos: uno ubicado en la zona céntrica de Sincelejo y dos grupos en la periferia de la ciudad.
DISCUSIÓN
En Sincelejo se mantuvo la preferencia de las palomas por la zona céntrica, evento similar a lo reportado por otros autores (Sacchi et al., 2002; Giunchi et al., 2007; Hetmañski y Jarosiewicz, 2008; Senar et al., 2009), específicamente por la zona comercial e histórica, donde se presentó el mayor número de individuos. La predilección hacia estos lugares se debe a la abundancia de negocios informales de venta de comidas y la comercialización de diferentes tipos de granos que generan desechos orgánicos que sirven de alimento a las palomas. Además, estas zonas presentan buenos sitios de anidamiento y lugares de refugio que favorecen la conglomeración de palomas (Ferman et al., 2010).
El alelo de mayor frecuencia en la población de Sincelejo fue Checker (C = 0.229), similar a lo reportado por Pardo et al. (2014) para la ciudad de Bogotá (C = 0.382). Asimismo, se destaca una alta frecuencia para el alelo Grizzle (G = 0.1317) (Cuadro 1) en comparación con Bogotá (Pardo et al., 2014) y Montería (Begambre, 2014), las cuales presentan frecuencias de G = 0.021 y G = 0.04, consecutivamente. También se encontraron frecuencias alélicas altas del marcador Ash–red (BA = 0.088) respecto a Bogotá (0.045) y Montería (0.03).
Obukhova (2007) postula la correlación entre la temperatura media ambiental y la coloración de las palomas domésticas, encontrándose un mayor número de aves melánicas en zonas frías con respecto a las zonas cálidas; evento que puede estar favoreciendo las bajas frecuencias de alelos que expresan colores melánicos en Sincelejo (C = 0.2290, T = 0.1458 y S = 0.0282) y favoreciendo dichos alelos en Bogotá (C = 0.382 y S = 0.250; Pardo et al., 2014).
La distribución de los polimorfismos en Sincelejo no está asociada a posibles casos de contaminación, debido a la concentración de palomas de coloraciones claras en sitios con elevado tráfico, escenarios con potenciales efectos genotóxicos (Carranza, 2011); resultados no acorde con la selección natural bajo «melanismo industrial», en la cual, la melanina captura iones de metales pesados y los almacenan en plumas y partes inertes del cuerpo, ayudando a la desintoxicación en palomas de coloración oscura (Chatelain et al., 2014); ni a efectos de endoparásitos, teniendo en cuenta que las palomas melánicas presentan una mayor habilidad en el control de infecciones (Jacquin et al., 2013).
Sincelejo mostró una heterocigosidad intermedia (He= 0.241) respecto a lo reportado para Bogotá con He= 0.273 (Pardo et al., 2014), Montería con H (Begambre, 2014) y Lorica con He= 0.16 (Begambre, 2014) y Lorica con He = 0.365 (Causil et al., 2016), debido posiblemente a que su pool genético inicial, sin efectos ambientales ni intervención humana, que hace que no cambien las frecuencias de los marcadores con el tiempo (Caujapé, 2006; Hetmañski y Jarosiewicz, 2008).
Sincelejo presentó la menor diferenciación genética entre las colonias frente a lo reportado en Bogotá (Pardo et al., 2014) y Montería (Begambre, 2014) debido al efecto del reciente incremento demográfico en Sincelejo (Chávez, 2016), lo que ha permitido la expansión de las palomas a nuevos territorios sin que haya transcurrido el tiempo suficiente para que ocurra una diferenciación genética de las poblaciones nuevas; además, es importante mencionar que el tamañomuestral fue menor en las ciudades reportadas, lo que teóricamente incrementaría la diferenciación interpoblacional generado por la deriva genética (Demarchi, 2009).
El elevado flujo genético encontrado en Sincelejo (Nm=33.31; Cuadro 2), permite deducir que las poblaciones se encuentran muy relacionadas genéticamente, exhibiendo una escasa diferenciación interpoblacional.
CONCLUSIONES
-
La población de palomas de Sincelejo mostró la mayor frecuencia alélica para C = 0.2290 y la menor para BA = 0.0882, predominando los marcadores que expresan coloraciones claras, asociadas a condiciones ambientales específicas, como las temperaturas elevadas.
-
La diversidad genética encontrada fue moderada, mostrando a la población de palomas no estar bajo influencia de selección o deriva genética que modifique el pool genético originario.
-
Existe una baja diferenciación interpoblacional en las colonias de palomas debido al elevado flujo genético y expansión demográfica reciente; fenómenos que evitan la deriva genética, principal fuente de diferenciación poblacional de esta especie sinantrópica.
LITERATURA CITADA
1. Begambre M. 2014. Abundancia, distribución, polimorfismos y estructura genética de la población de palomas Columba livia de Montería-Córdoba. Tesis de Biólogo. Montería: Univ. de Córdoba. 60 p. [ Links ]
2. Èanády A, Mošanský L. 2013. Population size and plumage polymorphism of feral pigeon (Columba livia forma urbana) from urban environment of Košice city (Slovakia). Zool Ecol 23: 104110. doi:10.1080/21658005.2013.-797149
3. Èanády A, Mošanský L. 2016. Plumage colour patterns and density of feral pigeon from urban areas of the Stropkov and Svidník cities (Ondavská Vrchovina Mts, Slovakia). Fol Oecol 8(2): 5-13
4. Carranza L. 2011. Cuantificación de micronúcleos en células de sangre periférica de mototaxistas que trabajan en la ciudad de Cartagena de Indias. Tesis de Magíster. Bogotá: Univ. Nacional de Colombia. 93 p. [ Links ]
5. Caujapé CJ. 2006. Brújula para botánicos desorientados en la genética de poblaciones Las Palmas de Gran Canaria, España: Exegen Ed. 133 p. [ Links ]
6. Causil A, Rodríguez D, Causil. 2016. Diversidad genética de palomas domésticas (Columba livia) en Lorica Colombia utilizando genes que codifican la coloración del plumaje. Rev Inv Vet Perú 27: 448-457. doi: 10.15381/rivep.v27i3.11476 [ Links ]
7. Chatelain M, Gasparini J, Jacquin L, Frantz A. 2014. The adaptive function of melanin-based plumage coloration to trace metals. Biol Lett 10: 20140164. doi: 10.1098/rsbl.2014.0164 [ Links ]
8. Chávez I. 2016. Sincelejo ciudad emergente o en emergencia. Las2Orillas. [Internet]. Disponible en: https://www las2orillas co/sincelejo-ciudad-emergente-o-en-emergencia/ [ Links ]
9. Del Hoyo J, Elliot A, Sargatal J. 1997. Handbook of the birds of the world. Vol 4. Sandgrouse to Cuckoos. Barcelona, España: Lynx ed. 679 p. [ Links ]
10. Demarchi A. 2009. Microsatélites distancias genéticas y estructuras de poblaciones nativas sudamericanas. Rev Argent Antropol Biol 11(1): 73-88. [ Links ]
11. Ferman L, Peter H, Montalti D. 2010. A study of feral pigeon Columba livia var in urban and suburban areas in the city of Jena Germany. Arx Misc Zool 8: 1-8. [ Links ]
12. Giunchi D, Gaggini V, Baldaccini N. 2007. Distance sampling as an effective method for monitoring feral pigeon (Columba livia f domestica) urban populations. Urban Ecosyst 10: 397-412. [ Links ]
13. Gómez De Silva H, Oliveras De Ita A, Medellín RA. 2005. Vertebrados superiores exóticos en México: diversidad, distribución y efectos potenciales. Instituto de Ecología, Universidad Nacional Autónoma de México. Bases de datos SNIB-CONABIO. Proyecto U020. México, DF, México. [ Links ]
14. Goudet J. 1995. FSTAT (version 1.2): a computer program to calculate F statistics. J Heredity 86: 485-486. doi: 10.1093/oxfordjournals.jhered.a111627 [ Links ]
15. Haag-Wackernagel D, Heeb P, Leiss 2006. Phenotype-dependent selection of juvenile urban feral pigeons Columba livia. Bird Stud 53: 163-170. doi: 10.1080/00063650609461429 [ Links ]
16. Hartl D, Clark A. 1997. Principles of population genetics. Sunderland, USA: Sinauer Associates. 683 p. [ Links ]
17. Hetmañski T, Jarosiewicz A. 2008. Plumage polymorphism and breeding parameters of various feral pigeon (Columba livia Gm) morphs in urban area (Gdañsk North Poland). Pol J Ecol 56: 683-691. [ Links ]
18. Jacquin L, Haussy C, Bertin C, Laroucau K, Gasparini J. 2013. Darker female pigeons transmit more specific antibodies to their eggs than do paler ones. Biol J Linn Soc 108: 647-657. doi: 10.1111/bij.12001 [ Links ]
19. Kumar S, Stecher G, Tamura K. 2016. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Mol Biol Evol 33: 1870-1874. doi: 10.1093/molbev/msw054 [ Links ]
20. Obukhova N. 2007. Polymorphism and phene geography of the blue rock pigeon in Europe. Russ J Gen 43: 609-619. doi: 10.1134/S1022795407050031 [ Links ]
21. Olalla A, Ruiz V, Ruvalcaba I, Mendoza R. 2009. Palomas, especies invasoras. CONABIO. Biodiversitas 82: 7-10. [ Links ]
22. Pardo E, Cavadía T, Alvarino G. 2014. Análisis de la diversidad genética de la paloma domestica (Columba livia) en Bogotá, Colombia, utilizando genes que codifican la coloración y diseño del plumaje. Rev Fac Cien Bas 13(1): 35-45 [ Links ]
23. Piñero D, Caballero M, Cabrera T, Cantero C H, Casas A, et al. 2008. La diversidad genética como instrumento para la conservación y el aprovechamiento de la biodiversidad: estudios en especies mexicanas. En: Soberón J, Halffter G, Llorente J (eds). Capital natural de México. Vol I. Conocimiento actual de la biodiversidad. México: Conabio. p 437-494. [ Links ]
24. Sacchi R, Gentilli A, Razzetti E, Barbieri F. 2002. Effects of building features on density and flock distribution of feral pigeons Columba livia var. domestica in an urban environment. Can J Zool 80: 48-54. doi: 10.1139/z01-202 [ Links ]
25. Senar J, Carrillo J, Arroyo L, Montalvo T, Peracho V. 2009. Estima de la abundancia de palomas (Columba livia var.) de la ciudad de Barcelona y valoración de la efectividad del control por eliminación de individuos. Arx Misc Zool 7: 62-71. [ Links ]
26. Villalba C, De la Ossa A, De la Ossa J. 2015. Densidad de paloma doméstica (Columbia livia domestica Gmelin, 1789) en el antiguo Mercado Público de Sincelejo, Sucre, Colombia. Rev Asoc Col Cienc 27: 72-79. [ Links ]
27. Yeh F, Yang R, Mao J, Boyle T. 1999. POPGENE version 3.1 Microsoft Windows-based freeware for population genetic analysis. University of Alberta. [Internet]. Available in: https://sites.ualberta.ca/~fyeh/popgene.pdf [ Links ]
Recibido: 25 de junio de 2017
Aceptado para publicación: 2 de noviembre de 2017