Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista de Investigaciones Veterinarias del Perú
versão impressa ISSN 1609-9117
Rev. investig. vet. Perú vol.29 no.2 Lima abr./jun. 2018
http://dx.doi.org/10.15381/rivep.v29i2.14482
ARTÍCULOS PRIMARIOS
Caracterización de microorganismos con potencial probiótico aislados de estiércol de terneros Brahman en Sucre, Colombia
Characterization of microorganisms with probiotic potential isolated from brahman calf manure in sucre, Colombia
Pedro Luis Castillo Arroyo1, César Augusto Betancur Hurtado1, Enrique Pardo Pérez2,3
1 Departamento de Ciencias Pecuarias, Facultad de Medicina Veterinaria y Zootecnia, Universidad de Córdoba, Colombia
2 Departamento de Biología, Facultad de Ciencias Básicas, Universidad de Córdoba, Colombia
3 Email: epardop@correo.unicordoba.edu.co
RESUMEN
El objetivo del estudio fue caracterizar microorganismos con potencial probiótico de estiércol de terneros lactantes Brahman en Sucre (Colombia). Se aislaron bacterias y levaduras de las muestras de estiércol y se determinó la capacidad probiótica de estas cepas mediante pruebas de resistencia a sales biliares (0.05, 0.1, 0.15 y 0.3%), resistencia a pH ácido (pH 3, 4, 5.6, 7), tolerancia a NaCl (2, 4, 6, 8, 10%) y actividad antagónica (Salmonella sp y Escherichia coli). Nueve microorganismos fueron identificados y tres pasaron las pruebas de tolerancia. Se determinó la capacidad antagónica frente a bacterias patógenas (Salmonella sp y E. coli), evidenciada por halos (mm) de las tres cepas seleccionadas. La identificación de las cepas se realizó por los métodos bioquímicos API 50 CHL V5.1 para Lactobacillus y API 20 AUX para levaduras. El análisis molecular de dos cepas (M13a y 103M2) identificó la cepa M13a como Enterococcus faecium y la cepa 103M2 como Candida krusei. En conclusión, Enterococcus faecium se convierte en una alternativa viable para la formulación de un biopreparado para mejorar los parámetros productivos y disminuir los trastornos gastrointestinales en los terneros lactantes.
Palabras clave: probióticos; ganado; cebú; Enterococcus; capacidad antagónica; cepa
ABSTRACT
The objective of the study was to characterize microorganisms with probiotic potential of manure of lactating Brahman calves in Sucre (Colombia). Bacteria and yeasts were isolated from manure samples and the probiotic capacity of these strains was determined by bile salt resistance test (0.05, 0.1, 0.15, 0.3%), resistance to acidic pH (pH 3, 4, 5.6, 7 ), tolerance to NaCl (2, 4, 6, 8, 10%) and antagonistic activity (Salmonella sp and Escherichia coli). Nine microorganisms were identified and three passed the tolerance tests. The antagonistic capacity was determined against pathogenic bacteria (Salmonella sp and E. coli), evidenced by halos (mm) of the three selected strains. The identification of the strains was carried out by the biochemical methods API 50 CHL V5.1 for Lactobacillus and API 20 AUX for yeasts. Molecular analysis of two strains (M13a and 103M2) identified strain M13a as Enterococcus faecium and strain 103M2 as Candida krusei. In conclusion, Enterococcus faecium becomes a viable alternative for the formulation of a biopreparation to improve the productive parameters and decrease gastrointestinal disorders in lactating calves.
Key words: probiotic; cattle; zebu; Enterococcus; antagonistic capacity; stains
INTRODUCCIÓN
La crianza de terneros es una de las etapas en la ganadería que más retos presenta a nivel sanitario, y donde se presentan altos índices de mortalidad debido, entre otros factores, a que los mecanismos de defensa del recién nacido no están desarrollados. Así mismo, el desarrollo de las porciones anteriores del aparato digestivo aún debe lograr el tamaño y proporciones que tendrán en su vida adulta (Mee, 2008).
La baja acidez del abomaso en el ternero lactante incrementa el potencial riesgo de bacterias y virus que ingresarán por vía oral, pasando directamente hacia el intestino, donde pueden causar patologías complejas como la diarrea (Bazeley, 2003). La diarrea es característica de un complejo de agentes etiológicos y que para su manifestación deben concurrir diversos factores epidemiológicos, incluyendo el agente etiológico (virus, bacterias y protozoos), el huésped, la transferencia de inmunidad pasiva y las condiciones ecológicas (Camargo et al., 2017).
Estas patologías infecciosas tienen una repercusión económica en la actividad ganadera, pues implican tratamientos veterinarios y mano de obra adicional, además del retraso en el desarrollo corporal de los animales afectados y la posibilidad de contagio a otros terneros que conviven con el afectado (Tepan, 2011). Estas pérdidas han incrementado el uso de antibióticos para proteger los animales y tratar las diarreas; sin embargo, su uso extensivo y prolongado ha favorecido la susceptibilidad de los terneros a microorganismos patógenos, generando resistencia a diversos antibióticos (Fuller, 1989).
Actualmente existe gran interés en reemplazar los antibióticos por otros compuestos más naturales como los probióticos (Rondón et al., 2013); los cuales son considerados como aditivos alimentarios de microorganismos vivos que afectan beneficiosamente al hospedero, influyendo su rendimiento productivo (García et al., 2011).
La microflora normal de becerras lactantes está constituida por microorganismos como Enterococcus faecium, Enterococcus faecalis, Lactobacillus salivarius y Lactobacillus casei (Pascual et al., 1996). Las especies bacterianas comúnmente utilizadas como probióticos en terneros incluyen a los Bacillus sp, Lactobacillus sp, Streptococcus sp, Bifidobacterium sp y levaduras tipo Saccharomyces sp (Figueroa et al., 2006; Mejía et al., 2007; Ávila et al., 2010; García et al., 2011). Con base a esto, el presente estudio tuvo como objetivo caracterizar cepas con potencial probiótico autóctonas y con P. Castillo et al. características específicas que contribuyan a disminuir los problemas gastrointestinales de los terneros lactantes Brahman en el departamento de Sucre, Colombia.
MATERIALES Y MÉTODOS
Área del Estudio
Esta investigación se llevó a cabo en los Municipios de Toluviejo, Coloso y Ovejas, pertenecientes a la subregión Montes de María, en el Departamento de Sucre, Colombia. El área bajo estudio está clasificada como bosque seco tropical (Holdridge, 2000), se encuentra entre 58 y 250 msnm, y registra una temperatura ambiental promedio de 30 °C y precipitaciones entre 1000 y 1200 mm/año.
Recolección de Muestras
Se recolectaron las heces de cuatro terneros por finca, para un total de 12 terneros lactantes Brahman. Los terneros no recibían alimento concentrado y no fueron tratados con antibióticos por lo menos tres meses antes del estudio. Se hizo un pool de heces con las muestras de cada finca, se pusieron en matraces de 250 ml con 100 ml de aceite mineral estéril en anaerobiosis y se colocaron en un agitador a 150 rpm durante 10 min a temperatura ambiente. Luego, se transportaron al laboratorio de Biotecnología de la Universidad de Córdoba (GRUBIODEQ) para el aislamiento.
Aislamiento de Microorganismos
Se suspendió un 1 g de heces en 9 ml de solución salina durante 5 minutos. Luego se realizaron diluciones seriadas hasta 106 en agua de peptona. Muestras de las diluciones 105 y 106 fueron sembradas en tres placas utilizando los medios selectivos Agar de Man, Rugosa, Sharpe (MRS) (Lactobacillus sp y Agar Sabouraud Dextrosa (SDA) para levaduras, a razón de 0.1 ml por placa. Estas fueron incubadas durante 48 horas a 37 °C en atmósfera de dióxido de carbono (CO ). Posteriormente fueron seleccionadas las colonias típicas, que fueron sembradas en 10 ml de caldo MRS, Nutritivo modificado o SDA para su crecimiento y conservación.
Se seleccionaron las colonias con características morfológicas diferentes y se purificaron con pases sucesivos en cultivo MRS. La pureza se verificó con la reacción de Gram, y luego se hizo la prueba de catalasa y oxidasa. Las cepas identificadas como catalasa y oxidasa negativas fueron empleadas para la evaluación de su potencial probiótico (Oyetayo, 2004).
Evaluación de las Propiedades Probióticas
De la siembra por agotamiento en el medio MRS se tomó una colonia y se repicó en 10 ml de caldo MRS. Se incubó a 37 ºC por 24 horas, para luego tomar 1 ml de cada cultivo para realizar diluciones seriadas. De la dilución 105 y 106 se tomó 0.1 ml y se sembró en los medios MRS para conteo de células viables. Conocida la concentración de los inóculos de cada microorganismo, se hizo las pruebas probióticas a cada cepa.
- Tolerancia a cambio de pH. Las cepas fueron sometidas a valores de pH de 3, 4, 5.6 y 7 (incubación a 37 °C durante 24 h). La sobrevivencia de determinó mediante la diferencia del número de microorganismos viables del inóculo con las presentes después del periodo de incubación (Zavaglia et al., 1998) y la resistencia (%) se calculó mediante la siguiente ecuación %R pH = [(UFC/ml) MRS pH x 100] / (UFC/ml) MRS (inóculo) (Kociubinski et al., 1999).
- Se estableció como criterio de selección aquellas cepas que resistieron el pH por encima del 50%. (Rondón et al., 2008; Ávila et al., 2010).
- Tolerancia a sales biliares. Se utilizaron cuatro concentraciones de sales (0.05, 0.1, 0.15, 0.3% p/v) ajustado a pH 7 con HCI 5% (incubación a 37 °C durante 24 h). La sobrevivencia y la resistencia fue comprobada mediante la determinación del número de células viables (UFC) (Rubio et al., 2008; Rondón et al., 2008; Ávila et al., 2010; Monteagudo, 2012).
- Tolerancia a altas concentraciones de NaCl. Se utilizaron caldos MRS, Nutritivo y SDA en cinco concentraciones de NaCl (2, 4, 6, 8, 10% p/v) (incubación a 37 °C durante 24 h). El crecimiento se determinó mediante la medición de la densidad óptica (DO) a 600 nm a 0 y 24 h (Rondón et al., 2008).
- Determinación de la capacidad antagónica. Los microorganismos que fueron seleccionadas por su potencial probiótico fueron enfrentados contra bacterias patógenas (Salmonella sp y E. coli), del banco de cepas del laboratorio GRUBIODEQ. Las cepas bacterianas se sembraron en forma masiva en agar Mueller Hinton. En la superficie de las placas se colocaron tres discos impregnados con los microorganismos seleccionados, se mantuvieron a 15 °C por 30 min y luego fueron incubados a 37 °C durante 48 h. La capacidad antagónica se evidenció por la presencia de halos (mm) y crecimiento alrededor de los discos. Este procedimiento se realizó por triplicado (Leiva et al., 2004; Mejía et al., 2007).
Identificación Bioquímica
Los microorganismos que presentaron las mejores propiedades probióticas se identificaron mediante el sistemaAPI 20 AUX Levaduras o API Lactobacilos. Se incubaron a 37 °C durante 48 horas.
Identificación Molecular
Bacterias
Para la extracción del DNA genómico de los microorganismos con características probióticas se utilizó el kit comercial QIAamp® DNA Stool Mini Kit (QIAGEN Corporation), siguiendo las instrucciones del fabricante. Luego se realizó la amplificación por PCR de la región de 1465 pb del gen ribosomal 16S del RNA. Se utilizaron los iniciadores (primers) 27F, 518F, 800R y 1492R para la identificación molecular de estas bacterias. Para la purificación se utilizó el MinElute Gel Extraction Kit (QIAGEN Corporation), siguiendo las instrucciones del fabricante.
El análisis taxonómico de la secuencia problema se realizó mediante la comparación contra las bases de datos NCBI (National Center for Biotechnology Information), Greengenes (Laurence Berkeley National Laboratory) y RDP (Ribosomal Database Project). El alineamiento y generación de un árbol de distancias se hizo utilizando las secuencias con mayor similitud a la secuencia problema. Se buscaron secuencias altamente similares a las obtenidas de la página web del GenBank mediante la herramienta BLAST (BasicLocal Aligment Search Tool) de la base de datos del NCBI, usando como medida un porcentaje de identidad, el cual es mayor a medida que las secuencias sean altamente parecidas. Las secuencias de nucleótidos que codifican los genes del 16S DNAr de bacterias fueron depositadas en la base de datos de la NBCI (http://www.ncbi.nlm.nih.gov).
Levaduras
Primero se realizó el aislamiento y purificación del DNA, luego la amplificación por PCR con los iniciadores ITS4 e ITS5 de la región genética denominada ITS (Internal Transcribed Spacer) del DNA ribosomal fúngico. Posteriormente se realizó la purificación de los fragmentos de PCR y secuenciación con los iniciadores ITS1 e ITS4. Luego se siguió el mismo procedimiento utilizado para las bacterias. El análisis taxonómico de la secuencia problema se hizo mediante comparación contra las bases de datos NCBI, UNITE1 (Unified System for the DNA based fungal species linked to the classification) y Warcup Fungal ITS.
Análisis Estadístico
A los datos obtenidos se les realizó una prueba de normalidad (Shapiro-Wilk) y una prueba de homogeneidad de varianzas (Prueba de Bartlett). Seguidamente se realizó un análisis de varianza del número de unidades formadoras de colonia (UFC/ml) de los microorganismos (nivel de significancia del 95%) a las pruebas probióticas evaluadas (tolerancia a pH, tolerancia a sales biliares). Además, se realizó la prueba de múltiples rangos para verificar la diferencia estadística de medias entre concentraciones evaluadas.
Para el análisis de la prueba de tolerancia a altas concentraciones de NaCl se efectuó un análisis descriptivo por medio de gráficas de barras, basado en las UFC/ml de las cepas y las concentraciones analizadas. Así mismo, para la capacidad antagónica se realizó un análisis de varianza, teniendo en cuenta la inhibición de patógenos representada por los halos (mm) de las cepas seleccionadas frente a Salmonella sp y E. coli, con un nivel de significancia del 95%.
Los análisis estadísticos se realizaron utilizando el programa Excel 2010 y el programa Statgraphics XVI v. 16.1.15.
RESULTADOS Y DISCUSIÓN
Se aislaron nueve colonias, de las cuales siete eran bacterias ácido lácticas codificadas como: M15a, M14, M13a, M2103b, M2103c, M2104, M1103b y dos levaduras: 102M2 y 103M2.
Pruebas Probióticas
Prueba de tolerancia a cambios de pH
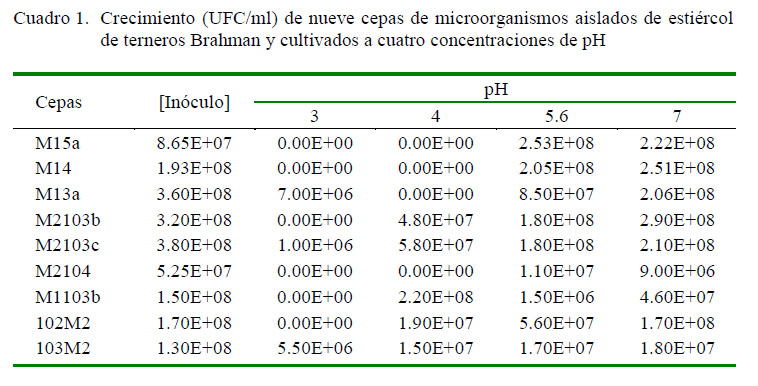
Tres de las nueve cepas aisladas lograron tolerar las condiciones de pH 3: M13a, M2103c y 103M2; mientras que las cepas M2103b, M1103b y 102M2 resistieron las concentraciones de pH 4 a 7. Por otro lado, las cepas M15a, M14 y M2104 no sobrevivieron los niveles de pH utilizados. (Cuadro 1)
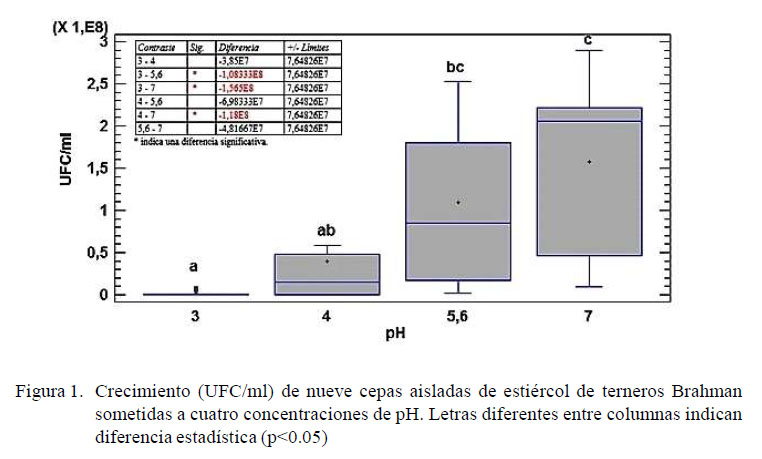
La capacidad ácido-tolerante de las bacterias M13a y M2103c es una de las características comunes entre los microorganismos del género Lactobacillus (Leveau y Bouix, 2000). Por otro lado, la habilidad de la levadura 103M2 para crecer en un amplio rango de pH puede explicarse por la existencia de sistemas intercambiadores en la membrana plasmática de las levaduras, los cuales pueden intercambiar iones a través de los sistemas secundarios (uniportadores catiónicos, simportadores catión/sustrato) (Ramírez y Peña, 2000). La prueba de múltiples rangos indicó que existe diferencia significativa (p<0.05) entre el crecimiento (UFC/ml) de las nueve cepas entre las cuatro concentraciones de pH (Figura 1).
La mayoría de las cepas tuvieron el mayor crecimiento en un pH de 7 y menor crecimiento en las concentraciones de pH ácidas, como era de esperarse, considerando que el estómago de los bovinos presenta valores de pH cercanos a 2.0, el cual funciona como barrera contra la entrada de bacterias externas dentro del tubo digestivo (Figueroa et al., 2006; Caballero, 2014). Esto demuestra que el pH juega un papel muy importante en la selección de cepas con potencial probiótico (Agarwal et al., 2000).
Prueba de tolerancia a sales biliares
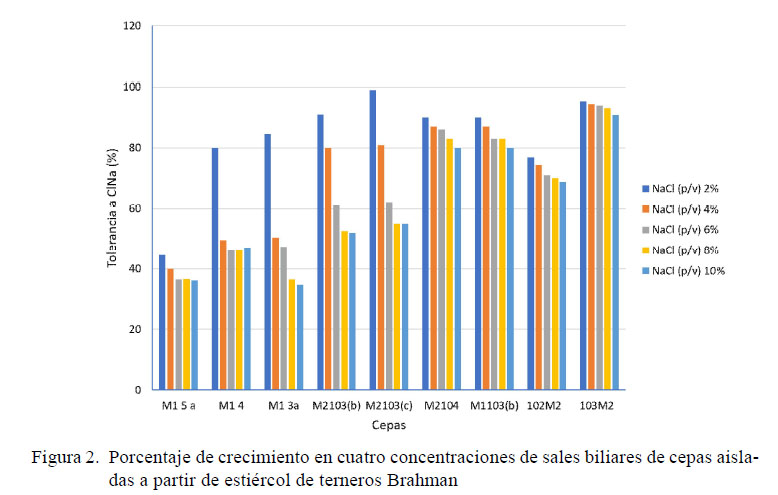
La cepa M2103c creció en las cuatro concentraciones, con porcentajes entre 18% (0.15%) y 100% (0.10%). La cepa M13a toleró concentraciones de sales biliares de 0.05, 0.10 y 0.15% con valores de 67, 39 y 56%, respectivamente, pero sin crecimiento en la concentración de 0.30%; a diferencia de la cepa 103M2 que solo desarrolló en dos concentraciones (0.05 y 0.10%) (Figura 2).
Las sales biliares representan una importante barrera antibacteriana, de allí que para la selección de bacterias probióticas se requiere un alto nivel de resistencia y de capacidad de crecimiento en este medio (Gómez et al., 1998; Kociubinski et al., 1999).
No se encontró diferencia significativa entre las medias de crecimiento (UFV/ml) de las cuatro concentraciones, de modo que estas cepas, en general, tienen la capacidad de tolerar las sales biliares, pudiendo desarrollar sus actividades metabólicas sin verse completamente inhibidas en el rumen de los bovinos (Caballero, 2014).
Prueba de tolerancia a altas concentraciones de NaCl
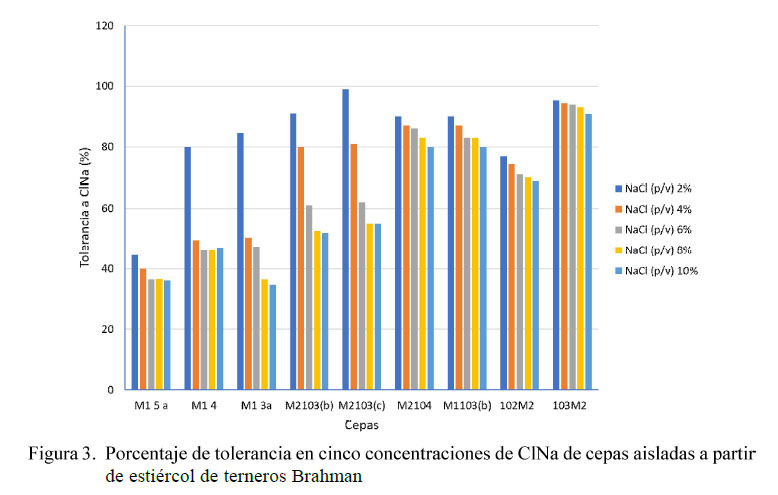
La mayor tolerancia frente al cloruro de sodio la presentó la cepa 103M2 con valores superiores a 90%, en tanto que la cepa M2103c presentó valores superiores al 50% en las cuatro concentraciones. Por otro lado, la cepa M13a disminuyó el crecimiento con el incremento de la concentración de NaCl (Figura 3). Estos resultados se deben a que la mayoría de las bacterias son relativamente insensibles a las variaciones de la presión osmótica y se adaptan a cambios fuertes en la concentración de solutos del medio, dado que poseen una pared celular mecánicamente rígida (Hernández, 1992).
La resistencia a altas concentraciones de NaCl de las cepas sin perder su capacidad de crecimiento les confiere amplias posibilidades de ser utilizadas en cultivos iniciadores en la industria láctea, como preservantes de carnes, vegetales y probióticos. Además, estos microorganismos en los alimentos conservan sus cualidades nutritivas, aumentan la vida media del producto e inhiben el desarrollo de microorganismos patógenos (O´Sullivan et al., 2002).
Selección de los Microorganismos
Tres de los nueve microorganismos aislados fueron seleccionados por pasar las pruebas de tolerancia (M13a, M2103c y 103M2) y fueron sometidas a la prueba de antagonismo. Solamente la cepa M13a no presentó halos de inhibición con alguno de los microorganismos patógenos, a diferencia de la cepa M2103c que inhibió el crecimiento de E. coli, pero no a Salmonella sp y de 103M2 que inhibió el crecimiento de ambas bacterias (p<0.05)
La capacidad de inhibición de estas bacterias puede atribuirse a que pueden producir sustancias antagónicas, tales como el ácido láctico, acético y otros compuestos volátiles que disminuyen el pH del medio, ácidos grasos de cadena corta, peróxido de hidrógeno y bacteriocinas (Zamora, 2003).
La levadura 103M2 fue la cepa con mejor desempeño inhibiendo a ambos patógenos con halos de inhibición de 8.56 y 9.50 mm. Es decir, esta cepa produce sustancias antimicrobianas que son capaces de contrarrestar in vitro el crecimiento de los patógenos evaluados, lo que confirma que el mecanismo de acción de las levaduras es diferente al de bacterias como Lactobacillus sp o Bifidobacterium sp, que producen bacteriocinas y ácido láctico (FayolMessaoidu et al., 2005).
De acuerdo con los resultados, se podrían incluir las cepas 103M2 y M2103c, para que en conjunto, superen las barreras del tránsito gastrointestinal de los terneros lactantes y puedan inhibir las especies patógenas, ya que la funcionalidad de un inóculo probiótico multicepa puede ser más efectiva y consistente que la de un monocepa (Gardiner et al., 2004; Timmerman et al., 2004; Frizzo et al., 2006).
Identificación Molecular de las Cepas
Teniendo en cuenta las pruebas probióticas se procedió a la identificación molecular de las cepas M13a y 103M2.
Cepa M13a
El clasificador de RDP pudo determinar que se trata de una secuencia de un microorganismo perteneciente al género Enterococcus con un 100% de confidencia. La base de datos de secuencias 16S RDP indica que la secuencia problema ensamblada es muy similar en la mayoría de su longitud con las cepas tipo identificadas como Enterococcus faecium. La búsqueda en la base de datos Greengenes indica que la secuencia estudiada es muy similar a secuencias de Enterococcus hirae o Enterococcus faecium. Por último, el análisis taxonómico de la secuencia problema ensamblada de 1083 pb contra la base de datos RefSeq del NCBI indica 100% de identidad en el 99% de su longitud, con secuencias del gen ribosomal 16s pertenecientes a Enterococcus faecium.
Molecularmente, la cepa M13a fue identificada como Enterococcus faecium, que pertenece al grupo de las bacterias ácidolácticas. Las cepas pertenecientes al género Enterococcus, incluidos E. faecium y E. faecalis, son conocidas por ser productoras de bacteriocinas, las cuales tienen una actividad antimicrobiana demostrada con bacterias patógenas (Zamora, 2003). Enterococcus faecium es utilizado como aditivo probiótico en animales de producción, es componente habitual de la flora intestinal, y tiene la capacidad de fijarse en la pared intestinal con alta velocidad de multiplicación, gracias a su afinidad por el medio (Flores, 2014).
Según Reyes et al. (2012), Enterococcus faecium puede ser utilizada para prevenir problemas diarreicos en cerdos jóvenes y, presumiblemente, puede tener un efecto benéfico en cerdos en la etapa de crecimiento-finalización al proporcionar un ambiente intestinal que permite mayor aprovechamiento de los nutrientes. Asimismo, se le asocia con una disminución de diarreas después del destete.
Cepa 103M2
El análisis taxonómico de la secuencia de 505 pb contra la base de datos nr/nt del NCBI indica 100% de identidad en el total de la longitud con secuencias de ITS pertenecientes a la especie Pichia kudriavzevii (Candida krusei), siendo así que se le clasifica como perteneciente a la especie Pichia kudriavzevii (Issatchenkia orientalis), cuyo anamorfo es denominado Candida krusei.
Este género no tiene antecedentes de ser utilizado como probiótico; sin embargo, debido a su naturaleza patogénica oportunista, llama la atención científica sobre la seguridad de su explotación industrial (Yadav et al., 2012).
CONCLUSIONES
-
Se logró identificar molecularmente a dos de nueve cepas aisladas. Estas fueron codificadas como M13a para Enterococcus faecium y 103M2 como Candida krusei.
-
Los resultados de pruebas in vitro demostraron que Enterococcus faecium podría ser utilizado potencialmente como probiótico.
-
E. faecium es asociado mayormente al tracto intestinal del cerdo, de allí que puede tener un efecto positivo para la disminución de diarreas posdestete.
-
Se requiere de más estudios para recomendar la cepa Candida krusei como suplemento nutracéutico.
-
Este trabajo es el primer reporte molecular de Enterococcus faecium en estiércol de terneros Brahman en el departamento de Sucre, Colombia.
LITERATURA CITADA
1. Agarwal N, Kamra D, Chaudhary L, Sahoo A, Pathak N. 2000. Selection of Saccharomyces cerevisiae strains for use as a microbial feed additive. Lett Appl Microbiol 31: 270-273. doi: 10.1046/ j.1472-765x.2000.00826.x [ Links ]
2. Ávila J, Ávila M, Tovar B, Brizuela M, Perazzo Y, Hernández H. 2010. Capacidad probiótica de cepas del género Lactobacillus extraídas del tracto intestinal de animales de granja. Rev Cient FCV-LUZ 20: 161-169. [ Links ]
3. Bazeley K. 2003. Investigation of diarrhoea in the neonatal calf. In Practice 25: 152-159. doi: 10.1136/inpract.- 25.3.152 [ Links ]
4. Caballero C. 2014. Aislamiento e identificación de bacterias ácido lácticas con potencial probiótico en bovinos Holstein. Tesis de Maestría. Montecillo, México: Colegio de Postgraduados. 85 p. [ Links ]
5. Camargo M, Párraga C, Mejía E, Escobar A, Colmenárez M. 2017. Subsistemas de crianza de becerros y su relación con el desarrollo de fincas doble propósito en el estado portuguesa. Rev Unell Cienc Tec 29: 39-46. [ Links ]
6. Fayol-Messaoidu D, Berger C, Coconnier M, Moal V, Servin A. 2005. pH-, lactic acid-, and non-lactic aciddependent activities of probiotic Lactobacilli against Salmonella enteric serovar typhimurium.Appl Env Microbiol 71: 6008-6013. doi: 10.1128/AEM.71.10.- 6008-6013.2005 [ Links ]
7. Figueroa J, Chi E, Cervantes M, Domínguez I. 2006. Alimentos funcionales para cerdos al destete. Vet Méx 37: 117-136. [ Links ]
8. Flores M. 2014. Efecto de probióticos (Saccharomyces cerevisiae y Enterococcus faecium) en el engorde y sanidad de cuyes Ayacucho. Tesis de pregrado. Ayacucho, Perú: Univ. Nacional de San Cristóbal de Huamanga. 78 p. [ Links ]
9. Frizzo L, Soto L, Bertozzi E, Sequeira E, Marti L, Rosmini M. 2006. Evaluación in vitro de las capacidades probióticas microbianas orientadas al diseño de inóculos probióticos multiespecie para ser utilizados en la crianza de terneros. Rev FAVE Cienc Vet 5: 69-80. doi: 10.14409/favecv.v5i1/2.1426 [ Links ]
10. Fuller R. 1989. Probiotics in man and animals. J Appl Bacteriol 66: 365-378. [ Links ]
11. García Y, García Y, Núñez O, Dihigo L, Moreira S, Nicoli J. 2011. Aislamiento e identificación de cepas de Lactobacillus de origen porcino con posibilidades de uso en preparados probióticos. Rev Comp Prod Porcina 18: 111-114. [ Links ]
12. Gardiner G, Casey P, Casey G, Brendan L, Lawlor P, Hill H, Fitzgerald G, et al. 2004. Relative ability of orally administered Lactoba-cillus murinus to predominate and persist in the porcine gastrointestinal tract. Appl Environ Microbiol 70: 1895-1906. doi: 10.1128/AEM.70.4.1895-1906.2004 [ Links ]
13. Gómez A, Kociubinski G, Pérez P, De Antoni G. 1998. Isolation and characterization of Bifidobacterium strains for probiotic formulation. J Food Prot 61: 865-873. [ Links ]
14. Hernández N. 1992. Efecto de la salinidad en la composición y concentración de osmoreguladores en levaduras halotolerantes. Tesis de Maestría. La Paz, México: Instituto Politécnico Nacional. 121 p. [ Links ]
15. Holdridge L. 2000. Ecología basada en zonas de vida. San José, Costa Rica: Instituto Interamericano de Cooperación para la Agricultura. 216 p. [ Links ]
16. Kociubinski G, Pérez P, De Antoni G. 1999. Screening of bile resistance and bile precipitation in lactic acid bacteria and bifidobacteria. J Food Pro 62: 905912. [ Links ]
17. Leiva S, Yañez M, Zaror L, Rodríguez H, García H. 2004. Actividad antimicrobiana de Actinomycetales aislados desde ambientes acuáticos de sur de Chile. Rev Med Chile 132: 151-159. doi: 10.4067/S0034-98872004000200003 [ Links ]
18. Leveau J, Bouix M. 2000. Microbiología industrial. Los microorganismos de interés industrial. Zaragoza, España: Ed Acribia. 595 p. [ Links ]
19. Mee J. 2008. Newborn dairy calf management. Vet Clin North Am Food Anim Pract 24: 1-17. doi: 10.1016/ j.cvfa.2007.10.002 [ Links ]
20. Mejía W, Rubio J, Calatayud D, Rodríguez A, Quintero A. 2007. Evaluación de dos probióticos sobre parámetros productivos en lechones lactantes. Zootecnia Trop 25: 301-306. [ Links ]
21. Monteagudo A, Rodríguez L, Rúa J, Martínez H, Navasa N, García M, Ferrero M. 2012. In vitro evaluation of physiological probiotic properties of different lactic acid bacteria strains of dairy and human origin. J Functional Foods 4: 531-541. doi: 10.1016/ j.jff.2012.02.014 [ Links ]
22. O´Sullivan L, Ross R, Hill C. 2002. Potential of bacteriocin-producing lactic acid bacteria for improvements in food safety and quality. Biochemie 84: 593-604. [ Links ]
23. Pascual M, Garriga M, Morfort J. 1996. Los probióticos en la alimentación animal. Eurocarne 44: 46-53. [ Links ]
24. Ramirez J, Peña A. 2000. Cation/ proton antiportersin yeats. Rev Latinoam Microbiol 42: 181-187. [ Links ]
25. Reyes I, Figueroa J, Cobos M, Sánchez M, Zamora V, Cordero J. 2012. Probiótico (Enterococcus faecium) adicionado a dietas estándar y con baja proteína para cerdos. Arch Zootec 61: 589-598. doi: 10.21071/ az.v61i236.2214 [ Links ]
26. Rondón A, Ojito Y, Arteaga F, Laurencio M, Milián G, Pérez, Y. 2013. Efecto probiótico de Lactobacillus salivarius C 65 en indicadores productivos y de salud de cerdos lactantes. Rev Cubana Cienc Agríc 47: 401-407. [ Links ]
27. Rondón A, Samaniego L, Bocourt R, Rodríguez S, Milián G, Ranilla M, Laurencio M, Pérez M. 2008. Aislamiento, identificación y caracterización parcial de las propiedades probióticas de cepas de Lactobacillus sp procedentes del tracto gastrointestinal de pollos de ceba. Cienc Tecnol Aliment 6: 56-63. [ Links ]
28. Rubio M, Hernández E, Aguirre R, Poutou P. 2008. Identificación preliminar in vitro de propiedades probióticas en cepas de S. cerevisiae. Revista MVZ Córdoba 13: 1157-1169. [ Links ]
29. Tepan R. 2011. Diarrea neonatal de los terneros. Tesis de pregrado. Cuenca, Ecuador: Univ. de Cuenca. 95 p. [ Links ]
30. Timmerman H, Koning C, Mulder L, Rombouts F, Beynen A. 2004. Monostrain, multistrain and multispecies probiotics. A comparison of functionality and efficacy. Int J Food Microbiol 96: 219-233. doi: 10.1016/j.ijfoodmicro.2004.05.012 [ Links ]
31. Yadav J, Bezawada J, Yan S, Tyagi R, Surampalli R. 2012. Candida krusei: biotechnological potentials and concerns about its safety. Can J Microbiol 58: 937-952. doi: 10.1139/ w2012-077 [ Links ]
32. Zamora L. 2003. Aislamiento identificación y conservación de cultivos de bacterias lácticas antagonistas de microbiota contaminante de sangre de matadero. Tesis Doctoral. Girona, España: Universitat de Girona. 259 p. [ Links ]
33. Zavaglia A, Kociubinski G, Pérez P, De Antoni G. 1998. Isolation and characterization of Bifidobacterium strains for probiotic formulation. J Food Protect 61: 865-873. [ Links ]
Recibido: 3 de agosto de 2017
Aceptado para publicación: 8 de marzo de 2018