Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista de Investigaciones Veterinarias del Perú
Print version ISSN 1609-9117
Rev. investig. vet. Perú vol.29 no.3 Lima July/Set. 2018
http://dx.doi.org/10.15381/rivep.v29i3.14841
ARTÍCULOS PRIMARIOS
Ausencia del virus de Necrosis Hematopoyética Epizoótica (EHNv) en truchas arcoíris (Oncorhynchus mykiss) enfermas en piscigranjas de la sierra del Perú
Absence of epizootic Hematopoietic Necrosis Virus (EHNv) in diseased rainbow trout (Oncorhynchus mykiss) from fish farms in the peruvian highlands
Karen Bautista D.1, Alberto Manchego S.1,2, Gina Castro S.1, Nieves Sandoval C.3
1 Laboratorio de Microbiología y Parasitología Veterinaria, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima, Perú
2 Laboratorio de Histología, Embriología y Patología Veterinaria, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima, Perú
3 E-mail: amanchegos@unmsm.edu.pe
RESUMEN
El objetivo del presente trabajo fue monitorear la detección del virus Necrosis Hematopoyética Epizoótica (EHNv) en truchas (Oncorhynchus mykiss) de piscigranjas de la Sierra del Perú. Se tomaron muestras de 111 peces enfermos (hígado, bazo, riñón anterior) de piscigranjas de las regiones de Áncash, Junín y Huancavelica, Perú. Las muestras de tejidos fueron procesados de inmediato para el aislamiento bacteriano y otro grupo de muestras fueron conservadas a -196 °C para el PCR. El ADN de cada tejido fue extraído mediante el método del Trizol y luego purificado con membranas de sílica (PureLinkGenomic DNA kit). El PCR se realizó utilizando los cebadores MCP-1 específicos para el EHNv siguiendo la metodología propuesta por la OIE y con el kit comercial VetPCRTM EHNV Detection para su confirmación. El aislamiento bacteriano se realizó con el sembrado en agar TSA y Anacker-Ordal (agar citófaga) de cada tejido y la identificación mediante tinción Gram y bioquímica bacteriana. No se detectó el ADN del EHNv en las muestras indicando que la prevalencia del virus es menor al 5% o no está presente en las piscigranjas muestreadas. Se aisló Yersinia ruckeri y Aeromonas spp en todas las muestras, indicando que la causa de la enfermedad y mortalidad presente en las piscigranjas se debían a la infección con estas bacterias.
Palabras clave: trucha arcoíris; virus necrosis hematopoyética epizoótica; PCR; asilamiento bacteriano
ABSTRACT
The objective of the present study was to monitor the detection of the Epizootic Hematopoietic Necrosis virus (EHNv) in trouts (Oncorhynchus mykiss) in fish farms of the highlands of Peru. Samples were taken from 111 diseased fish (liver, spleen, anterior kidney) from fish farms in the regions of Ancash, Junín and Huancavelica, Peru. The tissue samples were processed immediately for bacterial isolation and another group of samples were stored at -196 °C for PCR. The DNA of each tissue was extracted by the Trizol method and then purified with silica membranes (PureLink Genomic DNA kit). The PCR was performed using the MCP-1 primers specific for the EHNv, following the methodology proposed by the OIE and with the commercial kit VetPCRTM EHNV Detection for confirmation. Bacterial isolation was carried out by culturing on TSA and Anacker-Ordal agar (Cytophaga agar) of each tissue, and the identification by Gram stain and bacterial biochemistry. The EHNv DNA was not detected indicating that the prevalence of the virus is less than 5% or it is not present in the sampled fish farms. Yersinia ruckeri and Aeromonas spp were isolated in all the samples, indicating that the cause of the disease and mortality present in the fish farms was due to the infection with these bacteria.
Key words: rainbow trout; epizootic hematopoietic necrosis virus; PCR; bacterial isolation
INTRODUCCIÓN
La acuicultura a nivel mundial ha mostrado un incremento en la tasa anual de 8.3% durante los últimos 30 años, dentro de los cuales el sector destinado a la alimentación humana es el que ha presentado mayor desempeño. La acuicultura en el Perú está en proceso de continuo crecimiento, similar a la tendencia mundial (Kleeberg y Rojas, 2012). El pescado es actualmente el que aporta mayor contenido proteico en la dieta de la población humana a nivel mundial, superando a las aves, cerdos y vacunos (FAO, 2016).
Las enfermedades más prevalentes en piscigranjas del Perú donde se cultiva la trucha arcoíris son las de origen bacteriano. La yersiniosis causada por Yersinia ruckeri es la de mayor importancia (Sierralta et al., 2013); mientras que las infecciones producidas por bacterias del género Aeromonas son importantes a nivel mundial (Beaz-Hidalgo et al., 2010). Como medida que evite el ingreso de enfermedades exóticas a una región o país, la OIE ha determinado la promulgación de lista única de enfermedades de declaración obligatoria para animales terrestres y acuáticos, en los cuales los agentes patógenos de mayor peligro a nivel internacional deben ser monitoreados y declarados por los organismos oficiales (OIE, 2017a). Dentro de esta lista se encuentra el virus de la Necrosis Hematopoyética Epizoótica (EHNv).
El EHNv pertenece al género Ranavirus de la familia Iridoviridae que se caracteriza por su alta virulencia en truchas. Son virus relativamente grandes (120-200 nm de diámetro), de tipo icosaédricos y en algunos casos presentan una membrana lipídica interna entre el núcleo viral y la nucleocápside (Chinchar et al., 2009).
La infección por EHNv ha sido reconocida desde sus inicios enAustralia, donde fue reportada por primera vez en percas (Perca fluviatilis) y truchas arcoíris (Oncorhynchus mykiss) en brotes que se caracterizaron por la presencia de focos necróticos en los tejidos hematopoyéticos de riñones, hígado y bazo, causando altas tasas de mortalidad en juveniles. Actualmente, la enfermedad es considerada endémica en el sureste australiano, aunque con presencia en forma intermitente (Whittington et al., 1999).
No existen datos viables en los reportes de la OIE (OIE, 2017a) en América del Sur, lo cual puede indicar que el virus no está presente en las truchas o que tiene una prevalencia inferior al 5%. Debido a que el virus en la trucha arcoíris tiene un comportamiento distinto a otras especies susceptibles como la perca (Reddacliff y Whittington, 1996), es necesario identificar al virus en animales enfermos o moribundos. Estudios epidemiológicos llevados a cabo por el SENASA Argentina entre 2002 y 2013 mediante histopatología y PCR permitieron declarar al país como libre de la enfermedad en 2013 (SENASAArgentina, 2013).
En el presente estudio se buscó establecer la presencia del EHNv en peces enfermos y moribundos de piscigranjas de tres regiones de importancia en la piscicultura del Perú.
MATERIALES Y MÉTODOS
Lugar y Fecha de Estudio
El estudio fue realizado entre abril y junio de 2014. La recolección de especímenes de truchas arcoíris (Oncorhynchus mykiss) fue realizada en tres piscigranjas ubicadas en las provincias de Huancayo, Huaraz y Castrovirreyna de los departamentos de Junín, Ancash y Huancavelica, respectivamente. Las muestras se analizaron en los laboratorios de Ictiopatología y de Virología de la Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima.
Tamaño Muestral
La determinación del número de muestras mínimo para el presente estudio se basó en la información referencial publicada en el Manual de Pruebas Diagnósticas para Especies Acuáticas (OIE, 2017b). A partir de esta información, para un nivel de prevalencia de 5% y un tamaño de lote de aproximadamente 2000 especímenes, se calculó un tamaño de muestra mínimo de 60 peces. No obstante, se recolectaron 111 alevines enfermos (35 procedentes de Huaraz, 40 de Huancayo, y 36 de Castrovirreyna).
Toma de Muestras
Se realizó la captura de especímenes juveniles con sintomatología clínica de enfermedad (moribundos), en varias pozas de crianza de cada una de las piscigranjas bajo estudio y enviadas al laboratorio en bolsas de polietileno con 50% de la capacidad con aire. Para la eutanasia se procedió primeramente a baños de inmersión con hidrocloro de benzocaína (45 mg/l de agua) para la sedación completa de los peces y luego al sacrificio mediante la técnica de corte medular (Roberts y Shepherd, 1980).
La necropsia de los peces fue realizada mediante la metodología establecida para especies de laboratorio (Ostrander, 2000). Se realizó el examen in situ de los órganos internos y posteriormente se realizó la toma de muestras de hígado, bazo y riñón por animal. Se procedió al cultivo directo de las muestras para el aislamiento bacteriano y un pool por animal de una porción de cada órgano muestreado fue almacenado a -196 °C para realizar el PCR.
Aislamiento Bacteriano
Cada órgano obtenido fue procesado para el aislamiento bacteriano utilizando ansas estériles que se introdujeron directamente en el parénquima de los órganos y se sembraron en Agar Tripticasa de Soya (TSA) para el aislamiento de Yersinia ruckeri u otra bacteria Gram negativa y en agarAnacker-Ordal (agar citófaga) para aislamiento de bacterias del género Aeromonas. Las placas de agar sembradas fueron incubadas a 16 y 25 °C por 24-48 horas. Las bacterias de colonias bacterianas que crecieron fueron identificadas mediante coloración Gram y batería bioquímica.
Extracción de ADN
De los 111 peces muestreados, se extrajeron 2 g de cada órgano (riñón anterior, bazo e hígado), se colocaron en un mortero estéril con 2 ml de suero fisiológico y se machacaron hasta obtener un pool de tejidos por pez. Este machacado fue centrifugado por 5 minutos a 2500 g y el sobrenadante fue alicuotado (500 µl) en crioviales estériles de 1 ml y almacenados en congelación (-70 °C).
La extracción del ADN de cada pool se hizo a partir de las muestras mediante el método de trizol y se purificó con membranas de sílica (PureLinkGenomic DNA kit) según las indicaciones de los fabricantes, método diseñado para la extracción eficiente deADN proveniente de tejidos animales.
Reacción en Cadena de la Polimerasa (PCR)
Se realizó una PCR convencional con 5 µl de DNA por muestra para la amplificación de un fragmento de 321 pb de la proteína de la cápside del virus de Necrosis Hematopoyética Epizoótica de la familia Iridoviridae (OIE, 2012). Se utilizaron los siguientes cebadores (primers) MCP1: M151Forward (5’AAC-CCG-GCT-TTC-GGGCAG-CA-3’) y M152 Reverse (5’-CGGGGC-GGG-GTT-GAT-GAG-AT-3’) y los reactivos del kit PCR Super Mix (Invitrogen) en un volumen total de 25 µl, según indicaciones del fabricante.Así mismo, se utilizó como marcador de especie a la secuencia de amplificación de la beta-actina. Los ciclos de la reacción de la PCR fueron: 1 ciclo inicial de desnaturalización del ADN a 94 °C durante 2 min, 40 ciclos de desnaturalización durante 40 s a 94 °C, de hibridización durante 30 s a 50 °C, extensión durante 1 min a 72 °C, y de extensión final durante 5 min a 72 °C. Posteriormente, las muestras fueron incubadas a 4 °C hasta su revelado.
Luego de la amplificación del segmento de la secuencia MCP de EHNv mediante PCR, se procedió a la lectura de los productos o amplicones mediante electroforesis en geles de agarosa. En cada criovial se colocó 1.3 µl de bromuro de etidio. Se preparó, además, 2 µl de buffer de carga del marcador y 10 µl del marcador de peso molecular. Se prepararon geles de agarosa al 2% y en cada uno de los pocillos en los geles se colocaron 18 µl de la mezcla contenida en cada criovial. La corrida electroforética fue a 80v A:0.3/P3-4 por 1 h. Posteriormente, los geles fueron llevados a la campana de transiluminación para la lectura de los resultados (en función a las bandas obtenidas para la secuencia MP de EHNv).
PCR EHNV Detection Kit
Se siguieron las indicaciones del producto (BioinGentech, Chile), teniendo un amplificado de 187 pb en el control positivo y de 140 pb del control interno. Se preparó la mezcla de reacción para la muestra, el control positivo, negativo control, y el control interno, según las indicaciones del laboratorio fabricante. La reacción final debería contener 13.5 µl.
Los ciclos de la reacción de la PCR fueron: 1 ciclo inicial de desnaturalización del ADN a 94 °C durante 2 min, 30 ciclos de desnaturalización durante 30 s a 94 °C, alineación a 57 °C por 30 s, extensión a 72 °C durante 30 s y 1 ciclo de extensión final durante 5 min a 72 °C. Los productos fueron revelados por electroforesis en gel de agarosa al 1.5% conteniendo bromuro de etidio, corrida con una fuente eléctrica de 100v por 30 min.
Análisis de la Información
Los resultados del análisis de PCR a partir de las muestras de tejidos fueron interpretados como positivos o negativos a EHNv (presencia/ausencia de bandas diagnósticas en la electroforesis correspondiente a MCP).
RESULTADOS
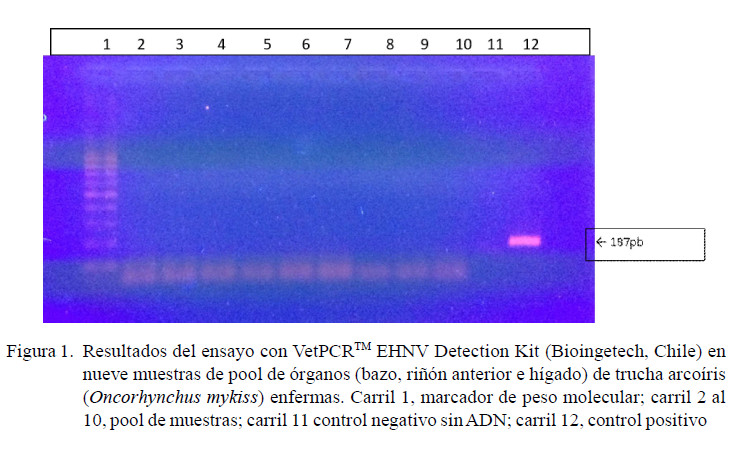
Los resultados de los ensayos de PCR no demostraron la presencia de material genético de EHNv en las 111 muestras de tejido de truchas arcoíris. En el PCR utilizando los cebadores MCP1 se obtuvieron cuatro bandas de productos inespecíficos (con tamaños diferentes a lo esperado) al revelarse en el gel de agarosa. El PCR utilizando el kit de detección EHNV de BioinGentech determinó la ausencia del genoma viral en todas las muestras, observándose la amplificación de 187 pb solo en el control positivo de la prueba (Figura 1).
Los hallazgos a la necropsia determinaron lesiones comunes, principalmente esplenomegalia y hemorragias en diversos órganos internos. El análisis bacteriológico determinó el aislamiento de agentes bacterianos de Yersinia ruckeri y Aeromonas spp en bazo e hígado de todos los peces muestreados.
DISCUSIÓN
El uso de pruebas de PCR es el método recomendado por el OIE (2012) para el diagnóstico del EHNv, y por el tipo de muestras de animales enfermos colectadas en el presente estudio, se puede estimar que las áreas muestreadas están libres del virus. Sin embargo, esta afirmación debe ser confirmada con un estudio continuo de todos los animales que mueren dentro de los patrones de «normalidad» en las piscigranjas (Whittington et al., 1994), debido a que generalmente el virus tiene una baja prevalencia y baja tasa de mortalidad (Whittington et al., 1999), pudiendo ocurrir epizootias causadas por este virus.
La ausencia del EHNv en el Perú es factible debido a que la OIE la tiene registrada como prevalente en las piscifactorías situadas en los embalses de los ríos Murrumbidgee y Shoalhaven, en Nueva Gales del Sur, Australia (Whittington et al., 2010), y no existe registros de comercio de productos acuícola entre el Perú y esta región de Australia. En Argentina, el Servicio Nacional de Sanidad y Calidad Agroalimentaria (SENASA) hizo un monitoreo de 2006 a 2008, pudiendo declarar al país en 2013 como libre de enfermedades en salmónidos, incluyendo la necrosis hematopoyética epizoótica (EHNV).
En el presente estudio, los peces estuvieron enfermos por infecciones bacterianas, habiéndose detectado microbiológicamente la presencia de las bacterias Yersinia ruckeri y Aeromonas spp en muestras de bazo e hígado, así como en algunas muestras de riñón. Estas bacterias son las más prevalentes en las piscigranjas del país (Bravo y Kojagura, 2004; Sirvas-Cornejo et al., 2011).
Durante el desarrollo del PCR utilizando los cebadores MCP propuestos por la OIE se determinó la presencia de algunas bandas inespecíficas de poco tamaño, por lo que se requirió el aumento de la temperatura de hibridación a 62 °C para reducir la amplificación inespecífica por el uso de tejidos de los peces, y que es una de las recomendaciones para el desarrollo de la prueba de PCR indicadas en el manual de pruebas de diagnóstico de la OIE (2012). Asimismo, para confirmar la negatividad de las muestras se usó el kit comercial VetPCRTM EHNv Detection Kit (BioinGentech), el cual tiene controles internos, positivos y negativos. La diferencia de estas dos pruebas moleculares se debe a las diferencias de los cebadores que amplifican regiones diferentes del genoma viral.
CONCLUSIONES
-
No se demostró la presencia de material genómico para la secuencia MCP de las especies pertenecientes a la familia Iridoviridae, ni la presencia del virus de la Necrosis Hematopoyética Epizoótica (EHNv).
-
Se indica que las especies de la familia Iridoviridae no se encuentran presentes en los sistemas de crianza de truchas evaluados en el presente estudio.
LITERATURA CITADA
1. Beaz-Hidalgo R, Alperi A, Buján N, Romalde J, Figueras M. 2010. Comparison of phenotypical and genetic identification of Aeromonas strains isolated from diseased fish. Syst Appl Microbiol 33: 149-153. doi: 10.1016/ j.syapm.2010.02.002 [ Links ]
2. Bravo S, Kojagura, V. 2004. First isolation of Yersinia ruckeri from rainbow trout (Oncorhynchus mykiss) in Peru. Bull EuropeanAssoc Fish Pathol 24: 104-108. [ Links ]
3. Chinchar VG, Hyatt A, Miyazaki T, Williams T. 2009. Family Iridoviridae: poor viral relations no longer. Curr Top Microbiol Immunol 328: 123-170. [ Links ]
4. [FAO] Organización de las Naciones Unidas para la Alimentación y la Agricultura. 2016. El estado mundial de la pesca y la acuicultura 2016. [Internet]. Disponible en: http://www.fao.org/publications/sofia/sofia/es/ [ Links ]
5. Kleeberg F, Rojas M. 2012. Pesquería y acuicultura en el Perú. Lima, Perú: Ed. Universidad de Lima. 286 p. [ Links ]
6. [OIE] World Organisation for Animal Health. 2012a. Manual of diagnostic test for aquatic animals. [Internet]. Available in: http://www.oie.int/international-standard-setting/aquatic-manual/access-online/ [ Links ]
7. [OIE] Organización Mundial de Sanidad Animal. 2017b. Código sanitario para los animales acuáticos. [Internet]. Disponible en: https://www.oie.int/doc/ged/D11947.pdf [ Links ]
8. Ostrander G. 2000. The laboratory fish. San Diego, USA: Academic Press. 678 p. [ Links ]
9. Reddacliff LA, Whittington RJ. 1996. Pathology of Epizootic Haematopoietic Necrosis virus (EHNV) infection in rainbow trout (Oncorhynchus mykiss Walbaum) and redfin perch (Perca fluviatilis L). J Comp Pathol 115: 103115. [ Links ]
10. Roberts RJ, Shepherd CJ. 1980. Enfermedades de la trucha y del salmón. España: Ed Acribia. 187 p. [ Links ]
11. [SENASA] Servicio Nacional de Sanidad y Calidad Agroalimentaria. 2013. Resolución 375-2013-SENASA-Argentina. [Internet]. Disponible en: http://www.senasa.gob.ar/resolucion3752013 [ Links ]
12. Sierralta V, León J, De Blas I, Bastardo A, Romalde J, Castro T, Mateo E. 2013. Patología e identificación de Yersinia ruckeri en trucha arco iris (Oncorhynchus mykiss) en piscigranjas de Junín, Perú. AquaTIC 38: 28-45. [ Links ]
13. Sirvas-Cornejo S, Sánchez-Robinet C, Peña-Domínguez C. 2011. Diagnóstico e identificación rápidos por PCR de Yersinia ruckeri aislada de Oncorhynchus mykiss procedentes de Canta, Lima, Perú. 2011. Rev Peru Biol 18: 349353. doi: 10.15381/rpb.v18i3.451 [ Links ]
14. Whittington RJ, Philbey A, Reddacliff GL, Macgown AR. 1994. Epidemiology of epizootic haematopoietic necrosis virus (EHNV) infection in farmed rainbow trout, Oncorhynchus mykiss (Walbaum): findings based on virus isolation, antigen capture ELISA and serology. J Fish Dis 17: 205-218. doi: 10.1111/j.13652761.1994.tb00216.x [ Links ]
15. Whittington RJ, Becker JA, Dennis MM. 2010. Iridovirus infections in finfish -critical review with emphasis on ranaviruses. J Fish Dis 33: 95-122. doi: 10.1111/j.1365-2761.2009.01110.x [ Links ]
16. Whittington RJ, Reddacliff LA, Marsh I, Kearns C, Zupanovic Z, Callinan RB. 1999. Further observations on the epidemiology and spread of Epizootic Haematopoietic Necrosis virus (EHNV) in farmed rainbow trout Oncorhynchus mykiss in southeastern Australia and a recommended sampling strategy for surveillance. DisAquat Org 35: 125-130. [ Links ]
Recibido: 5 de diciembre de 2017
Aceptado para publicación: 4 de mayo de 2018