Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.29 no.3 Lima jul./set. 2018
http://dx.doi.org/10.15381/rivep.v29i3.14831
ARTÍCULOS PRIMARIOS
Expresión y cuantificación de RORγt e IL-17 en mucosa intestinal de crías de alpacas (Vicugna pacos)
Expression and quantification of RORγt and Interleukin-17 in intestinal mucosa of newborn alpacA (Vicugna pacos)
Gustavo Murga A.1, Alberto Manchego S.1,4, Gina Castro S.1, Danilo Pezo C.3, Nieves Sandoval C.2
1 Laboratorio de Microbiología y Parasitología Veterinaria, Universidad Nacional Mayor de San Marcos, Lima, Perú
2 Laboratorio de Histología, Embriología y Patología Animal, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima, Perú
3 Estación Experimental Maranganí, Centro de Investigación IVITA, Universidad Nacional Mayor de San Marcos, Lima, Perú
4 E-mail: amanchegos@unmsm.edu.pe
RESUMEN
El objetivo del estudio fue determinar los niveles relativos de expresión de ARN mensajeros de RORγt e IL-17 en mucosa intestinal de crías de alpacas clínicamente sanas. Se formaron tres grupos etarios conformados por alpacas de 2 a 7 días (grupo 1, n=5), 8 a 20 días (grupo 2, n=6) y 26 a 47 días (grupo 3, n=6), sin distinción de sexo o raza. Se tomaronmuestras del segmento mediodel yeyuno, se extrajoelARNtotalyse empleó la técnica RT-PCR con cebadores. La expresión relativa de ARNm de RORγt e IL-17 fue determinada por el método 2-ΔΔCt usando como calibradores dos crías neonatas y empleándose como control endógeno a GAPDH. La cinética de expresión de RORγt mostró una tendencia constante durante la primera y tercera semana de vida, alcanzando su máxima expresión en los animales de 2 a 7 días de edad, a diferencia de IL-17 cuya expresión fue progresiva con la edad, evidenciándose una elevada estimulación y expresión desde los primeros días de edad.
Palabras clave: RORγt; interleucina 17; RT-PCR; cuantificación relativa; intestino; alpaca
ABSTRACT
The aim of the study was to determine the relative levels of expression of messenger RNAs of RORγt and IL-17 in intestinal mucosa of clinically healthy newborn alpacas. Three age groups were formed: 2 to 7 days (group 1, n=5), 8 to 20 days (group 2, n=6) and 26 to 47 days (group 3, n=6), regardless sex or breed. Samples were taken from the middle segment of the jejunum. The total RNA was extracted, and the RT-PCR technique was used with specific primers. The relative expression of mRNA of RORγt and IL-17 was determined by the 2-ΔΔCt method using two neonatal pups as calibrators and using GAPDH as an endogenous control. The kinetics of expression of RORγt showed a constant trend during the first and third week of life, reaching its maximum expression in animals from 2 to 7 days of age, unlike IL-17 whose expression was progressive with age, evidencing a high stimulation and expression from the first days of age.
Key words: RORγt; interleukin 17; RT-PCR; relative quantification; intestine; alpaca
INTRODUCCIÓN
Las superficies mucosas están constantemente expuestas a una gran cantidad de antígenos potencialmente perjudiciales. El único sistema de defensa que controla el balance entre la responsividad y no responsividad (tolerancia) para la defensa del hospedero consiste en una red de tejidos integrada por células linfoides, no linfoides y moléculas efectoras como los anticuerpos, quimioquinas y citoquinas (Neurath et al., 2002).
El subgrupo de células T helper 17 (Th17) aumenta el reclutamiento de neutrófilos al sitio de la inflamación para combatir patógenos bacterianos extracelulares. Este subgrupo es únicamente inducido por el estímulo combinado de células T naive (Th0) con interleucina 6 (IL-6) y el factor de crecimiento transformante beta 1 (TGF-β) mediante la activación del receptor nuclear huérfano RORγt (Zhu y Paul, 2008). Por otro lado, como parte del sistema inmune innato, las células linfoides innatas ILC3 son importantes para el desarrollo de tejidos linfoides para la contención de la microbiota, la inmunidad temprana a patógenos y la protección de células epiteliales, produciendo también IL-17 e IL-22, además de expresar el factor transcripcional RORγt (Powell y MacDonald, 2017).
Son escasos los estudios sobre las citoquinas proinflamatorias y reguladoras que participan en la inmunidad de la mucosa intestinal. Se ha estudiado la cinética de expresión in vitro de algunas interleucinas de alpacas en los últimos años, especialmente de la respuesta inmune innata (IL-1a, IL-1b, IL-6, TNF-α) (Bardález et al, 2013; Tambillo et al, 2013) y adaptativa (IL-2, IFN-γ, IL-4, IL-10) (Chiok, 2012).
Tomando estos datos, la importancia de esta citoquina y su factor de transcripción maestro en el equilibrio intestinal, el presente trabajo busca describir la cinética de expresión del ARNm de RORγt e IL-17 en la mucosa yeyunal de alpacas, con la finalidad de identificar los roles del sistema inmune en la defensa del organismo y adaptación de su entorno en los primeros 47 días de vida, periodo de gran susceptibilidad a enfermedades con elevadas tasas de mortalidad (Ameghino y DeMartini, 1991).
MATERIALES Y MÉTODOS
Procedencia de la Muestra
Las muestras fueron tomadas de alpacas de la Estación Experimental Maranganí del Centro de Investigación IVITA, en el departamento de Cusco, Perú, entre enero y marzo de 2015.
Se establecieron tres grupos etarios: G1, crías de 2 a 7 días de edad; G2, crías de 8 a 20 días; G3, crías de 26 a 47 días; cada uno constituido por seis crías de alpaca (exceptuando el primer grupo etario formado por cinco animales) que no manifestaron signos de enfermedad. Los calibradores (n=2) corresponden a neonatos de alpacas que estaban exentos de los componentes transferidos a través de la inmunidad pasiva, lográndose esto mediante la remoción inmediata de la cría en el momento del parto.
Manejo de las Muestras
Los animales fueron manejados según el protocolo de autorización N.° 2009-001 del Comité de Ética y Bienestar Animal de la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos, utilizándose para el sacrificio 1.5 mg/kg de xilacina y 7.5 mg/kg de ketamina, vía intramuscular, seguido de una sobredosis de 50 mg/kg, vía endovenosa, de pentobarbital sódico.
Durante la necropsia, se tomó una porción de 2 cm de longitud del yeyuno de cada animal. Estos segmentos fueron lavados con suero fisiológico al 0.9% para retirar contenido intestinal y fueron almacenados en nitrógeno líquido (-196 °C) inmediatamente después del lavado.
Las muestras fueron procesadas en el Laboratorio de Inmunología de la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos. Las muestras, una vez retiradas del tanque de nitrógeno líquido, se cortaron en tamaños que estuvieron entre 100 y 150 mg. Se trituraron en un mortero y pilón estériles, manteniéndose a temperatura de congelación a -20 °C en todo el proceso con la constante adición de nitrógeno líquido. Para la extracción delARN total se empleó el método combinado de «Trizol® Reagent» y el kit comercial «PureLink iT™ RNAAssay kit» (Invitrogen, EEUU). El ARN obtenido se cuantificó usando el kit Quant-it™ (Invitrogen, EEUU) siguiendo las instrucciones del fabricante y usando el fluorómetro Qubit™ (Invitrogen, EEUU). Luego, se procedió a realizar diluciones en agua libre de nucleasas para trabajar con 1 µg en un volumen de 2 µl por muestra y usarla en el RT-PCR tiempo real.
Elección de Oligonucleótidos
Se diseñaron los oligonucléotidos cebadores con base de secuencias de alpaca publicadas en el Genbank usando el programa BLAST del NCBI (www.ncbi.nlm.nih.gov/blast/) (Cuadro 1).
PCR Tiempo Real
Se empleó el ADNc de cada una de las muestras como templado para la reacción de PCR con el kit SuperScrip III First-Strand Synthesis SuperMix for qRT-PCR (Invitrogen, EEUU), siguiendo las recomendaciones del fabricante.
Para la reacción del PCR Tiempo Real se utilizó el kit SYBR® GreenER™ qPCR SuperMix Universal y el termociclador Applied Biosystem® 7500 Real-Time PCR System con el siguiente protocolo: 1 ciclo de 50 °C por 2 min (incubación de Uracilo-DNA Glicosilasa [UDG]), 1 ciclo de 95 °C por 10 min(inactivación deUDGyactivaciónde DNA polimerasa), seguido de 45 ciclos de 95 °C por 15 s (denaturalización), 60 °C por 60 s (hibridación y extensión) y lectura de placa. La temperatura de melting o curva de disociación de los productos amplificados se analizó a partir de 65 hasta 95 °C con lectura de placa cada 0.3 °C. Finalmente, los productos fueron conservados a 8 °C. Se utilizó el software Applied Biosystems 7500 v. 2.0.1 para analizar la curva de amplificación (Ct) y la temperatura de disociación (Tm) de los productos amplificados.
Análisis de Expresión del ARNm
Se usó el método comparativo 2-ΔΔCt (Livak y Schmittgen, 2001), empleando como calibrador el grupo de dos neonatos (0 días de edad) y como control endógeno el gen de gliceraldehído-3-fosfato deshidrogenasa (GAPDH).
Análisis Estadístico
Se utilizó la prueba de Shapiro-Wilk para determinar si las variables siguen la distribución normal. Los valores de 2-ΔΔCt de las muestras se usaron para calcular la varianza, desviación estándar (DE) y coeficiente de variación (CV). En las variables que siguieron la distribución normal se realizó la prueba de análisis de varianza de un factor y la prueba post-hoc de Bonferroni, en tanto que para las variables que no siguen la distribución normal se usó la prueba de Kruskal-Wallis y U de Mann-Whitney. En los análisis estadísticos se empleó el programa STATA v. 14, con una significación estadística de 0.05.
RESULTADOS
Se analizó la expresión de ARNm del gen de GAPDH (usado como control endógeno), así como de los genes RORγt e IL-17, encontrando niveles detectables de ARNm en todas las muestras.
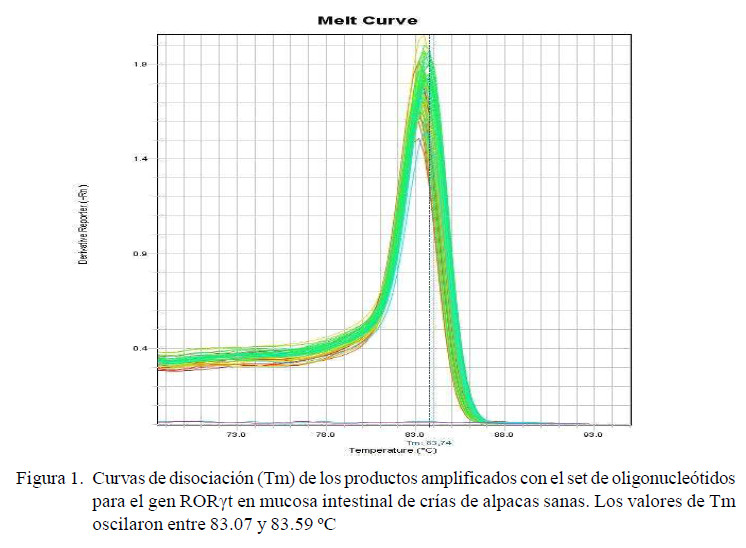
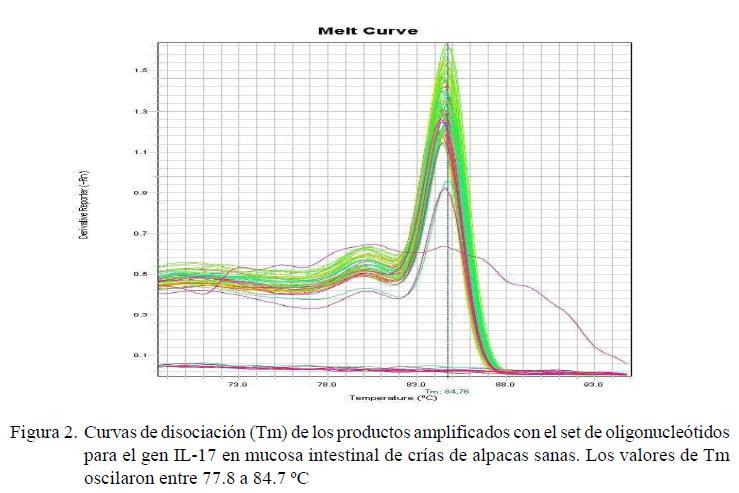
Los valores de la curva de disociación o temperatura de melting (Tm) de los productos amplificados mostraron un pico único en cada una de las muestras, presentando un rango de temperaturas que osciló entre 84.3 a 86.54 °C para GAPDH, de 83.07 a 83.59 °C para RORγt (Figura 1) y de 77.8 a 84.7 °C para IL-17 (Figura 2), tal y como se predijo en el diseño de los cebadores, demostrando la amplificación de productos únicos y específicos.
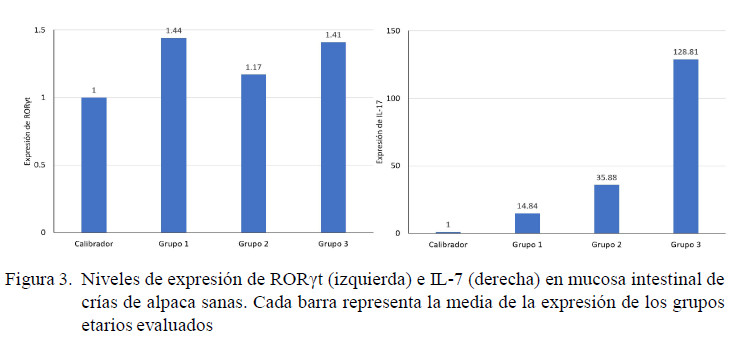
Los resultados de la cuantificación relativa mostraron niveles deARNm de RORãt constantes con la edad entre el primer y tercer grupo etario, encontrado valores promedio de 1.44 ± 0.36 en crías de 2 a 7 días (grupo 1), 1.17 ± 0.75 en crías de 8 a 20 días (grupo 2) y 1.41 ± 1.22 en crías de 26 a 47 días (grupo 3), teniendo como unidad lo expresado por el grupo calibrador, sin mostrar diferencia significativa entre grupos (Figura 3).
Los resultados de la cuantificación relativa mostraron niveles de ARNm de IL-17 ascendentes con la edad, encontrando valores promedio de 14.84 ± 13.72 (grupo 1), 35.88 ± 22.18 (grupo 2) y 128.81 ± 142.20 (grupo 3) veces lo expresado con respecto al grupo calibrador, sin mostrar diferencia estadística significativa entre grupos (Figura 3).
DISCUSIÓN
Los resultados muestran una expresión constante de RORγt y un incremento de la expresión de IL-17 con la edad en la mucosa del yeyuno de las crías de alpaca. Este factor de transcripción maestro (RORγt) participa expresándose en células linfoides innatas tipo 3 (ILC3) y varios subgrupos de células T como las células Th17, células NKT invariantes, células γδ y un subgrupo de neutrófilos productores de IL-17, documentado en humanos y ratones, con el fin de inducir el desarrollo de tejidos linfoides en el feto y en la lámina propia intestinal y activación de células B después del nacimiento (Eberl, 2016).
Los resultados obtenidos de RORγt pueden asociarse a una respuesta inmunológica transitoria debido a la presencia de nuevos antígenos alimenticios y microbiológicos generando un descenso en la expresión de esta a partir de la segunda semana. Resultados similares pueden apreciarse en otros trabajos donde se estudia la expresión de citoquinas de los grupos Th1 y Th2 en la mucosa intestinal de alpacas durante el primer mes de vida (More et al., 2011; Chiok, 2012; Burga, 2016).
RORγt es inducido en células Th17 en desarrollo mediante la activación del transductor de señal y activador de la transcripción 3 (STAT3) (Harris et al., 2007; Mathur et al., 2007). Hay un enriquecimiento de las células Th17 en el intestino (Lochner et al., 2008), donde los activadores de STAT3, tales como IL-6, IL-21 e IL-23 abundan, presuntuosamente en respuesta a grandes comunidades bacterianas residiendo en el lumen intestinal.
El descenso en el segundo grupo puede estar relacionado con la microbiota y la inmunidad adaptativa. Estas reprimen la actividad de las ILCs RORγt+ mediante competición por un nicho de citoquinas en común. Ambos tipos de células expresan, tanto IL17 como IL-22, de modo que podrían estar sujetas a similares regulaciones por un panel de citoquinas que incluyen IL-6, IL-21, IL23, IL-25 e IL-27 (Dong, 2008).
La expresión ascendente mostrada por IL-17 se asocia a múltiples factores, pero principalmente a la exposición de las crías a una gran cantidad de bacterias extracelulares como E. coli, Klebsiella pneumoniae, Clostridium perfringens, Streptococcus pyogenes, Staphylococcus aureus, Vibrio cholerae, Mycoplasma pneumoniae, Candida albicans, Coccidioides posadasii, Histoplasma capsulatum y Blastomyces dermatitidis (Chen y Kolls, 2017).
La expresión ascendente con la edad en este estudio puede estar relacionada a que RORγt no es el único factor de transcripción involucrado en la expresión de esta citoquina. Estudios han demostrado un rol crítico de STAT3 (Mathur et al., 2007) y RORC (gamma del receptor huérfano relacionado con RAR) (Ivanov et al., 2006) en el desarrollo del linaje Th17 en ratones y humanos.
La proteína Runx1 puede regular la transcripción de una manera dependiente de contexto mediante la unión a otros factores transcripcionales para formar coactivadores o complejos co-represores. Runx1 actúa no solo junto a RORγt previamente sintetizado, sino también induce nueva síntesis de RORγt (Zhang et al., 2008).
Otro receptor nuclear de ácido retinoico relacionado, RORγ, es también expresado en células Th17, tanto in vitro como in vivo. La expresión de RORγ es inducida por TGF-β e IL-6 mediante la vía STAT3. Las células T CD4+ deficientes en RORá poseen una diferenciación alterada de Th17, pero no totalmente eliminada. La deficiencia de RORγ daña la expresión de IL-17A, pero no de IL17F (Ivanov et al., 2009). Otros estudios han demostrado, además, que múltiples factores de transcripción también participan en la regulación del desarrollo de células Th17. Estas incluyen IRF4, BATF y RUNX1 (Kurebayashi et al., 2000).
Las alpacas nacen vulnerables e hipogammaglobulinémicas debido al tipo de placentación epiteliocorial difusa, teniendo como primeras líneas de defensa las respuestas inespecíficas innatas como lisozimas, lactoferrina, complemento (Fernández et al., 1994). Posteriormente, se inicia la respuesta de mucosa propia de la cría, por lo que se debe tener en cuenta la influencia de factores inmunoestimuladores como el calostro, que además de brindar a los neonatos la principal fuente de protección contra patógenos al contener principalmente IgG (Whitehead, 2009), también contiene factores de protección inespecíficos y células inmunes maternas viables incluyendo linfocitos T y B, neutrófilos y macrófagos, que realizan la función de estimular el desarrollo inmunológico (Le Jan, 1996).
En este trabajo los Tm de IL-17 fueron las de mayor variación. Es posible que estas diferencias sean resultados de las variaciones polimórficas o isoformas del gen, debido a que está encontrándose muchas isoformas de esta citoquina, ya que en la actualidad se indica que existe una familia de proteínas similares en conformación y secuencias aminoacídicas (Aggarwal y Gurney, 2002). No obstante, en el presente trabajo no se pudo establecer con certeza la existencia de polimorfismos o isoformas procedentes de mecanismos de recombinación génica propias de la generación de ARNm de esta citoquina. Es necesario la realización del secuenciamiento nucleotídico de los productos del RT-PCR tiempo real para identificar tales variaciones y emplear técnicas avanzadas para develar las implicancias de tales cambios y sus efectos sobre los individuos en caso se presenten.
CONCLUSIONES
La expresión de RORγt en crías de alpacas clínicamente sanas se mostró constante durante la primera y tercera semana de vida, alcanzando su máxima expresión en el grupo de animales de 2 a 7 días de edad.
La expresión de IL-17 en crías de alpaca clínicamente sanas manifestó una expresión progresiva con la edad, alcanzando su máximo valor en los animales de 26 a 47 días de edad estudiado.
Agradecimientos
Este trabajo fue financiado por el Programa de Innovación para la Competitividad y Productividad (INNOVATE – Perú) a través del Proyecto N.° 180 FINCyT-IB-2013.
LITERATURA CITADA
1. Aggarwal S, Gurney A. 2002. IL-17: prototype member of an emerging cytokine family. J Leukoc Biol 71: 1-8. doi: 10.1189/jlb.71.1.1 [ Links ]
2. Ameghino E, DeMartini J. 1991. Mortalidad en crías de alpacas. Bol Div IVITA, UNMSM. Lima. 71 p. [ Links ]
3. Bardález C, Manchego S, Chiok KL, Sandoval C, More B, Pezo D, Ramírez V. 2013. Cinética de expression del factor de necrosis tumoral alfa (TNF-α) e interleucina 1 alfa (IL-1α) en mucosa intestinal de crías de alpaca (Vicugna pacos) sanas y con enteropatía. Rev Inv Vet Perú 24: 381-389. [ Links ]
4. Burga CC. 2016. Expresión de citoquinas de la respuesta Th2 en mucosa intestinal de crías de alpaca. Tesis de Maestría. Lima, Perú: Univ. Nacional Mayor de San Marcos. 86 p. [ Links ]
5. Chen K, Kolls JK. 2017. Interleukin- 17A (IL-17A). Gene 614: 8-14. doi: 10.1016/j.gene.2017.01.016 [ Links ]
6. Chiok KL. 2012. Expresión de citoquinas de las respuestas Th1 (IFN-γ e IL-2) y Th2 (IL-4 e IL-10) en mucosa intestinal de crías de alpaca (Vicugna pacos) sanas y con enteropatía. Tesis de Maestría. Lima, Perú: Univ. Nacional Mayor de San Marcos. 126 p. [ Links ]
7. Dong C. 2008. Th17 cells in development: an updated view of their molecular identity and genetic programming. Nat Rev Immunol 8: 337-48. doi: 10.1038/nri2295 [ Links ]
8. Eberl G. 2016. RORãt, a multitask nuclear receptor at mucosal surfaces. Mucosal Immunol 10: 27-34. doi: 10.1038/mi.2016.86 [ Links ]
9. Fernández AS, Padola NL, Esterin SM. 1994. El calostro, fuente de transferencia de la inmunidad materna. Cienc Vet Córdoba 22: 1-5. [ Links ]
10. Harris TJ, Grosso JF, Yen HR, Xin H, Kortylewski M, Albesiano E, Hipkiss EL, et al. 2007. Cutting edge: an in vivo requirement for STAT3 signaling in Th17 development and Th17- dependent autoimmunity. J Immunol 179: 4313-4317. doi: 10.4049/jimmunol.-179.7.4313 [ Links ]
11. Ivanov II, McKenzie SB, Zhou L, Tadakoro EC, Lepelley A, Lafaille JJ, Cua JD, Littman RD. 2006. The orphan nuclear receptor RORγt directs the differentiation program of proinflammatory IL-17+ T helper cells. Cell 126: 1121-1133. doi: 10.1016/j.cell.2006.-07.035 [ Links ]
12. Ivanov II, Atarashi K, Manel N, Brodie EL, Shima T, Karaoz U, Wei D, et al. 2009. Induction of intestinal Th17 cells by segmented filamentous bacteria. Cell 139: 485-498. doi: 10.1016/j.cell.2009.09.033 [ Links ]
13. Kurebayashi S, Ueda E, Sakaue M, Patel DD, Medvedev A, Zhang F, Jetten MA. 2000. Retinoid-related orphan receptor γ (RORγ) is essential for lymphoid organogenesis and controls apoptosis during thymopoiesis. Proc Natl Acad Sci USA 97: 10132-10137. [ Links ]
14. Le Jan C. 1996. Cellular components of mammary secretions and neonatal immunity: a review. Vet Res 27: 403-417. [ Links ]
15. Livak KJ, Schmittgen TD. 2001. Analysis of relative gene expression data using real time quantitative PCR and the 2-ΔΔCt method. Methods 25: 402-408. doi: 10.1006/meth.2001.1262 [ Links ]
16. Lochner M, Peduto L, Cherrier M, Sawa S, Langa F, Varona R, Riethmacher D, et al. 2008. In vivo equilibrium of proinflammatory IL-17+ and regulatory IL-10+ Foxp3+ RORγt+ T cells. J Exp Med 205: 1381-93. doi: 10.1084/jem.20080034 [ Links ]
17. Mathur AN, Change HC, Zisoulis DG, Stritestky GL, Yu Q, O’Malley JT, Kapur R, et al. 2007. Stat3 and stat4 direct development of IL-17-secreting Th cells. J Immunol 178: 4901-4907. doi: 10.4049/jimmunol.178.8.4901
18. More J, Manchego A, Sandoval N, Ramírez M, Pezo D, Chiok KL, Rivera H. 2011. Detección genómica y expresión de péptidos antimicrobianos (α y β defensinas) en mucosa intestinal de crías de alpacas (Vicugna pacos). Rev Inv Vet Perú 22: 324-335. doi: 10.15381/ rivep.v22i4.332 [ Links ]
19. Neurath MF, Finott S, Glimcher LH. 2002. The role of Th1/Th2 polarization in mucosa immunity. Nat Med 8: 567- 573. doi: 10.1038/nm0602-567
20. Powell N, MacDonald TT. 2017. Recent advances in gut immunology. Parasite Immunol 39: e12340. doi: 10.1111/pim.12430
21. Tambillo L, Manchego S, Chiok KL, Sandoval N, More J, Rivera H. 2013. Evaluación in vitro de la respuesta leucocitaria de alpacas (Vicugna pacos) en presencia de antígenos clostridiales. Rev Inv Vet Perú 24: 510-523. doi: 10.15381/rivep.v24i4.2742 [ Links ]
22. Whitehead CE. 2009. Management of neonatal llamas and alpacas. Vet Clin North Am Food Anim Pract 25: 353-366. doi: 10.1016/j.cvfa.2009.03.006 [ Links ]
23. Zhang F, Meng G, Strober W. 2008. Interactions among the transcription factors Runx1, RORγt and Foxp3 regulate the differentiation of interleukin producing T cells. Nat Immunol 9: 1297- 1306. doi:10.1038/ni.1663 [ Links ]
24. Zhu J, Paul EW. 2008. CD4 T cells: fates, functions and faults. Blood 112: 1557-1569. doi: 10.1182/blood-2008-05- 078154 [ Links ]
Recibido: 16 de diciembre de 2017
Aceptado para publicación: 19 de junio de 2018