Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.29 no.4 Lima oct./dic. 2018
http://dx.doi.org/10.15381/rivep.v29i4.15188
ARTÍCULOS PRIMARIOS
Variabilidad genética del caballo (Equus caballus) mediante genes del pelaje en Sahagún, Córdoba, Colombia
Genetic variability of the horse (Equus caballus) using coat genes in Sahagún, Córdoba, Colombia
Cristian Moreno1, Luis Causil1, Enrique Pardo1,2
1 Departamento de Biología, Facultad de Ciencias Básicas, Universidad de Córdoba, Colombia
2 Email: epardop@correo.unicordoba.edu.co
RESUMEN
El objetivo de este estudio fue estimar la variabilidad genética de caballos criollos (Equus caballus) mediante genes asociados al pelaje. Se realizaron muestreos mediante excursiones rurales entre noviembre de 2016 y noviembre de 2017 en siete poblaciones en Sahagún, Córdoba. Se muestrearon animales adultos presentes en las fincas de cada población, donde se caracterizó fenotípicamente cada animal atendiendo a los marcadores autosómicos de codificación morfológica: Extension, Agouti, Cream, Gray, White, Tobiano, Overo,y Roan. Los parámetros genéticos como frecuencia alélica, diversidad genética y distancia genética fueron calculados a través del programa PopGene 1.31; la estructura poblacional se determinó mediante el programa FSTAT v. 2.9.3.2 y el dendrograma se realizó utilizando el programa MEGA 7. Los resultados indicaron no presencia para los marcadores White y Overo, mientras que el marcador Extension fue el de mayor frecuencia posiblemente favorecido por selección artificial, debido a temperamento y astucia. El marcador Tobiano fue el menos frecuente, hecho que estaría relacionado con la selección en contra de individuos que porten este marcador. Las poblaciones se encontraron en equilibrio Hardy-Weinberg, al igual que mostraron un exceso de heterocigotos y altos valores de flujo genético entre ellas.
Palabras clave: alelo; diversidad genética; heterocigosidad; distancia genética; equilibrio Hardy-Weinberg
ABSTRACT
The aim of this study was to estimate the genetic variability of domestic Creole horses (Equus caballus) through genes associated with the coat. Samples were taken through rural excursions between November 2016 and November 2017 in seven populations in Sahagun, Cordoba. Adult horses present in the farms of each population were sampled, where each animal was phenotypically characterized according to the autosomal markers of morphological coding: Extension, Agouti, Cream, Gray, White, Tobiano, Overo, and Roan. Genetic parameters: allele frequencies, genetic diversity and genetic distance were calculated through the program PopGene 1.31; population structure was determined by the program FSTAT v.2.9.3.2. and the dendrogram was done using the program Mega 7. The results indicated no presence for the White and Overo markers while Extension marker was the most representative in frequency possibly helped by artificial selection. Tobiano marker was the least frequent, which might be related to selection against individuals carrying this marker. Populations were found in Hardy-Weinberg equilibrium, as they showed an excess of heterozygotes and high values of genetic flow between them.
Keywords: allele; genetic diversity; heterozygosity; genetic distance; Hardy Weinberg equilibrium
INTRODUCCIÓN
La genética del pelaje en animales domésticos es un antiguo modelo de sistema para estudiar los aspectos fundamentales de acción e interacción de genes. La variación en el pigmento generalmente implica los continuos cambios en la biología, evolución e historia de la domesticación de la población animal (Imsland et al., 2016). Las poblaciones actuales de caballo criollo americano (Equus caballus) y, específicamente del caballo criollo colombiano, son el resultado de poblaciones expuestas a sucesivos cambios a través de la historia, introducidos por conquistadores europeos. Es por esta razón que sería de esperar que los caballos criollos estuvieran relacionados genéticamente con las razas andaluzas, españolas de origen celta, berberiscas y árabes (Giovambattista et al., 2010).
Los criadores de caballos consideran que el color del pelaje se asocia con el rendimiento del animal. Tal conexión sería el resultado de la interacción de los genes que controlan el color del pelaje, y al mismo tiempo estarían vinculados con los genes responsables para el rendimiento. En forma similar, también se producen efectos pleiotrópicos por causa de genes responsables de la coloración; por ejemplo, algunos fenotipos no melanóticos en el caballo se asocian con rasgos letales o semiletales (Stachurska y Jansen, 2015). El caballo criollo (Equus caballus), por ser una especie doméstica sometida a continuas presiones antrópicas, se caracteriza por su alto poder reproductivo y baja variabilidad genética (Librado et al., 2016).
Los colores pueden ser utilizados como una especie de marcador de posibles tendencias deseables o indeseables en las razas conservadas. En Valencia, Córdoba, se evaluó la variabilidad genética en cinco poblaciones de caballos criollos utilizando marcadores de pelaje (Yepes et al., 2017), mientras que, en Ciénaga de Oro, Córdoba, se estudió la estructura genética de siete poblaciones de caballos criollos (Correa et al., 2015). La importancia de este tipo de investigaciones se basa en que la diversidad de las especies domésticas es considerada como un importante componente de la biodiversidad. De igual forma, el pool génico de las razas criollas es importante por la conservación de genes, que son consideradas como un valioso patrimonio cultural, histórico y genético de los países en América. No obstante, las razas criollas han sufrido una drástica reducción poblacional y un detrimento en su grado de pureza, razón por la cual muchas de ellas están en riesgo de extinción (Giovambattista et al., 2010).
Razón por el cual, el objetivo de esta investigación fue evaluar la variabilidad genética del caballo doméstico (Equus caballus) usando genes asociados al pelaje en Sahagún, Córdoba.
MATERIALES Y MÉTODOS
El estudio se realizó en la zona rural de Sahagún (09º 13’ 54" Latitud Norte y 75º 49’11" Longitud Oeste), Colombia, con una temperatura ambiental promedio de 28 °C. Se muestrearon las poblaciones equinas de Los Galanes, Catalina, El Viajano, La Floresta, Las Llanadas, Los Amarillos y Rodania.
Los muestreos se realizaron entre noviembre de 2016 y noviembre de 2017 mediante excursiones rurales. Se muestrearon animales adultos presentes en las fincas de cada población, donde se caracterizó fenotípicamente cada animal atendiendo a los marcadores autosómicos de codificación morfológica Extension, Agouti, Cream, Gray, White, Tobiano, Overo y Roan, siguiendo la metodología propuesta por Bartolomé et al. (2008).
Se calcularon las frecuencias alélicas por marcador a nivel poblacional y a nivel global, al igual que las medidas de diversidad genética de Nei (Nei, 1972) como diversidad genética total (HT), diversidad dentro de las poblaciones (HS), diversidad entre poblaciones (DST), coeficiente de diversidad genética (GST), flujo génico (Nm), equilibrio Hardy-Weinberg y distancia genética entre las poblaciones, mediante el programa PopGene 1.31 (Yeh et al., 1999). La estructura genética de las poblaciones, atendiendo a los índices de fijación propuestos por Wright (FIS, FIT y FST) se calculó mediante el programa FSTAT v. 2.9.3.2 (Goudet, 1995). El dendograma se obtuvo a partir de la matriz distancia genéticas, mediante el programa MEGA7 (Kumar et al., 2016).
RESULTADOS Y DISCUSIÓN
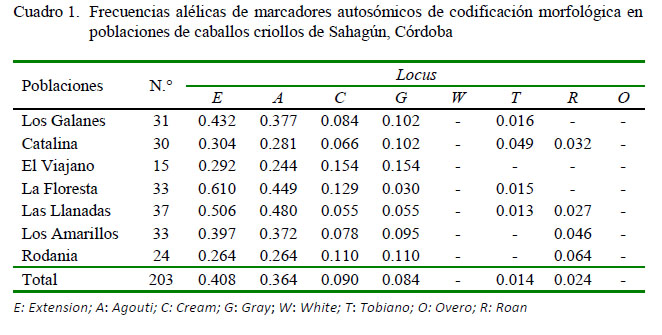
Se muestrearon 203 individuos en las siete poblaciones: Los Galanes (n=31), Catalina (n=30), El Viajano (n=15), La Floresta (n=33), Las Llanadas (n=37), Los Amarillos (n=33), Rodania (n=24). Al calcular las frecuencias alélicas para cada población (Cuadro 1), se observó que el marcador Extension fue el más frecuente (p=0.4086), específicamente en La Floresta (p=0.6108), Las Llanadas (p=0.5068) y Los Galanes (p=0.4320). En el caballo, los colores básicos, castaño, bayo y negro, se determinan por cuatro alelos, dos codificados por el locus Extension (E) (EE y Ee) y dos codificados por el locus Agouti (A) (AA y Aa). Castaño y negro siguen un modo recesivo de la herencia (EE / Ee y AA / Aa), siendo el castaño epistático sobre negro (Bellone, 2010). Esta interacción génica podría estar favoreciendo la expresión del marcador Extension. Igualmente, Finn et al. (2016) mostraron que existe una relación entre fenotipos del pelaje y comportamiento en caballos, revelando que los caballos castaños exhibieron mayor audacia y menor temor cuando fueron expuestos a objetos y animales no frecuentes en su ambiente.
Los resultados del presente estudio concuerdan con las investigaciones realizadas en Ciénaga de Oro y Valencia, Córdoba, donde Extension mostró las frecuencias más altas y sus resultados se basan en que variantes alélicas responsables de las coloraciones oscuras se ven favorecidas en comparación con aquellas que expresan tonalidades claras. Además, la capacidad de absorción de radiación calórica es más eficiente en animales de capa oscura que en aquellos de tonalidades claras, lo que permite suponer que los caballos oscuros están mejor adaptados a las condiciones imperantes en el clima tropical (Correa et al., 2015; Yepes et al., 2017).
Por otra parte, los marcadores Overo y White estuvieron ausentes en todas las poblaciones. La ausencia de ellos podría obedecer a la relación que existe entre esos marcadores y algunas anomalías o letalidades que pueden expresarse en condición de homocigosis; por ejemplo, el marcador White se hereda como un rasgo autosómico dominante. Se demostró que el fenotipo White es heterocigoto obligado (W / +) debido que en condición de homocigosis (WW) es letal en las primeras etapas del desarrollo embrionario (Haase et al., 2007; Stachurska y Ussing, 2007). Algo similar ocurre para el marcador Overo, el cual en estado homocigoto producen el síndrome letal de Overo Blanco o también llamada agangliosis, donde los potros con esta condición en su primer día de vida sufren cólicos como resultado de la obstrucción intestinal causada por ausencia de nervios intestinales (Charon y Lipka 2015).
El marcador Tobiano fue de los menos frecuente (p=0.0149). Solo se registró en cuatro poblaciones y hubo ausencia en las localidades de El Viajano, Los Amarillos y Rodania. El patrón de pelaje tobiano se hereda como un rasgo autosómico dominante. En un estudio realizado recientemente se encontró que la frecuencia del gen Tobiano fue baja (4.2%) y su avistamiento en ocho razas identifican al marcador Tobiano como más común en caballos domésticos que no han perdido los rasgos nativos. Es así que la ausencia de este marcador en la mayoría de las razas de caballos modernas es probablemente el producto de la selección en contra de individuos que porten este marcador (Reissmann et al., 2016). Otro marcador de baja frecuencia fue Roan (p=0.0249). El gen Roan segrega de manera dominante y en condición de homocigosis expresa letalidad (Rieder, 2009); razón por la cual exhibe una baja frecuencia.
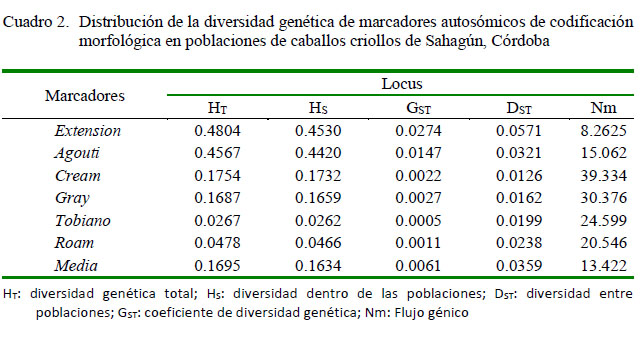
Para cada marcador, el nivel medio de variabilidad genética (Cuadro 2) fue bajo (HT=0.1695) siendo los marcadores Extension (HT=0.4804) y Agouti (HT=0.4567) los de mayor heterocigosidad; mientras que Tobiano (HT=0.0267) mostró la menor heterocigosidad. La diversidad dentro y entre las poblaciones fue baja, justificada por un alto flujo génico (Nm=13.4224). En este estudio el valor de diversidad genética total (HT) fue similar a los reportados en Ciénaga de Oro y Valencia (Correa et al., 2015; Yepes et al., 2017). El bajo valor de HT obtenido en Sahagún puede deberse a la poca variación entre las poblaciones analizadas, resultado del elevado movimiento de los individuos, demostrado por el alto flujo genético. Además, la baja diversidad genética encontrada en este estudio, pudiera ser el resultado de una continua selección artificial, teniendo en cuenta, el rol que ha desempeñado el caballo criollo (Equus caballus); razón por la cual el humano a través de la historia ha podido seleccionar animales de acuerdo a su habilidad, disposición, audacia y resistencia física, dando lugar a grupos específicos de caballos de características distintivas que se han podido transmitir a la descendencia (Gastaldo et al., 2017).
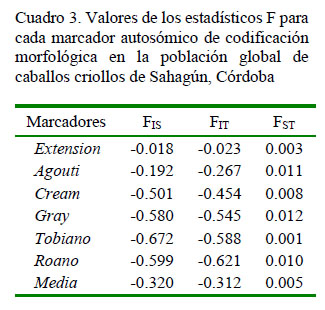
Los valores negativos obtenidos para el estadístico FIS (Cuadro 3) evidencian un exceso de heterocigotos de los individuos con respecto a cada población, indicando ausencia de consanguinidad, con valores que fluctuaron entre -0.018 para el marcador Extension y -0.672 para el gen Tobiano. En el estadístico FIT se obtuvo también un exceso de heterocigotos de individuos con respecto a la población total, con valores entre -0.023 y -0.621 para los marcadores Extension y Roan, respectivamente. Por otra parte, el valor promedio de FST resultó bajo, lo que indica escasa diferenciación génica entre poblaciones. En este estudio, el exceso de heterocigotos estaría relacionado con la existencia de un elevado intercambio de genes, lo cual evita eventos de endogamia dentro de las poblaciones, asumiéndose ausencia de consanguinidad entre los individuos de cada población.
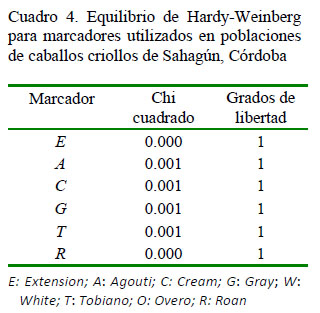
El estudio mostró a todas las poblaciones en equilibrio de Hardy-Weinberg (Cuadro 4). La consanguinidad es un proceso que reduce la heterocigosis y aumenta la homocigosis, lo cual podría conllevar a la aparición de defectos genéticos como infertilidad, disminución del crecimiento o aparición de fenotipos indeseables (Mamani et al., 2016). Es así que los criadores de caballos estarían utilizando pie de crías de distintas genealogías equinas para evitar esta erosión genética, evento que aumentaría la heterocigosidad en las poblaciones. Los bajos valores del índice FST evidencian ausencia de subestructuración en la población total, lo cual conlleva a las poblaciones de caballos de Sahagún a comportarse como una metapoblación.
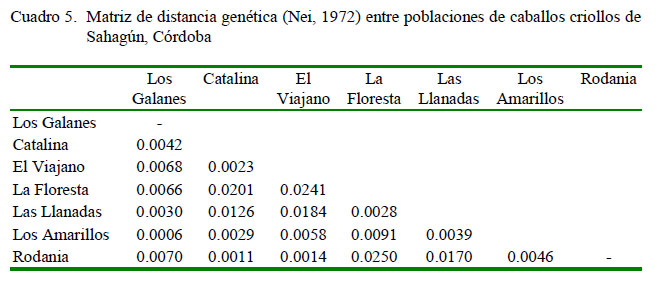
La distancia genética entre las poblaciones fue baja, siendo Los Galanes y Los Amarillos las poblaciones más cercanas (Cuadro 5), mientras que El Viajano y La Floresta, resultaron ser las de mayor diferencia, cifra poco significativa, pues no superó el 5%. En este contexto, la población de La Floresta presentó los mayores valores de distancia genética en comparación con el resto de las poblaciones. Los bajos valores de las distancias genéticas obtenidas entre las poblaciones estudiadas, podría responder a factores como la cercanía geográfica, puesto que se ha reportado que las distancias geográficas son directamente proporcionales a las distancias genéticas (Cruz-Salazar et al., 2014).
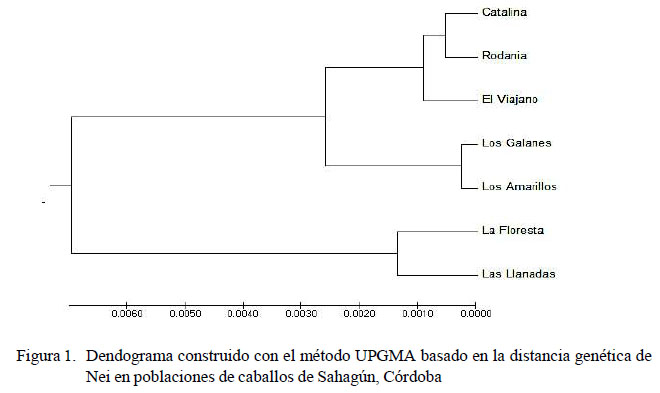
El dendrograma (Figura 1) evidencia la similitud genética entre las poblaciones de Catalina y Rodania, a las cuales se asocia la población de El Viajano, y a estas, Los Galanes y Los Amarillos. Además, se evidencia asociación entre las poblaciones de La Floresta y Las Llanadas, con un valor de distancia poco significativo, que permite deducir que las poblaciones en conjunto están muy relacionadas. Este hecho se podría explicar por el elevado flujo génico, además de que en el proceso de domesticación se han podido fijar algunos genes por preferencias antrópicas (Domínguez-Sánchez et al., 2015), al estar algunos fenotipos asociados a una mayor resistencia a duras jornadas de trabajo en el campo, lo cual los condujo a ser los preferidos por el humano, llegándose a una estrecha relación genética (poca diferenciación) entre las poblaciones de caballos.
CONCLUSIONES
-
El marcador Extension fue el que mostró la mayor frecuencia alélica, posiblemente favorecido por selección artificial.
-
El marcador Tobiano fue el menos frecuente; hecho que estaría relacionado con la selección en contra de individuos que porten este marcador.
-
Los bajos valores de variabilidad genética y el alto grado de flujo génico sugieren que las subpoblaciones se encuentran muy relacionadas genéticamente y se comportan como una metapoblación.
LITERATURA CITADA
1. Bartolomé E, Azor P, Gómez M, Peña F. 2008. La determinación genética del color de la capa en el caballo: bases y aplicación al caballo de la raza Pottoka. Universidad de Córdoba, España. [Internet]. Disponible en: http://pottoka.info/files/galeria/Genetica_color_capa_pottoka.pdf [ Links ]
2. Bellone RR. 2010. Pleiotropic effects of pigmentation genes in horses. Anim Genet 41: 100-110. doi: 10.1111/j.1365- 2052.2010.02116.x [ Links ]
3. Charon KM, Lipka KR. 2015. The effect of a coat colour-associated genes polymorphism on animal health – a review. Ann Anim Sci 15: 3-17. doi: 10.2478/aoas-2014-0066 [ Links ]
4. Correa L, Reyes A, Pardo E, Cavadía M. 2015. Genetic diversity detection of the domestic horse (Equus caballus) by genes associated with coat color. Rev MVZ Cordoba 20: 4779-4789. [ Links ]
5. Cruz-Salazar B, Ruiz-Montoya L, Navarrete-Gutiérrez D, Espinoza- Medinilla E, Vázquez-Domínguez E, Vázquez L. 2014. Diversidad genética y abundancia relativa de Didelphis marsupialis y Didelphis virginiana en Chiapas, México. Rev Mex Biodivers 85: 251-261. doi: 10.7550/rmb.36116 [ Links ]
6. Domínguez-Sánchez CA, Cervantes P, Pérez-Rico A, Delgado JV, Jiménez LM, Aguirre L, et al. 2015. Estructura genética del caballo local de Veracruz, México, usando microsatélites. AICA 6: 192-200. [ Links ]
7. Finn J, Haase B, Willet C, van Rooy D, Chew T, Wade C, Hamilton NA, et al. 2016. The relationship between coat colour phenotype and equine behaviour: a pilot study. Appl Anim Behav Sci 174: 66-69. doi: 10.1016/j.applanim.-2015.11.004 [ Links ]
8. Gastaldo A, Rodenbusch R, Fossati R, Azambuja CJ, Alho C. 2017. Population genetic study over 32,000 equines from Uruguay using seventeen forensically informative STR loci. Forensic Sci Int-Gen 26: e19-e22. doi: 10.1016/j.fsigen.2016.10.011 [ Links ]
9. Giovambattista G, Rogberg A, Ripoli M, Villegas E, Díaz S, Posik D, et al. 2010. La genética molecular de bovinos y equinos criollos en los albores del siglo XXI. BAG J Basic Appl Genet 21: 1-14. [ Links ]
10. Goudet J. 1995. FSTAT (version 1.2): a computer program to calculate Fstatistics. J Hered 86: 485-486. doi: 10.1093/oxfordjournals.jhered.a111627 [ Links ]
11. Haase B, Brooks SA, Schlumbaum A, Azor PJ, Bailey E, Alaeddine F, Mevissen M, et al. 2007. Allelic heterogeneity at the equine KIT locus in dominant white (W) horses. Plos Genet 3: e195. doi: 10.1371/journal.pgen.-0030195 [ Links ]
12. Imsland F, McGowan K, Rubin CJ, Henegar C, Sundström E, Berglund J, Schwochow D, et al. 2016. Regulatory mutations in TBX3 disrupt asymmetric hair pigmentation that underlies Dun camouflage color in horses. Nat Genet 48: 152-158. doi: 10.1038/ng.3475 [ Links ]
13. Kumar S, Stecher G, Tamura K. 2016. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets. Mol Biol Evol 33: 1870-1874. doi: 10.1093/molbev/msw054 [ Links ]
14. Librado P, Fages A, Gaunitz C, Leonardi M, Wagner S, Khan N, Hanghøj K, et al. 2016. The evolutionary origin and genetic makeup of domestic horses. Genetics 204: 423- 434. doi: 10.1534/genetics.116.194860 [ Links ]
15. Mamani-Mayta ME, Loza-Murguia MG, Torres-Tola E, Luna-Barrón R, Arteaga-Voigt D. 2016. Consanguinidad de tres poblaciones de ratas (Rattus novergicus) Sprague-Dawley de bioterio a traves de tres polimorfismos de longitud de secuencias simples (SSLP). J Selva Anim Sci 3: 58-76. [ Links ]
16. Nei M. 1972. Genetic distance between populations. Am Nat 106: 283-292. doi: 10.1086/282771 [ Links ]
17. Reissmann M, Musa L, Zakizadeh S, Ludwig A. 2016. Distribution of coatcolor- associated alleles in the domestic horse population and Przewalski’s horse. J Appl Genet 57: 519-525. doi: 10.1007/s13353-016-0352-7
18. Rieder S. 2009. Molecular tests for coat colours in horses. J Anim Breed Genet 126: 415-24. doi: 10.1111/j.1439-0388.2009.00832.x [ Links ]
19. Stachurska A, Jansen P. 2015. Cryptotobiano horses in Hucul breed. Czech J Anim Sci 60: 1-9. doi: 10.17221/7905-CJAS [ Links ]
20. Stachurska A, Ussing AP. 2007. Coat colour versus performance in the horse (Equus caballus). Pol J Nat Sci 22: 43-49. [ Links ]
21. Yeh F, Yang R, Boyle T. 1999. POPGENE Microsoft windows-based freeware for population genetic analysis. Release 1.31. University of Alberta, Edmonton, Canada. [Internet]. Disponible en: https://sites.ualberta.ca/~fyeh/popgene.pdf [ Links ]
22. Yepes W, Pardo E, Causil LA. 2017. Diversidad genética del caballo criollo (a) mediante genes asociados al pelaje en Valencia, Colombia. Rev Inv Vet Perú 28: 562-570. doi: 10.15381/rivep.v28i3.-13353 [ Links ]
Recibido: 7 de febrero de 2018
Aceptado para publicación: 7 de julio de 2018