Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.29 no.4 Lima oct./dic. 2018
http://dx.doi.org/10.15381/rivep.v29i4.15192
ARTÍCULOS PRIMARIOS
Identificación de bovinos persistentemente infectados y genotipo del virus de la diarrea viral en bovinos de Anta, Cusco, Perú
Identification of persistently infected cattle and genotype of bovine viral diarrhea virus in cattle of Anta, Cusco, Peru
Edgar Valdez G.1,3, Ignasio Pacheco P.1, Walter Vergara A.1, Juan Pinto L.1, Fiorela Fernández B.1, Fiorela Guzmán F.1, Dennis Navarro M.2, Hermelinda Rivera G.2
1 Laboratorio de Desarrollo y Validación de Pruebas Serológicas y Moleculares para la Investigación y Diagnóstico de Enfermedades Infecciosas, Escuela Profesional de Zootecnia, Facultad de Ciencias Agrarias, Universidad Nacional de San Antonio Abad del Cusco, Cusco, Perú
2 Laboratorio de Microbiología y Parasitología Veterinaria, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima, Perú
3 Email: evg1205@hotmail.com
RESUMEN
El objetivo del presente estudio fue identificar bovinos persistentemente infectados (PI) y el genotipo del virus de la diarrea viral (VDVB) en bovinos de pequeños ganaderos de cinco distritos de la provincia de Anta, Cusco, que resultaron negativos a anticuerpos contra el VDVB (n=558) en un estudio previo. La identificación de los animales PI se realizó en las 558 muestras de suero de bovinos Holstein, Brown Swiss y criollos hembras de varias edades, mediante la prueba de ELISA de captura. La identificación del genotipo viral fue realizada en cuatro muestras de bovinos PI mediante la prueba de RT-PCR en tiempo real. El 7.2% (40/558) de los bovinos resultaron positivos a antígeno viral en un primer análisis, donde el antígeno fue detectado en animales mayores a 6 meses hasta los 5 años en las tres razas. En el segundo análisis, realizado 30 días después, en los 40 positivos, 30% (12/40) continuaron siendo positivos a antígeno viral indicando que eran animales PI entre mayores a 6 meses y 4 años de cuatro de los cinco distritos de Anta. La prevalencia de los animales PI en la población bovina muestreada de la provincia de Anta fue de 2.2% (12/558). La prueba RT-PCR en tiempo real mostró la amplificación de un producto específico correspondiente al genotipo 1 (VDVB-1). No hubo amplificación de la secuencia objetivo del genotipo 2.
Palabras clave: bovinos; crianza semi-extensiva; virus de la diarrea viral bovina; antígeno del VDVB; genotipo viral
ABSTRACT
The aim of this study was to identify persistently infected cattle (PI) and to identify the viral diarrhea virus genotype (BVDV) in cattle of small farmers in five districts of the province of Anta, Cusco, which were negative for antibodies against BVDV (n=558) in a previous study. The identification of the PI animals was carried out in the 558 serum samples of female bovine Holstein, Brown Swiss and Creoles of various ages, by the capture ELISA test. The identification of the viral genotype was carried out in four samples of PI cattle through the RT-PCR real-time. The results showed that 7.2% (40/558) of the cattle were positive for viral antigen in a first analysis, where the antigen was detected in animals older than 6 months up to 5 years in the three breeds. In the second analysis carried out 30 day after in the 40 positives, 30% (12/40) continued to be positive for viral antigen indicating that they were PI animals between 6 months and 4 years in four of the five Anta districts. The prevalence of PI animals in the bovine population sampled in province ofAnta was 2.2% (12/558). Real-time RT-PCR showed the amplification of a specific product corresponding to genotype 1 (VDVB-1). There was no amplification of the target sequence of genotype 2.
Key words: bovine; herds; semi-extensive management; bovine virus diarrhea virus; BVDV antigen; genotype
INTRODUCCIÓN
La diarrea viral bovina (DVB) es una enfermedad endémica de la población de rumiantes domésticos y silvestres, pues desde que emergió en 1946 ha sido registrada en bovinos de todos los continentes, excepto en la Antártida (Ridpath, 2010). La enfermedad de DVB es causante de severas pérdidas económicas por afectar el desempeño reproductivo, así como el tracto respiratorio e inmunitario del animal (Grooms, 2004; Workman et al., 2016). La sintomatología clínica de la enfermedad ha variado en el transcurso de las últimas siete décadas, siendo en la actualidad principalmente de tipo aguda caracterizada por una ligera reducción en la producción de leche, ligero incremento de otras enfermedades en el hato, etc., y de tipo subclínica siendo usualmente no percibida para el ganadero (Ridpath, 2010).
Desde su aparición, la DVB ha llamado la atención de la comunidad científica mundial, cuya intensa producción intelectual sobre el tema ha permitido el casi completo conocimiento sobre la biología molecular del agente causal, la patogénesis, mecanismos de evasión de la respuesta inmune del hospedero como estrategias de sobrevivencia, etc., lo cual ha contribuido al desarrollo de herramientas diagnósticas y estrategias de control de la enfermedad (Lanyon et al., 2014; Schweizer y Peterhans, 2014).
El virus de la diarrea viral bovina (VDVB) junto con el virus de la Peste Porcina Clásica (PPC), la Enfermedad de la Frontera (BDV) y otros posteriormente aislados, pertenecen al género Pestivirus de la familia Flaviviridae (Liu et al., 2009; Neill, 2013). Se conocen dos genotipos denominados como VDVB-1 y VDVB-2 y posiblemente el VDVB-3, representado por las cepas HoBi-Like, originalmente aisladas de suero fetal bovino procedente de Brasil (Bauermann et al., 2013). El VDVB, a diferencia del PPC, presenta muchas variantes dentro de cada genotipo, siendo el genotipo-1 el de mayor distribución y con más cepas variantes a nivel mundial (Vilcek et al., 2004; Giammarioli et al., 2015).
Un aspecto importante del VDVB es que cepas cultivadas in vitro pueden ser no citopáticas (NCP), es decir, no lisan a las células durante su replicación, característica de casi todas las cepas de campo; mientras que un pequeño porcentaje de cepas pueden ser citopáticas (CP), que surgen a partir de una cepa NCP por mecanismos moleculares de recombinación a nivel de una proteína no estructural, la proteína N2/3 del virus (Peterhans et al., 2010; Neill, 2013). Las cepas del VDVB de ambos fenotipos, NCP y CP, infectan al bovino y otros rumiantes susceptibles ocasionando la enfermedad aguda o subclínica. Los signos clínicos y lesiones histopatológicas son ampliamente descritos pero es importante recalcar que la infección dependerá entre otros factores de la susceptibilidad del animal, el periodo de gestación, la integridad del sistema inmune, el fenotipo de la cepa infectante, etc. (Brownlie, 1991; Brodersen, 2014).
Si una vaca susceptible, pero no preñada, es infectada por una cepa CP o NCP, desarrollará una infección posiblemente ligera o subclínica, seguido de una viremia con una duración de un par de semanas mientras genera una eficiente respuesta inmunitaria humoral, principalmente por anticuerpos neutralizantes e inmunidad celular que eliminan al virus del organismo. Los niveles de anticuerpos neutralizantes inducidos por la glicoproteína E2, presente en la envoltura viral, se elevan hasta alcanzar una meseta de larga duración (Chase et al., 2004). Las consecuencias de la infección en la vaca preñada dependen del periodo de la gestación al momento de la infección. En general, los efectos pueden ser desde una infertilidad temporal, alargamiento del periodo entre partos, pérdida embrionaria, abortos, malformaciones congénitas, nacimiento de terneros persistentemente infectados (PI) o portadores del virus y hasta nacimientos de terneros con anticuerpos contra el VDVB, indicando que hubo una infección prenatal tardía (Schweizer y Peterhans, 2014).
El VDVB como cualquier microorganismo en la naturaleza busca permanecer en la población animal. En este sentido, científicos como Peterhans et al. (2010) han descrito estrategias de como el virus manipula los mecanismos de defensa modificando su virulencia y los mecanismos que desactivan la inmunidad innata, que es fundamental para el inicio de la inmunidad adaptativa (Brackenbury y Charleston, 2003). Es aquí donde se fundamenta la existencia de los animales persistentemente infectados (PI) o portadores del VDVB.
Los animales PI surgen cuando la vaca se infecta con una cepa NCP durante el primer tercio de la gestación (entre 80 a 125 días), que es cuando el sistema inmune fetal es aún inmaduro (Grooms, 2004; Chase, 2013). Este espacio es conocido como la «ventana», pues solo en ese estrecho margen se genera el ternero PI. Esto explica la baja prevalencia de estos animales, que a nivel mundial suele ser de 0.5 a 2% (Houe, 1992). Dado que la prevalencia de los animales PI es baja se requiere del análisis de grandes cantidades de muestras para ser identificados (Brodersen, 2014). Los animales PI no responden con anticuerpos contra el VDVB presente en su organismo, por lo que su identificación se basa en la ausencia de anticuerpos contra el VDVB, pero con presencia del virus en todos sus tejidos, secreciones y excreciones. Es por ello que son eficientes diseminadores del virus dentro del hato, del rebaño y de una zona ganadera (Houe, 1999).
En estudios realizados en bovinos de crianza intensiva de Lima, Arequipa, Tacna y otros lugares del Perú se han detectado entre 0.1 y 4% de animales portadores y hasta 10% dentro de un hato de crianza intensiva de aproximadamente 800 vacas en producción (Chacón et al., 2002; Jayashi et al., 2005, Huamán et al., 2007). No existen referencias sobre la identificación de animales PI en bovinos de crianza extensiva y semi-extensiva de zonas ganaderas altoandinas como la Pampa de Anta (Cusco), y otros lugares del país donde la ganadería es a base de pasturas para la producción de leche y subproductos lácteos en manos de pequeños ganaderos y donde las perspectivas de desarrollo ganadero podrían ser mayores. El objetivo del presente estudio fue identificar a los animales persistentemente infectados y determinar el genotipo de las cepas del VDVB que circulan en bovinos, según raza y edad, de cinco distritos de la provincia de Anta, Cusco, que resultaron seronegativos a DVBV.
MATERIALES Y MÉTODOS
Muestras de Suero
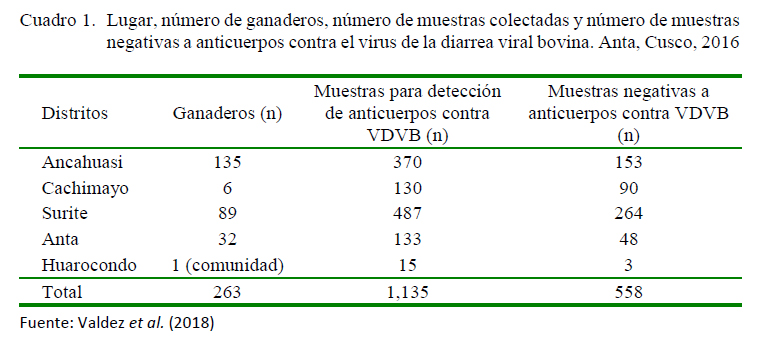
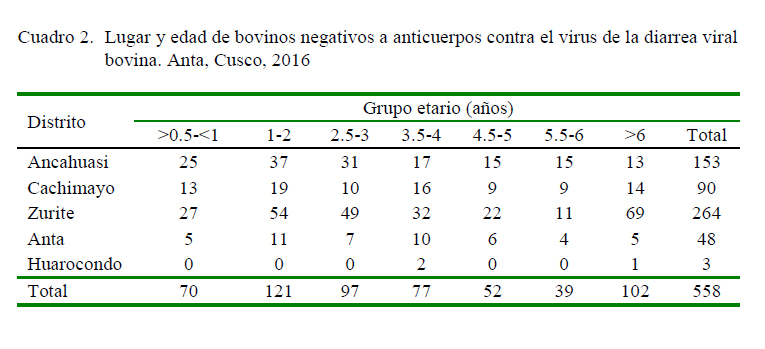
El estudio empleó 558 muestras de suero de bovinos hembras de diversas edades que resultaron seronegativas a anticuerpos contra el VDVB durante un estudio previo de seroprevalencia del VDVB en 1135 muestras de suero bovino de 263 pequeños ganaderos de cinco distritos de la provincia de Anta, Cusco (Cuadros 1 y 2) (ver Valdez et al., 2018). De las 558 muestras, 40 resultaron positivas a antígeno del VDVB. Treinta días después se volvió a colectar muestras de sangre de los 40 animales positivos para confirmar el estatus de animal PI. En el presente estudio no se incluyeron a los toros por problemas logísticos.
Animales Persistentemente Infectados (PI)
La detección de animales PI, seronegativos al VDVB, se hizo mediante el uso de un kit comercial de ELISA de captura (Idexx BVDV Ag Serum Plus Test), siguiendo las instrucciones del fabricante (Cuadro 1). Una muestra fue considerada positiva al antígeno del VDVB cuando el coeficiente M/P fue mayor o igual a 0.15, en el primer y segundo análisis realizado en los mismos animales, y negativo cuando el M/P fue menor a 0.15. El análisis serológico fue desarrollado en el Laboratorio de Desarrollo y Validación de Pruebas Serológicas y Moleculares para la Investigación y Diagnóstico de Enfermedades Infecciosas de la Escuela Profesional de Zootecnia de la Facultad de Ciencias Agrarias de la Universidad Nacional de San Antonio Abad del Cusco, Perú.
Identificación del Genotipo del VDVB en Animales PI
Se escogieron cuatro muestras de animales PI, uno de cada distrito con un coeficiente M/P de 0.3 a 0.8. Las muestras se mantuvieron congeladas a -20 °C y enviadas al Laboratorio de Virología de la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos, Lima, para la identificación del genotipo viral mediante la prueba de RT-PCR en tiempo real.
La extracción de ARN a partir de sueros de bovinos PI (n=4) se hizo empleando el kit comercial MagMAX-96 Viral RNA Isolation (Applied Biosystems, EEUU), el cual utiliza perlas magnéticas como sistema de purificación. Se realizaron cuatro lavados y la elución fue con 90 µl del Elution Buffer provisto por el kit. Los ARN extraídos fueron utilizados como moldes para la detección del VDVB-1 y VDVB-2, empleando dos pares de cebadores, dos sondas y controles del kit BVDV genesig® RT-PCR (Primerdesign, UK); y el master mix del Oasig OneStep qRT-PCR (Primerdesign, UK). La elaboración de la mezcla de reacción y las condiciones para la amplificación de los fragmentos del genoma blanco se hicieron siguiendo las instrucciones del fabricante en el termociclador qTOWER 2.2 Real-Time PCR System (Analytik Jena, Alemania).
RESULTADOS
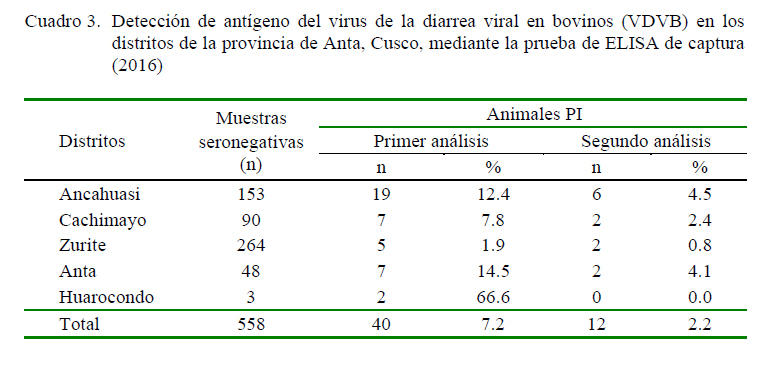
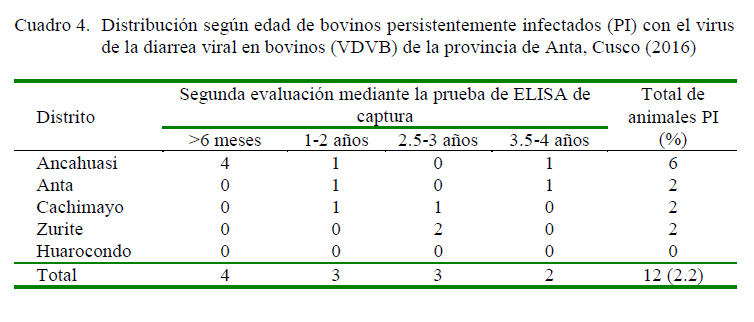
El 7.2% (40/558) de las muestras fueron positivas a antígeno del VDVB en un primer análisis. Las muestras positivas a antígeno del VDVB provinieron de animales de 6 meses hasta los 5 años. Los 40 animales cuyas muestras resultaron positivas al VDVB 30 días después y el segundo análisis realizado en estas muestras determinó que 12 de los 40 (30%) animales continuaron siendo positivos al VDVB y, por tanto, eran animales PI. La prevalencia de animales PI en la población de animales seronegativos muestreados de la provincia de Anta fue de 2.2% (12/558) (Cuadro 3). Los animales eran desde mayores a 6 meses hasta 3.5-4 años (Cuadro 4).
Los 28 bovinos restantes que resultaron negativos en el segundo análisis fueron considerados animales con infección aguda y virémicos.
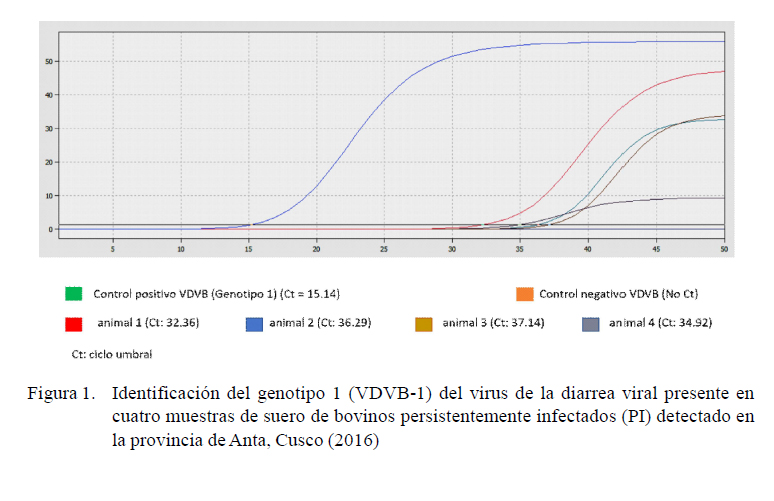
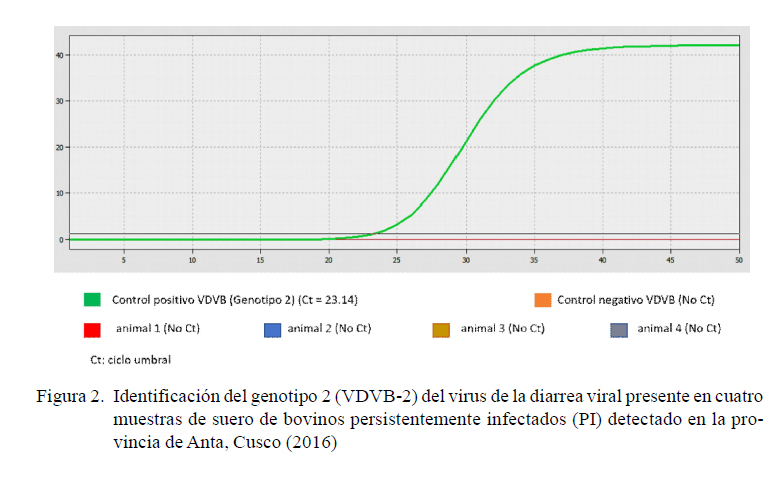
Los resultados de la prueba de RT-PCR en tiempo real de los cuatro sueros (uno de cada distrito) muestra la amplificación de un producto específico del genotipo 1 con valores Ct (Ciclo threshold o ciclo umbral) de 32.36, 36.29, 37.14 y 34.92 para cada muestra frente al control positivo del genotipo 1 con un Ct de 15.14 (Figura 1). No hubo amplificación de productos específicos para el genotipo 2 (Figura 2).
DISCUSIÓN
Los animales PI son importantes reservorios del VDVB y eliminan grandes cantidades de virus mientras viven, por lo que su rol es central en la epidemiología del virus. El 2.2% (12/558) de frecuencia de animales PI con el VDVB en pequeños hatos bovinos donde la frecuencia de anticuerpos contra el VDVB en los animales de la zona es más de 50% (Valdez et al., 2018) es similar a la prevalencia descrita a nivel global cuyo rango varía entre 0.5 y 2%, aunque puede ser mayor en algunos hatos (Houe, 1999; Wittum et al., 2001). Durante el muestreo, el 60% de los animales PI tuvieron apariencia física descritas en la literatura como de menor desarrollo corporal con relación a su edad, pobre condición corporal, etc. (Lanyon et al., 2014). Algunos terneros PI tuvieron erosiones discretas en la nariz que podrían deberse al pasto seco del campo o a lesiones ocasionados por VDVB u otros agentes infecciosos; pero también hubo animales PI de apariencia física normal. Los ganaderos indicaron que algunas de las vacas PI de 2-3 años tuvieron abortos y otros animales tenían largos intervalos entre partos.
La identificación de animales PI en una población bovina de crianza semi-extensiva a una altitud de 3200 msnm, representa el primer estudio que evidencia la presencia de estos animales en una zona altoandina sugiriendo que el VDVB es uno de los agentes infecciosos que estaría afectando la economía de los pequeños ganaderos al ocasionar fallas en el desempeño productivo y reproductivo de los animales. Además, el presente estudio muestra que la infección por el VDVB en la provincia de Anta se encuentra activa, pues 28 de los 40 (70%) animales positivos a antígeno viral en el primer análisis, fueron negativos 30 días después. Esto indica que estos animales estuvieron virémicos, pero que en dicho periodo de tiempo eliminaron al virus de la circulación sanguínea (Cuadro 3). Las causas de infecciones virales activas podrían deberse a varios factores, siendo las más importantes las ferias ganaderas, pasturas comunes, ausencia de bioseguridad, toros reproductores virémicos, etc. Es conocido que en áreas donde el VDVB presenta alta prevalencia existen animales PI quienes se encargan de mantener al virus en los animales en riesgo (Houe, 1999).
En un estudio local realizado en hatos de crianza semi-extensiva de tres comunidades ubicados a 4000 msnm con una seroprevalencia del VDVB de 56.2% no se detectaron animales PI (Cárdenas et al., 2011), ni en otro de crianza mixta semi-extensiva donde anticuerpos contra el VDVB fueron detectados en todas las especies, pero con una frecuencia mayor al 50% en bovinos (Rivera H, 2017, Lima, comunicación personal). Es probable que nazcan animales PI en ganaderías mixtas de las zonas altoandinas, pero posiblemente mueren antes de los seis meses por problemas diarreicos, respiratorios, desnutrición, friaje, etc. Estudios de prevalencia del VDVB han sido realizados en bovinos de crianza intensiva, semi-extensiva, y en ganaderías de crianza mixta de la región andina (Jayashi et al., 2005; Cabello et al., 2006; Quispe et al., 2008; Cárdenas et al., 2011), demostrándose la amplia distribución del VDVB; sin embargo, la identificación de animales PI ha sido detectado mayormente en hatos de crianza intensiva (Jayashi et al., 2005), incluso en hatos vacunados (Chacón et al., 2003) y en pequeños hatos bovinos de crianza semi-extensiva productoras de leche, como en las zonas de Majes, Arequipa (Huamán et al., 2007).
Los animales PI fueron identificados en animales entre más de 6 meses y 4 años (Cuadro 4). En la literatura se menciona que el 50% de los animales PI mueren en el primer año de vida debido a infecciones secundarias del tracto respiratorio o gastrointestinal (Duffel y Harkness, 1985; Taylor et al., 1997). Los ganaderos que contribuyeron con el estudio no usan registros, de allí que las edades registradas podrían estar sobreestimadas, aunque existen reportes de sobrevivencia de animales PI de hasta 4.5 años (Houe, 1992, Voges et al., 2006). Los dos animales PI adultos eran vacas y según Littlejohns y Horner (1990), la sobrevivencia en hembras suele ser mayor que en machos, posiblemente por el mejor cuidado en el manejo de las terneras por ser futuros vientres de reemplazo.
Los animales PI fueron identificados mediante la prueba de ELISA de captura por ser una prueba de menor costo, de alta sensibilidad (94-97%) y especificidad (100%) y porque grandes cantidades de muestras pueden ser procesadas simultáneamente (Brinkhof et al., 1996). En el caso del VDVB y en todos los pestivirus, la prueba detecta la proteína no estructural p80 (NS3), que se genera durante la replicación viral, mediante un anticuerpo monoclonal con alta sensibilidad y especificidad analítica para detectar todas las cepas del VDVB. La p80 es una proteína altamente conservada, inmunogénica y abundante en los animales PI y en animales virémicos (Dubovi, 2013).
El VDVB presente en los animales PI de la provincia de Anta perteneció al genotipo 1 (VDVB-1). Las cuatro muestras amplificaron productos específicos con valores Ct de 32.36, 36.29, 37.14 y 34.92, mientras que el control del genotipo 1 tuvo un Ct de 15.14. Estos valores Ct indican cantidades variables de ARN presente en las muestras de suero. Si el VDVB presente en las muestras hubiesen sido aislados in vitro posiblemente sus valores Ct habrían sido similares al Ct del control positivo, ya que el valor Ct es inversamente proporcional a la cantidad del genoma viral presente en las muestras (Figura 1). No se amplificó producto alguno del genotipo 2, observándose únicamente el valor Ct de 23.14 del control positivo (Figura 2).
El presente resultado confirma hallazgos previos sobre detección del genotipo 1 en aislados del VDVB de animales virémicos y PI de diversos lugares del país (Ståhl et al., 2009; Arainga et al., 2010), indicando que el genotipo 1 es el que está circulando en la población bovina del Perú. Se conoce que el VDVB genotipo 1 es el más común y el que presenta un alto grado de variaciones o subgenotipos identificados como 1a, 1b, 1k, mientras que el genotipo 2 posee dos subgenotipos 2a y 2b (Ridpath, 2010). No se conoce la real significancia clínica de los subgenotipos, pero se ha demostrado que existen diferencias antigénicas frente a paneles de anticuerpos monoclonales que podrían tener implicancias en el diagnóstico de laboratorio. Ridpath (2010) menciona que además de los genotipos y subgenotipos del VDVB y otros pestivirus, es importante mantener una vigilancia a nivel molecular de las cepas existentes a fin de identificar las variantes, pues se han venido aislando e identificando nuevos pestivirus.
CONCLUSIONES
-
Se concluye que los animales persistentemente infectados (PI) con el virus de la diarrea viral bovina están presentes en hatos de crianza semi-extensiva de pequeños ganaderos localizados en las zonas altoandinas de Cusco a 3200 msnm.
-
El 2.2% de animales PI en la provincia de Anta, Cusco fue similar a prevalencias descritas en otras partes del mundo.
-
El VDVB identificado en los animales PI pertenece al genotipo 1 (VDVB-1). El genotipo 2 (VDVB-2) no fue detectado.
Literatura citada
1. Arainga M, Rivera H, Huamán JC, Manchego A. 2010. Fenotipo y genotipo del virus de la diarrhea viral aislado de bovinos en el Perú. Rev Inv Vet Peru 21: 192-203. doi: 10.15381/rivep.v21i2.137 [ Links ]
2. Bauermann FV, Ridpath JE, Weiblen R, Flores EF. 2013. HoBi-like viruses: an emerging group of pestiviruses. J Vet Diagn Inves 25: 6-15. doi: 10.1177/1040638712473103 [ Links ]
3. Brackenbury LS, Charleston CB. 2003. Aspects of the innate and adaptative immune responses to acute infections with BVDV. Vet Microbiol 96: 337-344. doi: 10.1016/j.vetmic.2003.09.004 [ Links ]
4. Brinkhof J, Zimmer G, Westenbrik F. 1996. Comparative study on four enzyme-linked immunosorbent assay and cocultivation assay for the detection of antigens associated with the bovine viral diarrhea virus in persistently infected cattle. Vet Microbiol 50: 1-6. doi: 10.1016/0378-1135(95)00201-4 [ Links ]
5. Brodersen BW. 2014. Bovine viral diarrhea virus infections: manifestations of infection and recent advances in understanding pathogenesis and control. Vet Pathol 51: 453-464. doi: 10.1177/0300985813520250 [ Links ]
6. Brownlie J. 1991. The pathways for bovine viral diarrhea virus biotypes in the pathogenesis of disease. Arch Virol 3: 97-96 [ Links ]
7. Cabello K, Quispe R, Rivera H. 2006. Frecuencia de los virus parainfluenza 3, Respiratorio sincitial y diarrea viral bovina en un rebaño mixto de una comunidad campesina de Cusco. Rev Inv Vet Perú 17: 167-172. doi: 10.15381/rivep.v17i2.1535 [ Links ]
8. Cárdenas C, Rivera H, Arainga M, Ramírez M, De Paz J. 2011. Prevalencia del virus de la diarrea viral bovina y de animales portadores del virus en bovinos de la provincia de Espinar, Cusco. Rev Inv Vet Perú 22: 261-267. doi: 10.15381/rivep.v22i3.268 [ Links ]
9. Chacón J, Benito A, Rivera H. 2003. Detección de animales portadores del virus de la diarrea viral bovina en un establo vacunado y en otro sin vacunar del valle de Lima. Rev Acad Peru Cienc Vet 3: 14-23. [ Links ]
10. Chase CL. 2013. The impact of bovine viral diarrea virus infection on adaptative immunity. Biologicals 41: 52-60. doi: 10.1016/j.biologicals.2012.09.009 [ Links ]
11. Chase CL, Elmovalid G, Yousif AA. 2004. The immune response to bovine viral diarrhea virus: a constantly changing picture. Vet Clin Noth Am Food Anim Pract 20: 95-114. doi: 10.1016/j.cvfa.2003.11.004 [ Links ]
12. Dubovi EJ. 2013. Laboratory diagnosis of bovine viral diarrhea virus. Biologicals 41: 8-13. doi: 10.1016/j.biologicals.2012.06.004 [ Links ]
13. Duffel SJ, Harkness JW. 1985. Bovine virus diarrhea-mucosal disease infection in cattle. Vet Rec 117: 240-245. [ Links ]
14. Giammarioli M, Ceglie L, Rossi E, Bazzuchi M, Casiari C, Pitrini S, Mario De Mia G. 2015. Increased genetic diversity of BVDV-1: recent findings and implications thereof. Virus Genes 50: 147-151. doi: 10.1007/s11262-014-1132-2 [ Links ]
15. Grooms DL. 2004. Reproductive consequenses of infection with bovine viral diarrhea virus. Vet Clin Food Anim 20: 5-19. doi: 10.1016/j.cvfa.2003.11.006 [ Links ]
16. Houe H. 1992. Age distribution of animal persistently infected with bovine viral diarrhea virus in twenty-two Danish dairy herds. Can J Vet Res 56: 194-198. [ Links ]
17. Houe H. 1999. Epidemiological features and economical importance of bovine virus diarrhea virus (BVDV) infections. Vet Microbiol 64: 89-107. doi: 10.1016/S0378-1135(98)00262-4 [ Links ]
18. Huamán C, Rivera H, Arainga M, Gavidia C, Manchego A. 2007. Diarrea viral bovina y animales portadores del virus en hatos productores de leche de la irrigación de majes, Arequipa. Rev Inv Vet Perú 18: 141-149. doi: 10.15381/rivep.v18i2.1290 [ Links ]
19. Jayashi C, Gavidia C, Araínga M, Manchego A, Rivera H. 2005. Dinámica de seroconversión en hembras bovinas post eliminación de animales portadores del virus de la diarrea viral bovina. Rev Inv Vet Perú 16: 56-64. doi: 10.15381/rivep.v16i1.1536 [ Links ]
20. Lanyon SR, Hill FI, Reichel MP, Brownlie J. 2014. Bovine viral diarrhea: pathogenesis and diagnosis. Vet J 199: 201-209. doi: 10.1016/j.tvjl.2013.07.024 [ Links ]
21. Littlejohns IR, Horner GW. 1990. Incidence, epidemiology and control of bovine pestivirus infections and disease in Australia and New Zealand. Rev Sci Tech Off Int Epiz 9: 195-205. doi: 10.20506/rst.9.1.480 [ Links ]
22. Liu L, Xia H, Wahlberg N, Belak S, Baujle C. 2009, Phylogeny, classification and evolutionary insights into pestiviruses. Virology 385: 351-357. doi: 10.1016/jvirol.2008.12.004 [ Links ]
23. Neill JD. 2013. Molecular biology of bovine viral diarrhea virus. Biological 41: 2-7. doi: 10.1016/j.biologicals.2012.07.002 [ Links ]
24. Peterhans E, bachofen CL, Stalder H, Schweizer M. 2010. Cytopathic bovine viral diarrhea viruses (BVDV): emerging pestivirus doomed to extinction. Vet Res 41: 44. doi: 10.1051/vetres/2010016 [ Links ]
25. Quispe R, Ccama A, Rivera H, Arainga M. 2008. El virus de la diarrea viral en bovinos criollos de la provincia de Melgar, Puno. Rev Inv Vet Perú 19(2): 176-182. doi: 10.15381/rivep.v19i2.1165 [ Links ]
26. Ridpath J. 2010. Bovine viral diarrhea virus: global Status. Vet Clin N Am-Food A 26: 105-121. doi: 10.1016/j.cvfa.2009.10.007 [ Links ]
27. Schweizer M, Peterhans E: 2014. Pestivirus. Annu RevAnim Biosci 2: 141- 163. doi: 10.114/annurev-animal-022.513-114209. [ Links ]
28. Ståhl K, Benito A, Felmer R, Zuñiga J, Reinhardt G, Rivera H, Baule C, Moreno-Lopez J. 2009. Genetic diversity of bovine diarrhea virus (VDVB) from Peru and Chile. Pesq Vet Bras 29: 41-44. doi: 10.1590/S0100736X2009000100006 [ Links ]
29. Taylor LF, Jansen ED, Ellis JA, van den Hurk JV, Ward P. 1997. Performance, survival, necropsy and virological findings from calves persistently infected with bovine viral diarrhea virus originating from a single Saskatchewan beef herd. Can Vet J 38: 29-37. [ Links ]
30. Valdez E, Pacheco I, Vergara W, Pinto J, Fernández F, Guzmán F, Rivera H. 2018. Detección de anticuerpos contra el virus de la diarrea viral en bovinos de la provincia de Anta, Cusco, Perú. Rev Inv Vet Perú 29(4): xxx-xxxx. [ Links ]
31. Vilcek S, Durkovic B, Kolesarova M, Greiser-Wilke I, Paton D. 2004. Genetic diversity of bovine viral diarrhea virus (BVDV) isolates: identification of a new BVDV-1 genetic group. Vet Res 35: 609-615. doi: 10.1051/vetres:2004036 [ Links ]
32. Voges H, Young S, Nash M. 2006. Direct adverse effects of persistently BVDv infection in dairy heifers: a retrospective case control study. Vetscript 19: 22-25. [ Links ]
33. Wittum TE, Grotelueschen DM, Brock KV, Kvasnicka WG, Floyd JG, Kelling CL, Odde KG. 2001. Persistent bovine viral diarrhea virus infection in US beef herds. Prev Vet Med 49: 83-94. doi: 10.1016/S0167-5877(01)00181-7 [ Links ]
34. Workman AM, Heaton MP, Harhay GP, Smith TP, Grotelueschen DM, Sjeklocha D, Brodersen B, et al. 2016. Resolving bovine viral diarrhea virus subtypes from persistently infected U.S. beef calves with complete genome sequence. J Vet Diagn Invest 28: 519528. doi: 10.1177/1040638716654943 [ Links ]
Recibido: 17 de febrero de 2018
Aceptado para publicación: 23 de julio de 2018