Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.30 no.1 Lima ene./mar. 2019
http://dx.doi.org/10.15381/rivep.v30i1.15698
ARTÍCULOS PRIMARIOS
Salmonella spp en aves silvestres que habitan alrededor de una granja de cuyes tecnificada en el distrito de Manchay, Lima
Salmonella spp in wild birds living around a well-managed guinea pig farm in the district of Manchay, Lima
Kathya Espinoza Ramírez1, Siever Morales-Cauti1,2,3
1 Laboratorio de Microbiología, Facultad de Medicina Veterinaria y Zootecnia, Universidad Científica del Sur, Lima, Perú
2 Laboratorio de Microbiología y Parasitología Veterinaria, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima Perú
3 Email: sieverm@hotmail.com
RESUMEN
El presente estudio tuvo como objetivo determinar la presencia de Salmonella spp en aves silvestres que habitan en los alrededores de una granja de cuyes de crianza tecnificada. Se capturaron 185 aves silvestres de vida libre y se identificaron ocho especies: Passer domesticus (63.8%), Volatinia jacarina (16.8%), Troglodytes aedon (2.2%), Pyrocephalus rubinus (1.1%), Molothrus bonariensis (0.5%), Crotophaga sulcirostris (1.1%), Columbina cruziana (12.9%) y Forpus coelestis (1.6%). Las muestras se tomaron a través de hisopado cloacal. Se aisló Salmonella spp en 4.32% (8/185) de las aves silvestres, pero solo en Passer domesticus, Volatinia jacarina y Forpus coelestis. Así mismo, se usaron dos métodos de enriquecimiento, obteniéndose 2.2% (4/185) de muestras positivas a Salmonella spp con el enriquecimiento estándar y 3.8% (7/185) con el enriquecimiento tardío.
Palabras clave: Salmonella spp; aves silvestres; crianza tecnificada; salud pública
ABSTRACT
The objective of the present study was to determine the presence of Salmonella spp in wild birds that live in the surroundings of a well-managed guinea pig farm. Free-living wild birds were captured (n=185) and eight species were identified: Passer domesticus (63.8%), Volatinia jacarina (16.8%), Troglodytes aedon (2.2%), Pyrocephalus rubinus (1.1%), Molothrus bonariensis (0.5%), Crotophaga sulcirostris (1.1%), Columbina cruziana (12.9%) and Forpus coelestis (1.6%). Samples were taken through cloacal swab. Salmonella spp was isolated in 4.32% (8/185) of the wild birds, but only in Passer domesticus, Volatinia jacarina and Forpus coelestis. Likewise, two enrichment methods were used, obtaining 2.2% (4/185) of samples positive for Salmonella spp with standard enrichment and 3.8% (7/185) with delayed enrichment.
Key words: Salmonella spp; wild birds; breeding tech; public health
INTRODUCCIÓN
Las aves silvestres de vida libre son importantes dentro de su ecosistema y generalmente son consideradas como indicadores biológicos de diversos entornos saludables; sin embargo, desde el punto de vista de salud pública esta visión positiva no siempre es válida, porque pueden servir de reservorio de algunos enteropatógenos zoonóticos como Salmonella spp (Albureesh et al., 2007; Braconaro, 2012; Andrés -Barranco et al., 2014).
Salmonella es un bacilo gramnegativo de la familia Enterobacteriaceae y se divide en dos especies: S. bongori y S. enterica, donde se han podido identificar hasta 2500 serotipos (Figueroa y Verdugo, 2005). La mayoría de los serotipos tienen importancia en salud pública y salud animal a nivel mundial, por la infección denominada salmonelosis producida por S. enterica. Su principal reservorio es el intestino de animales homeotermos y poiquilotermos, actuando como diseminadores intermitentes a través de sus heces por largos periodos de tiempo (Uribe y Suarez, 2006; Gopinath et al., 2012; Andrés et al., 2013).
La microbiota gastrointestinal refleja la coevolución de los microorganismos con el animal hospedero y tiene un rol fundamental en la salud animal, respuesta inmune y enfermedad, los cuales también son influenciadas por factores como tipo de alimentación, estación del año o patrones de migración (Benskin et al., 2009; Van Dongen et al., 2013). El tipo de alimentación en aves silvestres es una clave determinante para la adquisición de enterobacterias. Algunas aves silvestres de comportamiento gregario y de alimentación oportunista aprovechan la oportunidad de obtener alimentos y agua en granjas comerciales donde encuentran un entorno favorable (Benskin et al., 2009; Andrés et al., 2013)
El objetivo del presente estudio fue determinar la presencia de Salmonella spp en aves silvestres de vida libre que habitaban alrededor de una granja de cuyes de crianza tecnificada como contribución al conocimiento de la microbiología y al rol de aves silvestres desde el punto de vista epidemiológico en granjas, siendo imperativo para el establecimiento de medidas de control y prevención de salmonelosis.
MATERIALES Y MÉTODOS
Lugar de estudio
El trabajo se llevó a cabo entre febrero y marzo de 2015 en una granja tecnificada de cuyes situada en el distrito de Manchay, Lima, Perú. El análisis de las muestras se realizó en el Laboratorio de Microbiología de la Universidad Científica del Sur, ubicado en el distrito de Villa El Salvador, Lima.
Población Muestral y Muestras
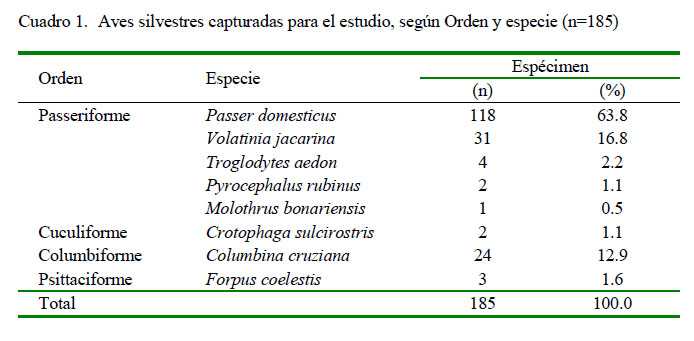
Se capturaron en forma aleatoria muestras de 185 aves de especies que pertenecen a los órdenes Passeriformes, Cuculiformes, Columbiformes y Psittaciformes. La captura se hizo con redes de niebla de 12 x 2.5 m ubicadas en varios puntos alrededor de la granja (Cuadro 1). Las aves fueron luego sujetadas mediante inmovilización manual (Ralph et al., 1996), e introducidas en bolsas de tela individuales para transportarlas al área donde se tomaron las muestras. Para tal efecto, la especie a la que pertenece cada ave fue identificada según lo describe Schulenberg et al. (2010), tomando en cuenta la descripción de sus características generales y mapas de distribución geográfica de cada grupo de especies de aves que han sido registradas en Perú.
La toma de las muestras se realizó a través de hisopados cloacales, utilizando hisopos estériles que fueron suavemente rotados durante 20 segundos con la finalidad de establecer un mayor contacto con la mucosa del tracto intestinal. Luego de la toma de la muestra, se marcaron las aves cortando la punta de una de las plumas axilares y se les liberó.
La muestra con el hisopo fue introducida en tubos Falcon con medio de transporte bacteriano Stuart (Merck) y transportada al laboratorio en cajas térmicas con gel refrigerante a 4 °C.
Aislamiento de Salmonella spp
Enriquecimiento selectivo
Se realizó un enriquecimiento temprano con caldo Rappaport-Vassiliadis, donde se inoculó el hisopo y se incubó a 41 °C por 24 h. Luego se realizó un enriquecimiento tardío y se dejó incubar por 5 días a temperatura ambiente.
Siembra en placas
Se re-suspendieron los caldos de enriquecimiento Rappaport-Vassiliadis tomándose una muestra con un ansa en aro para realizar el sembrado por agotamiento en un medio sólido específico comoAgar Xilosa Lisina Desoxicolato (XLD) y se incubaron a 37 °C por 24 h. Luego de la incubación, los cultivos se clasificaron en colonias negativas y sospechosas a Salmonella spp, considerando que estas colonias presentaban centro negro debido a la producción de sulfhídrilo. Una vez elegida la colonia sospechosa se procedió a realizar la identificación por pruebas bioquímicas.
Identificación Bioquímica
Para la identificación bioquímica de las colonias sospechosas se usaron las pruebas de catalasa, oxidasa, triple azúcar hierro (TSI), citrato Simmons, lisina hierro (LIA), sulfuro indol motilidad (SIM) y caldo urea. Los cultivos fueron llevados a incubación a 37 °C por 24 h, siguiendo los procedimientos utilizados en el formato de instrucciones de la técnica de identificación bioquímica bacteriana del Instituto Nacional de Salud (Caffer y Terragno, 2008).
RESULTADOS
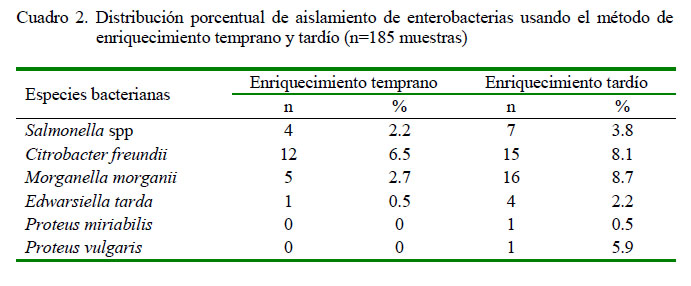
El enriquecimiento temprano permitió aislar Salmonella spp en el 2.2% (4/185) de las muestras y con el enriquecimiento tardío se logró aislar en el 3.8% (7/185) de las muestras. No era un objetivo propuesto para este trabajo, pero el tipo de enriquecimiento (Rappaport-Vassiliadis) y el agar XLD permitieron la identificación de otros géneros de enterobacterias (Cuadro 2.)
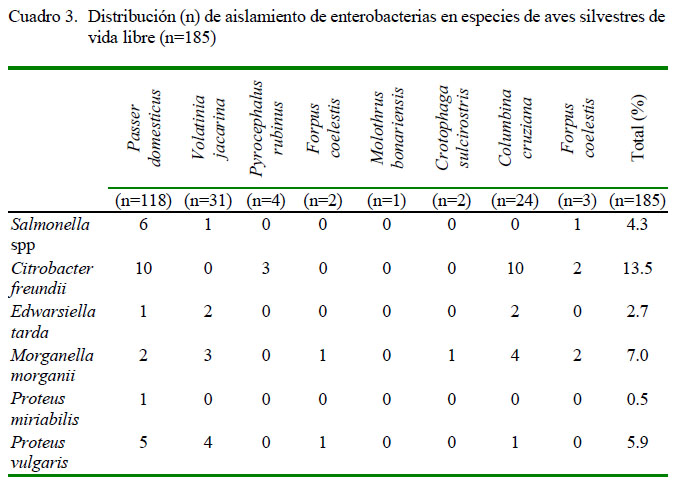
De las 185 muestras procesadas y de acuerdo con las características bioquímicas, 4.3% (8/185) correspondieron a cepas positivas a Salmonella spp, donde se aisló la bacteria en estudio 5.1% (6/118) de Passer domesticus, 3.2% (1/31) de Volatinia jacarina y 1/3 de Forpus coelestis (Cuadro 3).
DISCUSIÓN
El 4.3% (8/185) de las muestras de hisopado cloacal en aves de vida libre capturadas en los alrededores de una granja de cuyes fueron positivas a Salmonella spp mediante identificación bioquímica. Diversos estudios epidemiológicos reportan frecuencias entre 0.04% y más de 20% de Salmonella spp en aves silvestres, variación que depende de ciertos factores que faciliten la transmisión entre aves, ya sea por hacinamiento en estaciones de alimentación, contacto con granjas que tienen una mala bioseguridad, etc. (Skov et al., 2008; Benskin et al., 2009; Andrés-Barranco et al., 2014).
La diseminación de estos microorganismos patógenos depende de varios factores bióticos y abióticos que pueden afectar la supervivencia de los microor-ganismos en un determinado ecosistema, como la relación de sinantropía de algunas aves de comportamiento gregario y alimentación oportunista, lo cual genera un comensalismo obligatorio, como es el caso de Passer domesticus (Koepcke, 1952; Marzluff et al., 2001). Así, la mayoría de las aves capturadas en este estudio fue de esta especie, y los resultados indican que presenta el mayor riesgo de infección, ya que normalmente viven en grupos en los que puede transmitirse Salmonella, vía oro-fecal (Mirzaie et al., 2010). Así mismo, Stewart y Birch (2000) reportaron en esta especie la transmisión por vía cloacal de algunos enteropatógenos, incluyendo Salmonella spp.
El aislamiento de Salmonella spp se da principalmente por técnicas microbiológicas, el cual consiste en el uso de un preenriquecimiento y medios selectivos; sin embargo, en este estudio no se realizó el preenrquecimiento, ya que favorecería el sobrecrecimiento de la flora acompañante e inhibiría el crecimiento de Salmonella spp. Por ello, se optó por usar un caldo de enriquecimiento selectivo (Rappaport-Vassiliadis) y la técnica de un enriquecimiento estándar y otro tardío, buscando beneficiar e incrementar la tasa de recuperación de esta bacteria, toda vez que el aislamiento de Salmonella spp en aves silvestres es baja (Nietfeld et al., 1998; Rybolt et al., 2004; OIE, 2017).
La técnica de enriquecimiento tardío generalmente se usa en granjas avícolas.Waltman et al. (1991) usaron el enriquecimiento tardío, logrando aislar Salmonella spp en el 64% de aves de corral. Nietfeld et al. (1998) demostraron que el enriquecimiento tardío con caldo Rappaport-Vassiliadis era superior a otros caldos de enriquecimiento selectivos para Salmonella a partir de hisopados rectales en cerdos. Así mismo, este método ha sido usado para el aislamiento de Salmonella en aves de vida libre (Khidhir y Aref, 2010) a partir de hisopados cloacales, así como en tortugas Trachemys sp (Meza, 2015).
En este estudio se logró identificar enterobacterias diferentes a Salmonella, ya que el tipo de enriquecimiento (Rappaport-Vassiliadis) y el agar XLD permitió el aislamiento de especies de otros géneros presentes en las muestras trabajadas, tales como Citrobacter freundii (13.5%), Morganella morganii (7.03%), Edwarsiella tarda (2.7%), Proteus miriabilis (0.5%) y Proteus vulgaris (5.9%) (Cuadro 3). Por otro lado, Caffer y Terragno (2008) mencionan que algunas cepas de Salmonella se pueden confundir con otras enterobacterias como C. freundii, P. miriabilis, P. vulgaris, E. tarda y M. morganii debido a que poseen características bioquímicas similares (producen sulfhídrilo) y también se pueden encontrar en la flora bacteriana normal, pues son consideradas como patógenos oportunistas. Por este motivo, se incluyó un diagnóstico diferencial, ya que al sembrarlas en el agar XLD crecen como colonias sospechosas de color negro.
Finalmente, dado que la prevalencia de Salmonella en aves de vida libre presenta una gran variación entre estudios, no es posible determinar si la variación es el resultado de diferentes métodos de muestreos y diseño de estudio aplicativo o si refleja una diferencia significativa en la prevalencia de la enfermedad entre diferentes localidades y países.
Agradecimientos
Los autores agradecen a la Blga. Letty Salinas Sánchez y a Jacqueline Hernández, del Departamento de Ornitología del Museo de Historia Natural de la Universidad Nacional Mayor de San Marcos por el entrenamiento para la captura y reconocimiento de aves, así como para la recolección de muestras.
LITERATURA CITADA
1. Abulreesh HH, Goulder R, Scott GW. 2007. Wild birds and human pathogens in the context of ringing and migration. Ringing & Migration 23: 193-200. doi: 10.1080/03078698.2007.9674363 [ Links ]
2. Andrés-Barranco S, Vico JP, Garrido V, Samper S, Herrera-León S, de Frutos C, Mainar-Jaime RC. 2014. Role of wild bird and rodents in the epidemiology of subclinical salmonellosis in finishing pigs. Foodborne Pathog Dis 11: 689-697. doi: 10.1089/fpd.2014.1755 [ Links ]
3. Andrés S, Vico JP, Garrido V, Grilló MJ, Samper S, Gavín P, Herrera-León S, et al. 2013. Epidemiology of subclinical salmonellosis in wild birds from an area of high prevalence of pig salmonellosis: phenotypic and genetic profiles of Salmonella isolates. Zoonoses Public Hlth 60: 355-365. doi: 10.1111/ j.1863-2378.2012.01542.x [ Links ]
4. Benskin CM, Wilson K, Jones K, Hartley IR. 2009. Bacterial pathogens in wild birds: a review of the frequency and effects of infection. Biol Rev Camb Philos 84: 349-373. doi: 10.1111/j.1469- 185X.2008.00076.x [ Links ]
5. Braconaro P. 2012. Caracterização das microbiotas bacteriana e fúngica presentes em cloacas de passeriformes silvestres confiscados do tráfico que serão submetidos a programas de soltura. Tese de Mestrado. Brasil: Univ. de São Paulo. 72 p. [ Links ]
6. Caffer M, Terragno R. 2008. Manual de procedimientos para la caracterización de Salmonella. Instituto Nacional de Enfermedades Infecciosas. Buenos Aires, Argentina: Instituto Nacional de Enfermedades Infecciosas. 37 p. [ Links ]
7. Figueroa IM, Verdugo RA. 2005. Mecanismos moleculares de patogenicidad de Salmonella spp. Rev Latinoam Microbiol 47: 25-42. [ Links ]
8. Gopinath S, Carden S, Monak D. 2012. Shedding light on Salmonella carriers. Trends Microbiol 20: 320-327. doi: 10.1016/j.tim.2012.04.004 [ Links ]
9. Khidhir Z, Aref E. 2010. Isolation of Salmonella from some species of wild birds in Sulaimania. Al- Mustansiriya J Sci 21: 49-51. [ Links ]
10. Koepcke M. 1952. El gorrión europeo en el Perú. Mar del Sur 22: 63-72. [ Links ]
11. Marzluff J, Bowman R, Donnelly R. 2001. Avian ecology and conservation in an urbanizing world. Boston: Kluwer Academic Publ. 585 p. [ Links ]
12. Meza D. 2015. Identificación, serotipificación y determinación del perfil de sensibilidad de Salmonella spp aisladas de cloacas de tortugas de orejas rojas (Trachemys sp) en cautiverio. Tesis de Médico Veterinario. Lima, Perú: Univ. Científica del Sur. 33 p. [ Links ]
13. Mirzaie S, Hassanzadeh M, Ashrafi I. 2010. Identification and characterization of Salmonella isolates from captured house sparrow. Turk J Vet Anim Sci 34: 181-186. doi: 10.3906/vet-0810-43 [ Links ]
14. Nietfeld JC, Kelly B, Dritz SS, Feder I, Galland JC. 1998. Comparison of conventional and delayed secondary enrichment for isolation of Salmonella spp from swine samples. J Vet Diagn Invest 10: 285-287. [ Links ]
15. [OIE] Organización Mundial de Sanidad Animal. 2017. Salmonelosis. En: Manual de las pruebas de diagnóstico y de las vacunas para los animales terrestres 2017. [Internet]. Disponible en: http://www.- o i e . i n t / f i l e a d m i n / H o m e / e s p / Heal th_standards/ta hm/2.09.08_- SALMONELLOSIS.pdf [ Links ]
16. Ralph J, Geupel G, Pyle P, Tomas M, DeSante D, Mila B. 1996. Manual de métodos de campo para el monitoreo de aves terrestres. USA: USDA. 46 p. [ Links ]
17. Rybolt ML, Wills RW, Byrd JA, Doler TP, Bailey RH. 2004. Comparison of four Salmonella isolation techniques in four different inoculated matrices. Poultry Sci 83: 1112-1116. doi: 10.1093/ ps/83.7.1112 [ Links ]
18. Schulenberg T, Stotz D, Lane D, O´Neill J, Parker T. 2010. Aves del Perú. Lima, Perú: Ed. Corbidi. 660 p. [ Links ]
19. Skov MN, Madsen JJ, Rahbek C, Lodal J, Jespersen JB, Jørgensen JC, Dietz HH, et al. 2008. Transmission of Salmonella between wildlife and meatproduction animals in Denmark. J Appl Microbiol 105: 1558-1568. doi: 10.1111/ j.1365-2672.2008.03914.x
20. Stewart R, Birch T, 2000. Cloacal microbes in house sparrow. Condor 102: 679-684. doi: 10.2307/1369801 [ Links ]
21. Uribe C, Suarez MC. 2006. Salmonelosis no tifoidea y su transmisión de alimentos de origen aviar. Colomb Medica 37: 151-158. [ Links ]
22. van Dongen WF, White J, Brandl HB, Moodley Y, Merkling T, Leclaire S, Blanchard P, et al. 2013. Age-related differences in the cloacal microbiota of wild bird species. BMC Ecol 13:11. doi: 10.1186/1472-6785-13-11 [ Links ]
23. Waltman WD, Horne AM, Pirkle C, Dickson TG. 1991. Use of delayed secondary enrichment for the isolation of Salmonella in poultry and poultry environments. Avian Dis 35: 88-92. doi: 10.2307/1591299 [ Links ]
Recibido: 25 de abril de 2018
Aceptado para publicación: 1 de octubre de 2018