Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.30 no.1 Lima ene./mar. 2019
http://dx.doi.org/10.15381/rivep.v30i1.15670
ARTÍCULOS PRIMARIOS
Detección fenotípica de mecanismos de resistencia antimicrobiana de Escherichia coli aisladas de infecciones entéricas de porcinos provenientes de granjas de producción tecnificada
Phenotypic detection of antimicrobial resistance mechanisms of Escherichia coli isolates from enteric infections in pigs from technified farms
Michelle Monterroso C.1,3, Guillermo Salvatierra R.2, André Sedano S.1, Sonia Calle E.1,4
1 Laboratorio de Microbiología y Parasitología Veterinaria, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima, Perú
2 Universidad San Ignacio de Loyola, Lima, Perú
3 E-mail: m.monterroso.c@gmail.com
4 E-mail: scallee@unmsm.edu.pe
RESUMEN
El objetivo del estudio fue detectar fenotípicamente los mecanismos de resistencia antimicrobiana de 36 aislados de Escherichia coli a betalactámicos, quinolonas y aminoglucósidos mediante la técnica de Kirby-Bauer. Se utilizaron 36 aislados de E. coli procedentes de porcinos de granjas tecnificadas, obtenidos durante el periodo 20102015. Se utilizaron 15 antimicrobianos de importancia en medicina humana y veterinaria. Se detectó resistencia principalmente al ácido nalidíxico (89%, 32/36), cloxacilina (83%, 30/36) y amoxicilina-ácido clavulánico (69%, 25/36). Solo un 3% (1/36) presentó AmpC inducible, 42% (15/36) evidenció una posible mutación en gyrA y el 14% (5/36) al menos dos posibles mutaciones en gyrAo gyrA+parC. Además, el 33% (12/36) evidenció altas probabilidades de presencia de genes qnr. Las enzimas del mecanismo de resistencia a aminoglucósidos fueron positivas en un 39% (14/36) deAAC (6’), 28% (10/36)ANT (2") y 11% (4/36) deAAC (3) IV.
Palabras clave: Escherichia coli; porcino; mecanismos de resistencia; resistencia antimicrobiana; salud pública
ABSTRACT
The aim of this study was to detect the antimicrobial resistance mechanisms of 36 Escherichia coli isolates to beta-lactams, quinolones and aminoglycosides using the Kirby-Bauer technique. Thirty-six E. coli isolates from pigs of technified production farms obtained during the 2010-2015 period were used. Fifteen antimicrobials of importance in human and veterinary medicine were tested. The isolates showed resistance mainly to nalidixic acid (89%, 32/36), cloxacillin (83%, 30/36) and amoxicillin-clavulanic acid (69%, 25/36). Only 3% (1/36) wereAmpC producers, 42% (15/36) showed a possible mutation in gyrA and 14% (5/36) at least two possible mutations in gyrA or gyrA+parC. In addition, 33% (12/36) showed high probabilities of presence of qnr genes. The enzymes associated toaminoglycosideresistance mechanismwere positivein 39%(14/36) toAAC(6’),28% (10/36) toANT (2") and 11% (4/36) toAAC (3) IV.
Key words: Escherichia coli; pigs; resistance mechanisms; antimicrobial resistance; public health
INTRODUCCIÓN
La aparición de cepas resistentes se ha convertido en un problema de índole mundial, tanto en medicina veterinaria como en el tratamiento de infecciones en humanos (Prescott, 2000; Stine et al., 2007). Dada la importancia del problema se han puesto en marcha programas de vigilancia y monitoreo de la resistencia antimicrobiana (OIE, 2016).
Para medir la susceptibilidad in vitro de los microorganismos patógenos a antimicrobianos se utiliza la técnica de Kirby-Bauer (Cona, 2002). Esta técnica permite detectar â-lactamasas de espectro extendido (BLEE), â-lactamasas AmpC, resistencia a quinolonas por mutaciones en los genes de la DNA girasa, topoisomerasa IV o mediadas por plásmidos, así como los patrones de resistencia a aminoglucósidos debidos a la expresión de enzimas modificadoras (Martínez y Calvo, 2010).
Por esas razones, la Organización Mundial de la Salud (OMS) y la Organización Mundial de SanidadAnimal (OIE) recomiendan el monitoreo de microorganismos multidrogoresistentes entre los que destaca Escherichia coli (OMS, 2001; OIE, 2016). La prevalencia de este patógeno resistente es un buen indicador de la presión selectiva por el uso indebido de antimicrobianos y de problemas de resistencia ligado a bacterias patógenas (Van den Bogaard et al., 2000). Las cepas de E. coli resistentes a antimicrobianos pueden constituir un reservorio de genes transferibles a otras poblaciones bacterianas potencialmente patógenas (Lim et al., 2010). Por ejemplo, plásmidos que confieren resistencia a múltiples antimicrobianos pueden transferirse desde E. coli a bacterias como Salmonella Typhimurium u otros patógenos de importancia, no sólo en el ámbito animal, sino además en la salud pública (Kikuvi et al., 2007).
El objetivo del estudio fue detectar fenotípicamente los mecanismos de resistencia a betalactámicos, quinolonas y aminoglucósidos de 36 aislados de E. coli mediante la técnica de Kirby-Bauer siguiendo las recomendaciones del Clinical and Laboratory Standards Institute (CLSI, 2016).
MATERIALES Y MÉTODOS
Material Experimental
Se utilizaron 36 aislados de E. coli procedentes de porcinos, en su mayoría lechones, de granjas tecnificadas obtenidos durante el periodo 2010-2015. Las muestras fueron remitidas al Laboratorio de Microbiología y Parasitología de la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos, Lima. El tipo de muestra recibida fue de órganos derivados de necropsias (hígado, bazo, vesícula biliar, intestinos, pulmón y nódulos linfáticos). La identificación de las bacterias fue llevada a cabo mediante pruebas bioquímicas.
Los aislados fueron criopreservados en viales de 1.5 ml utilizando caldo de cultivo Tripticasa de Soya (TSB) y Glicerol al 87% en una proporción 50/50, y almacenados en el cepario del laboratorio a -20 °C. Los aislados fueron reactivados mediante siembra en agar nutritivo Tripticasa de Soya (TSA) yAgar MacConkey (MC) (Sánchez y Corrales, 2005).
Método Kirby-Bauer
Para la detección de la resistencia se utilizaron 15 antimicrobianos mediante la técnica Kirby-Bauer de difusión en agar. El procedimiento se realizó según recomendaciones del CLSI (2016). Los antimicrobianos utilizados fueron amoxicilina con ácido clavulónico (20/10µg), ceftazidima (30 µg/dl), cefotaxima (30 µg), cefepime (30 µg), aztreonam (30 µg), cefoxitina (30 µg), ceftriaxona (30 µg), cloxacilina (1 µg), gentamicina (10 µg), tobramicina (10 µg), neomicina (30 µg), amikacina (30 µg), kanamicina (30 µg), ácido nalidíxico (30 µg) y ciprofloxacina (5 µg).
Detección de Betalactamasas de Espectro Extendido (BLEE)
Se utilizó el método de Jarlier (Jarlier et al., 1988), colocando un disco de amoxicilinacon ácido clavulánico en el centro de una placa de Petri y discos de ceftazidima, cefotaxima y cefepime rodeando a este a 25 mmde distancia.Adicionalmenteseutilizóel disco de aztreonam. La presencia de BLEE se manifestó por el efecto sinérgico del inhibidor y los discos generando el efecto cola de pez o balón de futbol americano (Jarlier et al., 1988; Famiglietti et al., 2005; Lezameta et al., 2010).
Detección de Betalactamasas Tipo AmpC
Las betalactamasas de la clase molecular C de Ambler hidrolizan cefalosporinas de primera (cefalotina) y segunda generación (cefuroxima), incluidas las cefamicinas (cefoxitina y cefotetán) y, en menor medida, las de tercera generación (cefotaxima, ceftriaxona, ceftazidima) (Navarro et al., 2011). No existen métodos fenotípicos estandarizados por el CLSI; no obstante, se han diseñado diversos procedimientos con elevada sensibilidad. Para el presente estudio se utilizaron dos métodos:
CAZ
CRO
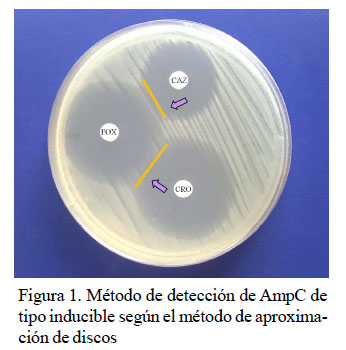
Método de aproximación de discos
FOX
Propuesto por Sanders y Sanders en 1979, aplicable a betalactamasas AmpC inducibles. Se utilizó cefoxitina (30 µg/dl) como inductor a una distancia de 27 mm de centro a centro de la ceftazidima (30 µg/dl) y ceftriaxona (30 µg/dl) como reveladores (Figura 1). El microorganismo producirá una betalactamasa inducible si se observa un halo de inhibición truncado del antimicrobiano sustrato, testigo o revelador (Rojas y Valle, 2009).
Detección de AmpC usando inhibidores específicos
En caso de cepas AmpC plasmídicas constitutivas se utilizan técnicas que incluyan la utilización de inhibidores deAmpC. Se coloca un disco de cloxacilina (500 µg) y a una distancia de 25 mm de centro a centro se colocan discos de ceftazidima (30 µg) y cefotaxima (30 µg). La prueba da positiva para la producción de AmpC cuando se observa un halo de inhibición de las cefalosporinas adyacente a la cloxacilina (Mirelis et al., 2006; Rojas y Valle, 2009).
Detección Fenotípica de Mecanismos de Resistencia a Quinolonas
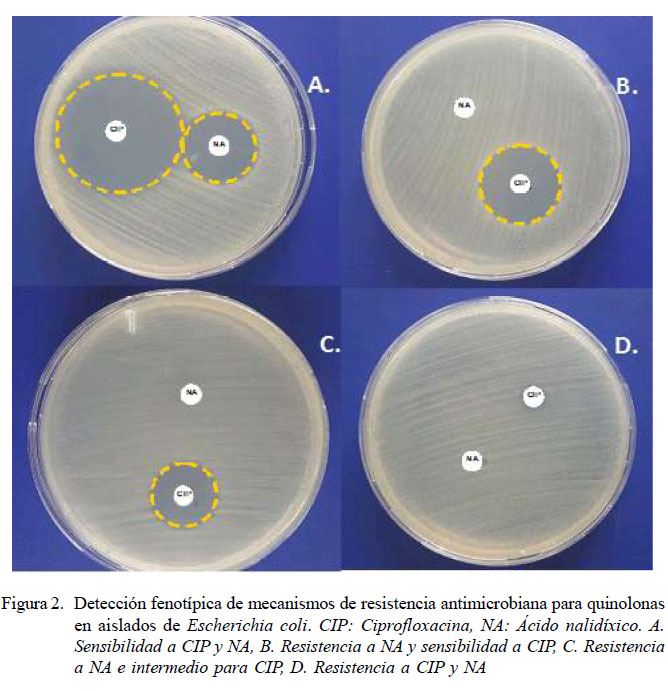
La resistencia es debida a una acumulación de mutaciones en los genes de las topoisomerasas, sobre todo en gyrAy parC (Ruiz, 2003; Jacoby, 2005; Navarro et al., 2011) (Figura 2). Se utilizaron tres protocolos preestablecidos (Navarro et al., 2011):
-Resistencia a ácido nalidíxico y sensibilidad a ciprofloxacino. Probablemente presentan una mutación en gyrA.
-Resistencia a ácido nalidíxico y sensibilidad intermedia a ciprofloxacino. Muy probablemente son aislados con al menos dos mutaciones en gyrA o gyrA+parC.
-Resistencia al ácido nalidíxico y ciprofloxacino. Alta probabilidad de presencia de genes qnr y de otros genes plasmídicos, sin alteraciones adicionales en las topoisomerasas.
Detección Fenotípica de Mecanismos de Resistencia a Aminoglucósidos
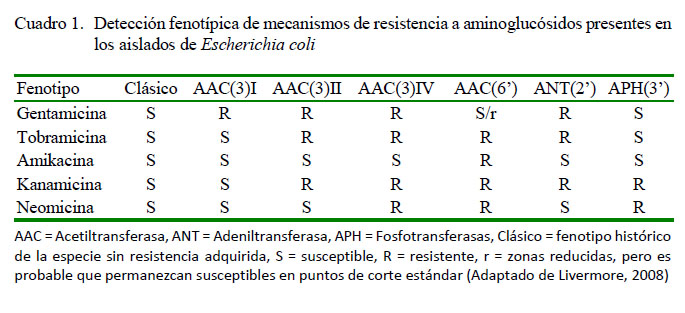
Existe una gran y diversa población de enzimas modificadoras de aminoglucósidos. Su detección se basa en la lectura de los halos de inhibición generados por una gama de aminoglucósidos, obteniéndose según patrón de resistencia exclusivo de una enzima específica. Este método es denominado interpretación interpretativa (Cuadro 1) (Livermore, 2008).
RESULTADOS
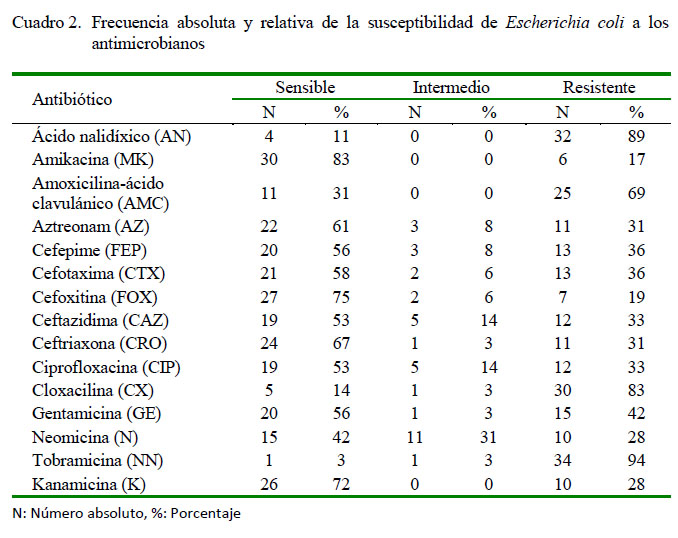
Todos los aislados de E. coli (36/36) fueron resistentes a por lo menos un antimicrobiano (Cuadro 2). Las frecuencias más altas de resistencia fueron para el ácido nalidíxico (89%, 32/36), cloxacilina (83%, 30/36) y amoxicilina-ácido clavulánico (69%, 25/36). No se identificó fenotipo de resistencia compatible con BLEE.
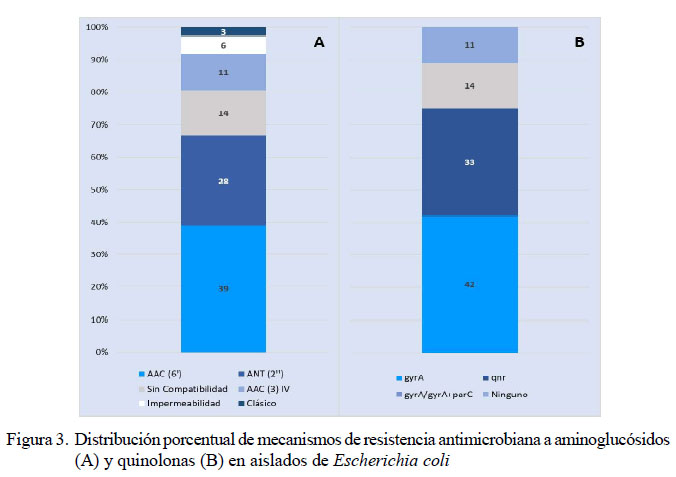
El 3% (1/36) de los aislados presentó fenotipo de resistencia compatible con betalactamasas tipo AmpC. El 11% (4/36) no evidenció mecanismos de resistencia a quinolonas, siendo sensible a ambos antimicrobianos.Así mismo, se evidenció que el 42% (15/36) presentó una mutación en gyrA, el 14% (5/36) presentó al menos dos mutaciones en gyrAo gyrA+parC, yel 33% (12/36) presentó una alta probabilidad la presencia de genes qnr (Figura 3A).
De los 36 aislados estudiados, el 39% (14/36) presentó un patrón de resistencia para la enzima AAC (6’), el 28% (10/36) para la enzima ANT (2") y el 11% (4/36) para la enzima AAC (3) IV. Solo el 3% (1/36) presentó un patrón clásico sin resistencia adquirida, mientras que un 6% (2/36) presentó resistencia a todos los antimicrobianos (impermeabilidad). El 14% (5/36) no presentó compatibilidad con ninguno de los patrones preestablecidos (Figura 3B).
El 86% de los aislados presentó fenotipo multidrogorresistente (MDR), debido a la resistencia a tres o más familias de antimicrobianos.
DISCUSIÓN
El estudio evidenció altos porcentajes de resistencia los aislados de Escherichia coli provenientes de porcinos al ácido nalidíxico, cloxacilina yamoxicilina-ácido clavulánico. El ácido nalidíxico rara vez se utiliza hoy en día debido a su toxicidad (Rubinstein, 2001); sin embargo, es usado en estudios de resistencia para evidenciar posibles mutaciones en genes como gyrAo parC. El alto porcentaje de resistencia encontrado fue similar a lo descrito por Ruiz et al. (2004), quienes reportaron 91% de resistencia, donde la mutación en gyrA es suficiente para generar la resistencia a este antimicrobiano.
Seral et al. (2010) describieron que la sensibilidad a la amoxicilina-ácido clavulánico (AMC) descarta de manera inicial la presencia de alguna betalactamasa de tipo AmpC y que posiblemente una BLEE se la causa de la resistencia. De manera similar, se encontró un alto porcentaje de resistencia para AMC (69%) y la detección de betalactamasa de tipo AmpC en un 3%. Sin embargo, la resistencia a esta combinación pudo deberse a otras razones como una elevada producción de BLEE o su resistencia a inhibidores, IRTs, enzimas OXA, hiperproducción de SHV-1, TEM-1, K1, etc.
Los altos niveles de resistencia (69%) a AMC detectados generan una gran preocupación desde el punto de vista clínico y epidemiológico, ya que esta combinación de antimicrobianos es el tratamiento de primera elección para muchas infecciones por E. coli. La resistencia a los inhibidores de betalactamasas, como el ácido clavulánico en ente-ro-bacterias se ha convertido en un problema de emergencia mundial (Oteo et al., 2008; Di Conza et al., 2014).
Solo el 3% (1/36) presentó un fenotipo de resistencia compatible a AmpC inducible. Este tipo de betalactamasa puede ocasionar fracasos terapéuticos descritos en infecciones causadas por aislados hiperproductores de AmpC inducible en tratamientos con betalactámicos (Navarro et al., 2011).
El 42% (15/36) de los aislados presentó resistencia al ácido nalidíxico y sensibilidad a ciprofloxacina, evidenciando así una posible mutación en gyrA. Esta mutación se da en el codón 87 de gyrA, sin que se hayan detectado mutaciones en parC, lo cual produce un descenso en la susceptibilidad a fluoroquinolonas. No obstante, se requieren mutaciones adicionales en gyrA, gyrBy parC para la generación de resistencia frente a fluoroquinolonas (Miró et al., 2004; Ruiz et al., 2004).
Estudios iniciales en E. coli demostraron que la resistencia a las quinolonas se produce naturalmente por mutaciones en regiones definidas de las proteínas gyrA y en menor medida en gyrB (Yoshida et al., 1991). Además, se ha determinado que mutaciones en las regiones equivalentes de las proteínas ParC o ParE (topoisomerasa IV) producen resistencia a quinolonas (Heisig, 1996). Se ha descrito que una sola mutación no da como resultado una resistencia clínicamente relevante; sin embargo, la combinación de mutaciones en los genes gyrAo gyrBy parCo parE que codifican para las subunidades A y B de topoisomerasas II y IV, respectivamente, genera resistencia (Talens-Visconti et al., 2002).
El 33% (12/36) presentó resistencia al ácido nalidíxico y ciprofloxacino lo cual sugiere una alta probabilidad de la presencia de genes qnr. Rodríguez-Martínez et al. (2011) observaron que las proteínas qnr facilitan la selección de mutantes resistentes a quinolonas de alto nivel.
Martinez-Martínez y Ruiz de Alegría (2009) detallaron los fenotipos implicados en los mecanismos de resistencia asociados a enzimas modificadoras de aminoglucósidos, muchos de los cuales se detectaron en este estudio. Se conocen tres tipos de enzimas modificadoras de aminoglucósidos: Ofosfotransferasas [APHs, genes aphA, strA, strB], N-acetiltransferasas [ACCs, genes aac (3"), aac (6")], y O-adeniltransferasas [ANTs, genes aadA, aadB, también denominados ANT (Michael et al., 2006; van Hoek et al., 2011). La eficacia de los aminoglucósidos disminuye por el surgimiento de las enzimas modificadoras de aminoglucósidos (AME) presentes en en casi todos los géneros bacterianos (Ramirez y Tolmasky, 2010).
El estudio evidenció un alto porcentaje de aislados de Escherichia coli con diversos mecanismos que brindan resistencia a un amplio rango de antimicrobianos. La aparición de enterobacterias resistentes se encuentra relacionada al uso de estos agentes en las producciones pecuarias (WHO, 2014). Por ello, la detección de aislados de E. coli resistentes en porcinos en granjas resalta la necesidad de monitorear el uso controlado de antimicrobianos en la producción.
CONCLUSIONES
-
El 3% (1/36) de los aislados de Escherichia coli evidenció la presencia de betalactamasa de tipo AmpC.
-
El 42% (15/36) evidenció una posible mutación en gyrA, el 33% (12/36) presentó alta probabilidad de genes qnr y el 14% (5/36) al menos dos posibles mutaciones en gyrAo gyrA+ parC.
-
Las enzimas del mecanismo de resistencia a aminoglucósidos expresadas fueron de 39% (14/36) para AAC (6’), 28% (10/36) paraANT (2") y 11% (4/36) para AAC (3) IV.
LITERATURA CITADA
1. [CLSI] Clinical and Laboratory Standards Institute. 2016. CLSI M100- S26 Performance standards for antimicrobial susceptibility testing: twenty-sixth informational supplement. CLSI 36(1) M100-S26. 251 p. [ Links ]
2. Cona E. 2002. Condiciones para un buen estudio de susceptibilidad mediante test de difusión en agar. Rev Chil Infectol 19(Supl 2): 77-81. doi: 10.4067/ S0716-10182002019200001 [ Links ]
3. di Conza JA, Badaracco A, Ayala J, Rodríguez C, Famiglietti A, Gutkind GO. 2014. â-lactamases produced by amoxi cill in-clavul anate-r esi stant enterobacteria isolated in Buenos Aires, Argentina: a new blaTEM gene. Rev Argent Microbiol 46: 210-217. doi: 10.1016/S0325-7541(14)70075-6
4. Famiglietti A, Quinteros M, Vázquez M, Marín M, Nicola F, Radice M, Pasterán F, et al. 2005. Consenso sobre las pruebas de sensibilidad a los antimicrobianos en Enterobacteriaceae. Rev Argent Microbiol 37: 57-66. [ Links ]
5. Heisig P. 1996. Genetic evidence for a role of parC mutations in development of high-level fluoroquinolone resistance in Escherichia coli. Antimicrob Agents Ch 40: 879-885.
6. Jacoby GA. 2005. Mechanisms of resistance to quinolones. Clin Infect Dis 41(Suppl 2): 120-126. doi: 10.1086/ 428052
7. Jarlier V, Nicolas MH, Fournier G, Philippon A. 1988. Extended broadspectrum beta-lactamases conferring transferable resistance to newer betalactam agents in Enterobacteriaceae: hospital prevalence and susceptibility patterns. Rev Infect Dis 10: 867-878. [ Links ]
8. Kikuvi SK, Lee HS, Nam HM, Cho YS, Kim JM, Song SW, Park YH, et al. 2007. Antimicrobial resistance observed in Escherichia coli strains isolated from fecal samples of cattle and pigs in Korea during 2003-2004. Int J Food Microbiol 116: 283-286. doi: 10.1016/j.ijfoodmicro.2006.12.014 [ Links ]
9. Lezameta L, Gonzales-Escalante E, Tamariz JH. 2010. Comparación de cuatro métodos fenotípicos para la detección de beta-lactamasa de espectro extendido. Rev Peru Med Exp Salud Pública 27: 345-351 [ Links ]
10. Lim JY, Hong JB, Sheng H, Shringi S, Kaul R, Hovde CJ. 2010. Phenotypic diversity of Escherichia coli O157:H7 strains associated with the plasmid O157. J Microbiol 48: 347-357. doi: 10.1007/s12275-010-9228-4 [ Links ]
11. Livermore DM. 2008. Defining an extended- spectrum beta-lactamase. Clin Microbiol Infec 14: 3-10. doi: 10.1111/ j.1469-0691.2007.01857.x [ Links ]
12. Martínez--Martínez L, Calvo J. 2010. El problema creciente de la resistencia antibiótica en bacilos gramnegativos: situación actual. Enferm Infec Micr Cl 28: 25--31. doi: 10.1016/S0213- 005X(10)70027-6 [ Links ]
13. Martínez-Martínez L, Ruiz de Alegría C. 2009. Escherichia coli resistente a gentamicina y sensible a amikacina. En: Alós JI, Cantón R, Martínez-Martínez L, Vila J (eds). Atlas del antibiograma. España: Biomérieux. p 141-143. [ Links ]
14. Michael G, Butaye P, Cloekaert A, Shwarz S. 2006. Genes and mutations conferring antimicrobial resistance in Salmonella: an update. Microbes Infect 8: 1898-1914. doi: 10.1016/j.micinf.- 2005.12.019 [ Links ]
15. Mirelis B, Rivera A, Miró E, Mesa R, Navarro F, Coll P. 2006. A simple phenotypic method for differentiation between acquired and chromosomal AmpC β-lactamases in Escherichia coli. Enferm Infec Micr Cl 24: 370-372. doi: 10.1157/13089690 [ Links ]
16. Miró E, Vergés C, García I, Mirelis B, Navarro F, Coll P, Prats G, et al. 2004. Resistencia a quinolonas y betalactámicos en Salmonella enterica, y su relación con mutaciones en las topoisomerasas, alteraciones en la permeabilidad celular y expresión de un mecanismo de expulsión activa. Enferm Infec Micr Cl 22: 204-211. doi: 10.1016/ S0213-005X(04)73067-0 [ Links ]
17. Navarro F, Calvo J, Cantón R, Fernández-Cuenca F, Mirelis B. 2011. Detección fenotípica de mecanismos de resistencia en microorganismos gram-negativos. Enferm Infec Micr Cl 29: 524-534. doi: 10.1016/j.eimc.2011.- 03.011 [ Links ]
18. [OIE] Organización Mundial de la Salud Animal. 2016. Boletín: Manejo adecuado de los antibióticos. [Internet]. Disponible en: http://www.oie.int/esp/ session2017/pdf/E_IF_2016.pdf [ Links ]
19. [OMS] Organización Mundial de la Salud. 2001. Estrategia mundial de la OMS para contener la resistencia a los antimicrobianos. Ginebra: OMS. [Internet]. Disponible en: http:// www.antibioticos.mscbs.gob.es/PDF/ resist_OMS_estrategia_mundial_contra_- resistencias.pdf [ Links ]
20. Oteo J, Campos J, Lázaro E, Cuevas O, García-Cobos, Pérez-Vázquez, de Abajo FJ, et al. 2008. Increased amoxicillin-clavulanic acid resistance in Escherichia coli blood isolates, Spain. Emerg Infect Dis 14: 1259-1262. doi: 10.3201/eid1408.071059 [ Links ]
21. Prescott JF. 2000. Antimicrobial drug resistance and its epidemiology. In: Prescott JF, Baggot JD, Walker RD (eds). Antimicrobial therapy in veterinary medicine. Iowa, USA: Iowa State University Press. p 21-31. [ Links ]
22. Ramirez M, Tolmasky M. 2010. Aminoglycoside modifying enzymes. Drug Resist Update 13: 151-171. doi: 10.1016/j.drup.2010.08.003 [ Links ]
23. Rodríguez-Martínez JM, Cano ME, Velasco C, Martínez-Martínez L, Pascual A. 2011. Plasmid-mediated quinolone resistance: an update. J Infect Chemother 17: 149-182. doi: 10.1007/ s10156-010-0120-2 [ Links ]
24. del Valle D. 2009. Betalactamasas tipo AmpC: generalidades y métodos para detección fenotípica. Rev Soc Venezolana Microbiol 29: 78-83. [ Links ]
25. Rubinstein E. 2001. History of quinolones and their side effects. Chemotherapy 47(Suppl 3): 44-4848. doi: 10.1159/000057838 [ Links ]
26. Ruiz J. 2003. Mechanisms of resistance to quinolones: target alterations, decreased accumulation and DNA gyrase protection. J Antimicrob Chemoth 51: 1109-1117. doi: 10.1093/jac/dkg222
27. Ruiz J, Navia M, Marco F, Vila J. 2004. Mecanismos de resistencia a betalactámicos y ácido nalidíxico en aislados clínicos de Salmonella enterica serotipo hadar y bsilla. Enferm Infec Micr Cl 22:252-253. doi: 10.1016/S0213- 005X(04)73078-5 [ Links ]
28. Sánchez LC, Corrales LC. 2005. Evaluación de la congelación para conservación de especies autóctonas bacterianas. Nova 3: 21-29. [ Links ]
29. Sanders CC, Sanders WE. 1979. Emergence of resistance to cefamandole: possible role of cefoxitin inducible â-lactamases. Antimicrob Agents Chemother 15: 792-797. [ Links ]
30. Seral C, Pardos M, Castillo FJ. 2010. Betalactamasas de espectro extendido en enterobacterias distintas de Escherichia coli y Klebsiella. Enferm Infec Micr Cl 28(Supl 1): 12-18. doi: 10.1016/ S0213-005X(10)70003-3 [ Links ]
31. Stine OC, Johnson JA, Keefer- NA, Perry KL, Tigno J, Qaiyumi S, Stine MS, et al. 2007. Widespread distribution of tetracycline resistance genes in a confined animal feeding facility. Int J Antimicrob Ag 29: 348-352. doi: 10.1016/ j.ijantimicag.2006.11.015 [ Links ]
32. Talens-Visconti, Garrigues TM, Cantón E. 2002. Quinolonas y Streptococcus pneumoniae. Mecanismo de acción y resistencia. Rev Esp Quim 15: 313-324. [ Links ]
33. van Den Bogaard AE, London N, Stobberingh EE. 2000. Antimicrobial resistance in pig faecal samples from the Netherlands (five abattoirs) and Sweden. J Antimicrob Chem 45: 663--671. doi: 10.1093/jac/47.6.763 [ Links ]
34. van Hoek AH, Mevius D, Guerra B, Mullany P, Roberts AP, Aarts HJ. 2011. Acquired antibiotic resistance genes: an overview. Front Microbiol 2: 203. doi: 10.3389/fmicb.2011.00203 [ Links ]
35. [WHO] World Health Organization. 2004. Joint FAO/OIE/WHO Expert workshop on non-human antimicrobial usage and antimicrobial resistance: Scientific assessment. [Internet]. Disponible en: http://www.fao.org/3/a-bq- 500e.pdf [ Links ]
36. Yoshida H, Bogaki M, Nakamura M, Yamanaka LM, Nakamura S. 1991. Quinolone resistance-determining region in the DNA gyrase gyrB gene of Escherichia coli. Antimicrob Agents CH 35: 1647-1650. doi: 10.1128/ AAC.34.6.1271 [ Links ]
Recibido: 28 de marzo de 2018
Aceptado para publicación: 10 de octubre de 2018