Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.30 no.1 Lima ene./mar. 2019
http://dx.doi.org/10.15381/rivep.v30i1.15690
ARTÍCULOS PRIMARIOS
Detección molecular del virus de Lengua Azul en Culicoides insignis y en ovinos de Pucallpa, Perú
Molecular detection of Bluetongue virus in Culicoides insignis and sheep of Pucallapa, Peru
Dennis Navarro M.1, Miguel Rojas M.1, Jessica Jurado P.1, Alberto Manchego S.1, Mercy Ramírez V.1, Ana Castillo E.1, Hermelinda Rivera G.1,3
1 Laboratorio de Microbiología y Parasitología Veterinaria, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima, Perú
2 Estación Experimental del Centro de Investigación IVITA, Pucallpa, Ucayali, Perú
3 E-mail: hriverag@unmsm.edu.pe
RESUMEN
El objetivo del presente estudio fue detectar el ARN del virus de Lengua Azul (VLA) en Culicoides insignis y en muestras de sangre de ovinos de crianza extensiva en una localidad de Pucallpa, Ucayali. En una primera etapa se obtuvieron muestras de sangre de ovinos (n=46) de tres granjas para detectar anticuerpos en suero contra VLA mediante la prueba de inmunodifusión en gel agar (IDGA) y ELISA de competición. El 46.7, 81.3 y 20.0% de ovinos fueron seroreactores por IDGA, de los cuales el 96% fueron positivos a anticuerpos contra VLA mediante la prueba de ELISA. En la segunda etapa se capturaron 1143 Culicoides spp en las granjas seropositivas y se colectaron 15 muestras de sangre de ovinos. Se identificaron 1000 hembras de Culicoides insignis. Las hembras se agruparon en pools de 100 cada uno (C. insignis =10 pools y Culicoides spp = 1). Para el análisis molecular se realizó la extracción de ARN total a partir de las muestras de sangre de ovino y de los Culicoides, seguido de un PCR anidado utilizando dos pares de cebadores específicos que amplificaron el seg 7 de VLA. Solo un pool de C. insignis y una muestra de sangre de ovino evidenciaron una banda de 1070 pb aproximadamente. La presencia del ARN viral en el pool de C. insignis y en el ovino sugieren el posible rol de C. insignis como potencial vector para la transmisión de VLA en los ovinos de las granjas en estudio.
Palabras clave: ovinos; Culicoides spp; anticuerpos; Seg 7; virus de Lengua Azul; trópico
ABSTRACT
The aim of the present study was to detect the RNA of the Bluetongue virus (BTV) in Culicoides insignis and in blood samples of sheep reared under an extensive production system in Pucallpa, Peru. In a first phase, sheep blodd samples (n=46) were obtained from three farms to detect serum antibodies against BTV by agar gel immunodiffusion (AGID) and a competitive ELISA. Results showed that 46.7, 81.3 and 20.0% of sheep had antibodies by IDGA, of which 96% were positive to antibodies against VTV by the ELISA test. In the second phase, 1143 Culicoides spp were captured and 15 sheep blood samples were collected in the seropositive farms. One thousand females of Culicoides insignis were identified. The females were grouped into pools of 100 each (C. insignis = 10 pools and Culicoides spp = 1). For the molecular analysis, the extraction of total RNA was carried out from the ovine blood samples and Culicoides, followed by a nested PCR using two pairs of specific primers that amplified seg 7 of BTV. Only one pool of C. insignis and one sheep blood sample showed a band of approximately 1070 bp. The presence of viral RNA in the pool of C. insignis and in the ovine suggest the possible role of C. insignis as a potential vector for the transmission of BTV in sheep of the farms under study.
Key words: sheep; Culicoides spp; antibodies; Seg 7; Bluetongue virus; jungle
INTRODUCCIÓN
El cambio climático y el calentamiento global están generando cambios en la distribución y abundancia de mosquitos vectores y los arbovirus que ellos transmiten (Hunter, 2003). La propagación y migración de muchas especies de mosquitos ha incrementado la presencia y diseminación de agentes virales hacia países donde no existían reportes de algunas infecciones, ocasionando la ocurrencia de enfermedades emergentes o reemergentes de importancia para la salud animal y pública (Purse et al., 2005). Entre la amplia gama de mosquitos vectores, se cree que los mosquitos Culicoides spp son particularmente más sensibles a cambios climáticos (Purse et al., 2008), debido a que son poiquilotermos y utilizan una gran variedad de hábitats con áreas semiacuáticas para su desarrollo; por tanto, las tasas demográficas y las interacciones con los hospederos son sensibles a variaciones sutiles de temperatura y humedad (Purse et al., 2015).
El género Culicoides Latreille, 1809 (Diptera: Ceratopogonidae) está compuesto por aproximadamente 1400 especies (Harrup et al., 2015). Treinta especies han sido asociadas a nivel mundial a la transmisión del virus de Lengua azul (VLA). Uno de ellos es C. sonorensis, el principal vector en América del Norte (Tabachnick, 1996). En contraste, en América del Sur existe muy poca información sobre los vectores implicados en la transmisión de VLA, siendo Culicoides insignis el posible vector predominante (Gorchs y Lager, 2001), seguido de C. pusillus (Ronderos et al., 2003). En el Perú se han descrito 31 especies de Culicoides, entre ellas C. insignis en Loreto y C. pusillus en Cajamarca y Madre de Dios (Felippe-Bauer et al., 2008), pero no se ha evaluado su implicancia como vectores en la transmisión de VLA u otros agentes virales.
La enfermedad de Lengua azul afecta a rumiantes domésticos y silvestres, siendo los ovinos los más susceptibles. Es producida por el VLA, miembro del grupo Orbivirus de la Familia Reoviridae (Ratinier et al., 2011).
VLA posee un genoma ARN de doble cadena dividido en 10 segmentos que codifican 7 proteínas estructurales y 4 proteínas no estructurales. Las proteínas esturcturales VP2 y VP5 forman parte de la cápside externa y son altamente variables, determinando los serotipos del virus. Las proteínas VP7 y VP3 forman parte del core interno y son las más abundantes, inmunogénicas y constituyen el antígeno del grupo Orbivirus. También los epítopos específicos para VLA, enfermedad hemorrágica epizoótica, y peste equina africana (Ratinier et al., 2011; Purse et al., 2015). Los anticuerpos inducidos por la VP7 posinfección y detectado por pruebas serológicas específicas indica positividad al VLA, pero no identifica al serotipo o serotipos presentes en una determinada zona o área.
VLA presenta 27 serotipos identificados hasta el momento. Esta alta frecuencia de serotipos es debido a su genoma segmentado donde los procesos de recombinacion son frecuentes (Caporale et al., 2014; Purse et al., 2015; Schulz et al., 2016). El VLA es transmitido por dípteros del género Culicoides. El virus replica en las células del sistema vascular de su hospedero mamífero, ocasionando edemas en la cabeza, coronitis en las patas, problemas reproductivos y, además, restringe el comercio internacional de animales en pie y de germoplasma por estar en la lista de enfermedades de declaración obligatoria ante la Organización Mundial de Salud Animal (OIE) (Tabachnick, 1996; Wilson y Mellor, 2009). La enfermedad es endémica en zonas tropicales y subtropicales donde el virus persiste entre algunas especies de Culicoides spp y vertebrados domésticos y silvestres, los cuales se infectan, pero no desarrollan la enfermedad.
En las últimas dos décadas, el VLA ha ocasionado severos brotes en rumiantes domésticos y silvestres en países europeos, ubicados fuera del rango de 40°N y 35°S, como sucedió en España, Bélgica y Alemania, debido a la migración de Culicoides spp a latitudes cada vez mayores donde los rumiantes son susceptibles (Purse et al., 2005; Meroc et al., 2009). En los brotes causados por un solo serotipo del VLA en países europeos, más de 800 000 cabezas de ovinos fueron afectados. Así mismo, los brotes ocurridos en Bélgica entre 2006 y 2007 ocasionaron pérdidas por 180 millones de euros al año (Wilson y Mellor, 2009).
En el Perú, la presencia de VLA solo ha sido demostrada serológicamente en ovinos, camélidos y en animales silvestres de áreas tropicales (Rosadio et al., 1984; Rivera et al., 1987, 2013). Recientemente, Navarro et al. (2018) identificaron a Culicoides insignis en áreas cercanas a granjas de ovinos seropositivos al VLA en Pucallpa, Ucayali, sugiriendo ser los potenciales vectores involucrados en la transmisión de VLA. El objetivo del presente estudio fue detectar el ARN del virus de Lengua azul en ovinos seropositivos y en Culicoides insignis capturados en áreas cercanas a los ovinos en Pucallpa en un contexto de vigilancia epidemiológica del VLA.
MATERIALES Y MÉTODOS
Lugar del Estudio
El estudio fue realizado en tres granjas de ovinos de pelo seropositivos a VLA en la localidad de Santa Rosa de Lima, distrito de Campo Verde, provincia Coronel Portillo, Perú, ubicado una altitud media de 193 m. Los ovinos fueron nativos de la zona; sin embargo, en la década del 80 se importaron ovinos de pelo de Brasil que fueron distribuídos en diversas zonas del trópico central del país (H. Rivera, 2016, Lima comunicación personal), pudiendo haber descendientes cruces de estos animales en la muestra. La captura de los mosquitos fue relizada en la misma localidad.
Muestras
En una primera etapa se colectaron muestras de sangre de ovinos mayores a seis meses y aparentemente sanos (n=46) provenientes de tres granjas de ovinos. Las muestras fueron obtenidas de la vena yugular o cefálica utilizando tubos al vacío sin anticoagulante y centrifugadas a 800 g por 5 min para la obtención del suero en la Estación Pucallpa del Centro de Investigación IVITA. Los sueros resultantes fueron transportados al laboratorio de virología de la Facultad de Medicina Veterinaria (FMV) de la Universidad Nacional Mayor de San Marcos (UNMSM) en Lima y conservados en congelación a -20 °C hasta su análisis.
En una segunda etapa se realizó la captura de 1143 Culicoides spp en las granjas positivas a VLA, de los cuales 1000 fueron hembras y 43 fueron machos. Los machos fueron descartados del estudio pues las hembras sonn las vectores. La captura se hizo siguiendo la metodología utilizada por Navarro et al. (2018). Ademas, se tomaron muestras de sangre adicionales de ovinos seropositivos (n=15) de la vena yugular o cefálica utilizando tubos al vacío con anticoagulante EDTA. Los mosquitos capturados se identificaron con el apoyo de la Sección de Entomología del Instituto de Medicina Tropical Daniel Alcides Carrión de la Facultad de Medicina de la UNMSM y el Instituto de Ecología (INECOL) de Xalapa, Veracruz (México). Las hembras fueron agrupadas en pools (100 especímenes por pool). En total se tuvieron 10 pools de C. insignis y 1 pool de Culicoides spp. Tanto los pools de mosquitos Culicoides como la sangre entera de los ovinos fueron conservados en nitrógeno líquido (-196 °C).
Detección de Anticuerpos
Para la detección de anticuerpos contra el grupo Orbivirus (virus de Lengua azul y virus de la Enfermedad Hemorrágica Epizoótica [VEHE]) se utilizó la prueba de inmunodifusión en gel agar (IDGA) (VMRD, EEUU). Así mismo, los sueros fueron analizados por la prueba de ELISA de competición, basada en un anticuerpo monoclonal competitivo, el cual es específico para distinguir anticuerpos contra VLA (VMRD, EEUU). El desarrollo de ambas pruebas fue siguiendo las especificaciones del fabricante.
ARN Total de las Muestras
Los pools de Culicoides fuerón machacados en un mortero con nitrogeno líquido y fueron homogenizados con 1.5 ml de PBS (pH 7.4). El machacado fue centrifugado a 2000 g por 10 min a 4 ºC. El sobrenadante fue filtrado en membrana de nitrocelulosa con diámetro de poro de 0.22 µm. La suspensión obtenida y la sangre de ovino fueron utilizadas para la extracción de ARN total empleando el kit comercial Norgen’s Total RNA Purification (Cód. 37500, Norgen, Canadá).
Sintesis de ADN Complementario (ADNc)
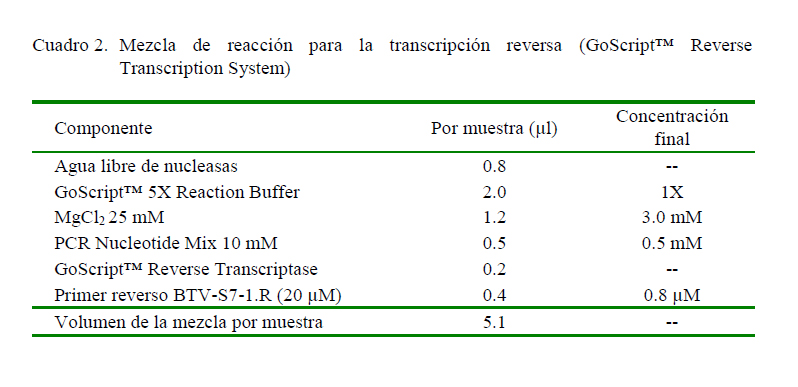
El ARN extraido fue utilizado como molde para la síntesis de ADN complementario (ADNc) empleando el kit comercial GoScript™ Reverse Transcription System (Promega, EEUU) y el cebador antisentido BTV-S7-1.R (Cuadro 1). Previamente se desnaturó 5 µl del ARN extraído a 97 ºC por 5 min, y luego fue mantenido a 4 ºC por 5 min para añadirle 5 µl de la mezcla de reacción para la transcripción reversa, según se indica en el Cuadro 3. El protocolo de retrotrascripción fue 25 ºC por 5 min, 42 ºC durante 45 min y luego 4 ºC por 5 min.
PCR y PCR Anidado
Las pruebas de PCR y PCR anidado fueron realizadas empleando el kit comercial GoTaq® Colorless (Promega, EEUU) y los cebadores especificos para amplificar la secuencia del segmento 7 de VLA, donde el primer par de cebadores amplifican un producto de 1156 pb y el segundo par de 1070 pb (Cuadro 1).
Para la primera PCR, la elaboración de la mezcla de reacción siguió las instrucciones del fabricante con 0.8 µM de cada cebador BTV-S7-1F y BTV-S7-1R (Cuadro 1). Las condiciones para la amplificación de los cebadores fueron 95 °C durante 5 min como desnaturación inicial, seguido de 40 ciclos de 95 °C durante 40º s, 44 °C durante 1 min, 72 °C por 2 min y una extensión final de 72 °C por 10 min.
Para el PCR anidado se siguieron igualmente las instrucciones del fabricante, con 0.8 µM del segundo par de cebadores BTV- S7-2F y BTV-S7-2R (Cuadro 1). Se utilizó 5 µl del ADN amplificado de la primera PCR en diferentes diluciones. Las condiciones para la amplificación de los cebadores fueron 95 °C durante 5 min como desnaturacion inicial, seguido de 40 ciclos de 95 °C durante 40º s, 54 °C durante 1 min, 72 °C por 2 min, y una extensión final de 72 °C por 10 min.
Todos los productos de PCR fueron sometidos a electroforesis en gel de agarosa (1.5% de agarosa en 0.5X TBE) por 1 h y teñidos con bromuro de etidio para su visualización por irradiación UV.
RESULTADOS
Análisis Serológico
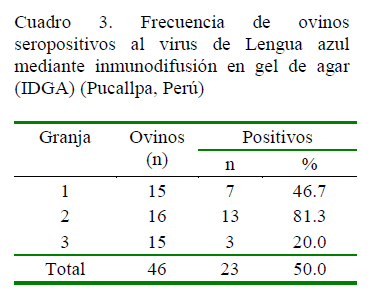
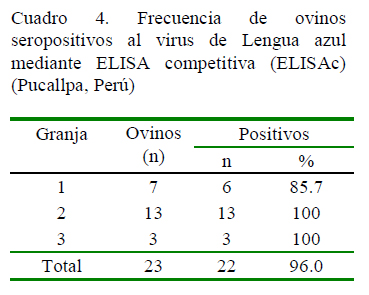
El 50% (23/46) de las muestras de suero de ovino presentaron anticuerpos contra VLA o VEHE (Cuadro 3), mediante la prueba de IDGA. Las muestras positivas y débiles positivas por IDGA fueron analizadas por el ELISA de competición, resultando que el 96% (22/23) presentaron anticuerpos contra VLA (Cuadro 4).
En el presente estudio no se incluyen los resultados del análisis del secuenciamiento del segmento 7 y del segmento 2 del ARN del VLA detectado en el pool de C. insignis y en la sangre del ovino de la misma área para conocer la relación molecular del viral en ambas especies y conocer el serotipo presente.
Detección del Segmento 7 del VLA
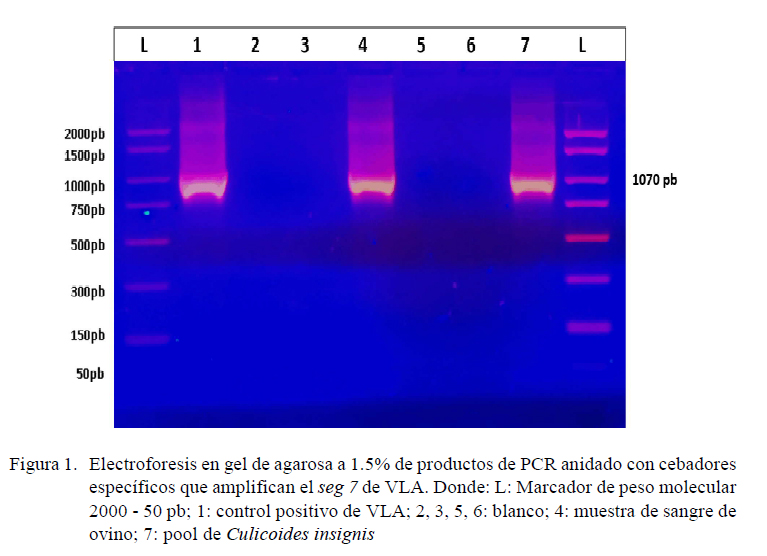
En solo uno de los 11 pooles de Culicoides spp y en una de las 15 muestras de sangre de ovino se observó la banda de 1070 pb que corresponde al segmento 7 de VLA (Figura 1).
DISCUSIÓN
El ecosistema, así como la presencia de ovinos, caballos y aves de corral en el lugar de estudio fueron condiciones favorables para la presencia y actividad de los mosquitos del género Culicoides (Mellor et al., 2000; Purse et al., 2015). Al examen clínico, los ovinos de las granjas no presentaron lesiones en los epitelios de la boca ni en los bordes coronarios de las patas. Por otro lado, las granjas se caracterizaron por la presencia de humedales, charcos, arbustos y heces en descomposición; características que representan las diferentes zonas de descanso y reproducción de mosquitos (Purse et al., 2015).
El 50% de los ovinos presentaron anticuerpos contra el grupo Orbivirus detectado por la prueba de IDGA, y de estos, el 96% correspondió a anticuerpos contra VLA determinado por ELISAc, ya que solo una muestra positiva al serogrupo Orbivirus fue negativa a anticuerpos contra el VLA (Cuadros 3 y 4). La prueba de ELISAc posee 99% de especificidad y 100% de sensiblidad en la detección del VLA, ya que interviene un anticuerpo monoclonal que detecta un epitopo específico en la proteína VP7 del VLA (Reddington et al., 1991; VMRD, 2005), a diferencia de la prueba de IDGA. La muestra de suero del animal seropositivo por IDGA, pero negativo a ELISAc, podría tratarse de una reacción cruzada entre VEHE y VLA.
En América del Sur, anticuerpos contra el VLA han sido detectados en ovinos de Ecuador (8%), Venezuela (94.7%) y Argentina (95%), en bovinos de Colombia (51.8%), y en búfalos (25.9-93%) y pecaries (39%) de Brasil (Lager, 2004). Cabe mencionar que algunos de estos resultados serológicos han sido realizados mediante la prueba de IDGA, por lo que es difícil afirmar que los anticuerpos detectados sean inducidos por VLA, ya que existen evidencias de la presencia del VEHE en la región sudamericana (Homan et al., 1985; Verdezoto et al., 2018). No obstante, estos estudios demuestran la amplia distribución del VLA, así como, uno o más vectores competentes de su transmisión en el continente sudamericano (Meiswinkel et al., 2004;
Darpel et al., 2011). El desarrollo de los signos clínicos de la enfermedad depende si la infección es endémica o no. La infección es endémica en el trópico y subtrópico por las constantes infecciones y reinfecciones por el virus de uno u otro serotipo y, como consecuencia, los animales presentan anticuerpos, pero rara vez presentan signos clínicos (MacLachlan, 2004; Caporale et al., 2014).
Navarro et al. (2018), en un estudio previo, capturaron 7930 mosquitos, de los cuales el 94.8% fueron identificados como Culicoides insignis, indicando la abundancia de esta especie de mosquitos en el área de estudio. Resultados similares fueron reportados en el noreste de Brasil (98%) (Silva y Carvalho, 2013), 86% en la amazonia brasileña (Carvalho et al., 2017) y 91.8% en Coratei-Paraguay (Ronderos et al., 2003), entre otros, lo cual indica que C. insignis es una especie de amplia distribución en la región (Lager, 2004) y, que al parecer, está presente en toda la amazonia peruana (FelippeBauer et al., 2008). C. insignis es también la especie involucrada en la transmisión del VLA en el sureste de los EEUU, el Caribe, y en América Central y del Sur (MacLachlan et al., 2007), de allí que estos mosquitos podrían ampliar su rango de distribución altitudinal debido al cambio climático (LorcaOró et al., 2014), yendo hacia zonas de mayor altitud, constituyendo un riesgo para el desarrollo de la ganadería andina en general.
La detección de una banda de 1070 pb correspondiente al segmento 7 que codifica la proteína estructural interna VP7 del VLA detectado en un pool (1/1000) (0.1%) de C. insignis y en una muestra (1/15) (6.6%) de sangre de ovinos (Figura 1) indican que C. insignis es al menos uno de los potenciales vectores de VLA y que los anticuerpos detectados en los ovinos de las granjas en estudio fueron inducidos por la VP7 que es la proteína interna (core) más abundante, inmunogénica y conservada entre los 27 serotipos existentes del virus (Bouet-Cararo et al., 2014). La presencia de la banda correspondiente al segmento 7 en muestras de C. insignis, a diferencia del pool de Culicoides spp, indica que este mosquito podría ser uno de los principales vectores en Perú, como lo es en otros países de América del Sur y del Caribe (Lager, 2004; Meiswinkel et al., 2007).
La detección de la banda del segmento 7 del ARN viral en un pool de C. insignis y en una muestra de sangre de ovino podría deberse a un bajo indice de infección de VLA en estos mosquitos durante la época del muestreo, como lo indica Gerry et al. (2001). Sin embargo, en estudios de infección experimentales en C. insignis con dos serotipos distintos se demostró que la tasa de infección con el serotipo 2 fue entre 45 y 62% y con el serotipo 11 entre 20 y 60% (Tanya et al., 1992). Otro estudio similar a nivel de campo con C. sonorensis, otro vector en América del Norte, América Central y en la región del Caribe, se encontró que el índice de infección del serotipo 10 fue de 0.4% en un periodo de tres años con variaciones en un lapso de cuatro semanas de 0 a 2.2%, en zonas donde la enfermedad de Lengua azul es mayormente de tipo subclínico (Gerry et al., 2001), situación muy similar al Perú, donde existen animales seropositivos, pero sin enfermedad clínica.
Tabachnick (2013) lista varios posibles efectos del clima sobre los vectores y sus posibles influencias sobre la transmisión de enfermedades. Es así que el incremento de la temperatura influye directamente sobre la capacidad vectorial, incluso la ubicación y tipo de trampas de captura (McDermott et al. 2015). Un ejemplo es C. brevitarsis, el cual posee baja competencia vectorial; es decir, que la habilidad del vector para soportar la replicación viral es baja; sin embargo, debido a que posee un alto indice de picadura por día es un importante vector de VLA en Australia. En contraste, C. fulvus cuya competencia es mayor, pero posee una capacidad vectorial baja, sobre todo debido a su baja abundancia y distribución geografica, lo cual disminuye su potencial de transmisión (Mullens et al., 2015).
La detección del segmento 7 del VLA en una muestra de ovino de apariencia normal indica que el animal estuvo virémico al momento del muestreo. El periodo de viremia en el ovino raramente persiste por más de 14 días, a diferencia del bovino cuyo estado de viremia puede ser de 90-120 días (Alvarez et al., 2017). En Brasil, con un ecosistema similar a la amazonia peruana, han ocurrido brotes de la enfermedad clínica de Lengua azul en ovinos de Paraná y de Rio de Janeiro, con signos clínicos de letargia, hipermotilidad intestinal, pirexia, edema facial e hiperemia, pequeñas ulceraciones en la lengua y laminitis (Clavijo et al., 2002; Alvarez et al., 2014). Asimismo, cervidos neotropicales en cautiverio han manifestado la enfermedad de Lengua azul en Brasil (Kawanami, 2016).
La enfermedad de Lengua azul es conocida hace más de 100 años, cuando afectó a ovinos Merinos introducidos a Sudáfrica. Su distribución es global desde la mitad del siglo 20 con excepción de la Antártida. Su amplia distribución e impacto en la ganadería justificó para ser incluida en la Lista de Enfermedades de Declaración Obligatoria a la OIE (Maclachlan, 2010). En el Perú no hay reportes de seroprevalencia del VLA en los diversos ecosistemas fuera de las zonas tropicales, como los valles internadinos y andinos donde se desarrolla la ganadería ovina, bovina y de camélidos, y menos aún reportes de ocurrencia clínica de la enfermedad de Lengua azul.
El Perú puede ser uno de los países con mayores riesgos para la emergencia de enfermedades transmitidos por mosquitos ya que posee diversos ecosistemas propicios para el desarrollo de Culicoides, los cuales podrían encontrar una población de ovinos u otros rumiantes susceptibles a VLA. Ante el inminente cambio climático se considera necesario la determinación de la seroprevalencia de VLA en zonas ganaderas, identificar los diversos serotipos presentes en el país, levantar un mapa de la ubicación de las diversas especies de Culicoides spp, etc., con el fin de conocer la epidemiología y establecer la vigilancia sanitaria no solo de VLA, sino también de otras enfermedades emergentes transmitidas por mosquitos.
CONCLUSIÓN
Se determinó la presencia del seg 7 del virus Lengua azul en un pool (1/10) de hembras Culicoides insignis y en una muestra de sangre (1/15) de ovino seropositivo de granjas de crianza extensiva de Pucallpa, Ucayali.
Agradecimiento
Los autores agradecen al Blgo. Mg. Abraham Cáceres por sus valiosas opiniones y aportes sobre la identificación de los Culicoides spp capturados en Pucallpa, Ucayali. Así mismo, los autores agradecen al MV Mg. Juan Rondon Espinoza por su apoyo en los trabajos de campo.
Literatura citada
1. Alvarez MF, Amaral PC, da Silva R, Zandonadi F. 2017. Língua azul: desmistificando a doença. Rev Acad Ciênc Anim 15: 21-31. doi: 10.7213/academica.15.S02.2017.A04 [ Links ]
2. Alvarez MF, dos Santos M, del Fava C, de Oliveira GR, Pituco EM, Zandonadi FZ. 2014. Outbreak of Bluetongue virus serotype 4 in dairy sheep in Rio de Janeiro, Brazil. J Vet Diagn Invest 26: 567-570. doi: 10.1177/1040638714538020 [ Links ]
3. Bouet-Cararo C, Contreras V, Caruso A, Top S, Szelechowski M, Bergeron C, et al. 2014. Expression of VP7, a Bluetongue virus group specific antigen by viral vectors: analysis of the induced immune responses and evaluation of protective potential in sheep. Plos One 9: e111605. doi: 10.1371/journal.pone.0111605.g003 [ Links ]
4. Caporale M, Gialleornardo L, Janowics A, Wilkie G, Shaw A, Savini G, Van Rijn PA, et al. 2014. Virus and host factors affecting the clinical outcome of Bluetongue virus infection. J Virol 68: 10399-10411. doi: 10.1128/JVI.01641-14 [ Links ]
5. Carvalho LPC, Pereira AM, Farias ES, Almeida JF, Rodrigues MS, Resadore F, et al. 2017. A study of Culicoides in Rondônia, in the BrazilianAmazon: species composition, relative abundance and potential vectors. MedVet Entomol 31: 117122. doi: 10.1111/mve.12208 [ Links ]
6. Clavijo A, Sepulveda L, Riva J, Pessoa-Silva M, Tailor-Ruthes A, Lopez JW. 2002. Isolation of Bluetongue virus serotype 12 from an outbreak of the disease in South America. Vet Rec 151: 301-302. doi: 10.1136/vr.151.10.301 [ Links ]
7. Darpel KD, Langner KFA, Nimtz M, Anthony SJ, Brownlie J, Takamatsu H, Mellor PS, et al. 2011. Saliva proteins of vector Culicoides modify structure and infectivity of Bluetongue virus particles. PLos One 6: e17545. doi: 10.1371/journal.pone.0017545. [ Links ]
8. Felippe-Bauer ML, Cáceres A, da Silva C, Valderrama-Bazan W, Gonzales-Perez A, Martins J. 2008. New records of Culicoides Latreille (Diptera: Ceratopogonidae) from Peruvian Amazonian region. Biota Neotrop 8: 33- 38. doi: 10.1590/S1676-060320080-00200002 [ Links ]
9. Gerry AC, Mullen B A, Maclachlan NJ, Mecham JO. 2001. Seasonal transmission of Bluetongue virus by Culicoides sonorensis (Diptera: Ceratopogonidade) at a southern California dairy and evaluation of vectorial capacity as a preditor of Bluetongue virus transmission. J Med Entomol 38: 197-209. [ Links ]
10. Gorchs C, Lager I. 2001. Lengua azul. Actualización sobre el agente y la enfermedad. RevArgent Microbiol 33: 122-132. [ Links ]
11. Harrup LE, Bellis GA, Balenghien T, Garros C. 2015. Culicoides Latreille (Diptera: Ceratopogonidae) taxonomy: current challenges and future directions. Infect Genet Evol 30: 249-266. doi: 10.1016/j.meegid.2014.12.018 [ Links ]
12. Homan EJ, Taylor WP, Ruiz LD, Yuill TM. 1985. Bluetongue virus and epizootic haemorrhagic disease of deer virus serotypes in northern Colombian cattle. J Hyg-Cambridge 95: 165-172. doi: 10.1017/S0022172400062409 [ Links ]
13. Hunter P. 2003. Climate change and waterborne and vector-borne disease. J Appl Microbiol 94: 37-46. doi: 10.1046/ j.1365-2672.94.s1.5.x [ Links ]
14. Kawanami AE. 2016. Vírus da Língua azul em cervídeos netropicais e bovídeos domésticos. Tesis Doctoral. Jaboticabal: Universidade Estadual Paulista. 99 p. [ Links ]
15. Lager IA. 2004. Bluetongue virus in South America: overview of viruses, vectors, surveillance and unique features. Vet Ital 40: 89-93. [ Links ]
16. Legisa D, Gonzalez F, de Stefano G, Pereda A, Dus Santos MJ. 2013. Phylogenetic analysis of Bluetongue virus serotype 4 field isolates from Argentina. J Gen Virol 94: 652-662. doi: 10.1099/vir.0.046896-0 [ Links ]
17. Lorca-Oró C, López-Olvera JR, Ruiz- Fons F, Acevedo P, García-Bocanegra I, Oleaga Á, Pujols J. 2014. Long term dynamics of Bluetongue virus in wild ruminants: relationship with outbreaks in livestock in Spain. Plos One 9: e100027. doi: 10.1371/journal.-pone.-0100027 [ Links ]
18. MacLachlan NJ, Zientara S, Stallknecht DE, Boone JD, Goekjian VH, Sailleau C, Balasuriya UB. 2007. Philogenetic comparison of the S10 genes of recent isolates of Bluetongue virus from United State and French Martinique island. Virus Res 129: 236-240. doi: 10.1016/j.virusres.2007.07.009 [ Links ]
19. MacLachlan NJ. 2004. Bluetongue: pathogenesis and duration of viraemia. Vet Ital 40: 462-467. [ Links ]
20. MacLachlan NJ. 2010. Global implication of the recent emergence of Bluetongue virus in Europe. Vet Clin N Am-Food A 26: 163-171. doi: 10.1016/j.cvfa.2009.10.012 [ Links ]
21. McDermott EG, Mayo CE, Gerry AC, Laudier D, Maclachlan NJ, Mullens BA. 2015. Bluetongue virus intection creates light averse Culicoides vectors and serious erros in transmission risk estimates. Parasite Vector 8: 460. doi: 10.1186/s13071-015-1062-4 [ Links ]
22. Meiswinkel R, Gomulski LM, Delecolle JC, Goffredo M, Gasperi G. 2004. The taxonomy of Culicoides vector complexes unfinished business. Vet Ital 40: 151-159. [ Links ]
23. Meiswinkel R, van Rijn PA, Leijs P, Goffredo M. 2007. Potential new Culicoides vector of Bluetongue virus in northern Europe. Vet Rec 161: 564565. doi: 10.1136/vr.161.16.564 [ Links ]
24. Mellor PS, Boorman J, Baylis M. 2000. Culicoides biting midges: their role as arbovirus vectors. Annu Rev Entomol 45:307-340. doi: 10.1146/ annurev.ento.45.1.307 [ Links ]
25. Meroc E, Herr C, Verheyden B, Hooyberghrs J, Houdart P, Raemaekers M, Vandenbussche F, et al. 2009. Bluetongue in Belgium: episody II. Transbound Emerg Dis 56: 3948. doi: 10.1111/j.1865-1682.2008.01063.x [ Links ]
26. Mullens BA, McDemott EG, Gerry AC. 2015. Progress and knowledge gaps in Culicoides ecology and control. Vet Ital. 51: 313-323. doi: 10.12834/VetIt.560.2675.1 [ Links ]
27. Navarro D, Rivera H, Cáceres A, Rondon J. 2018. Identificación morfológica de Culicoides spp descritos como transmisores de orbivirus capturados en granjas de ovinos de Pucallpa Perú. Rev Inv Vet Perú 29: 302-309. doi: 10.15381/rivep.v29i1.14203 [ Links ]
28. Purse BV, Mellor PS, Rogers DJ, Samuel AR, Mertens PP, Baylis M. 2005. Climate change and the recent emergence of Bluetongue in Europe. Nat Rev Microbiol 3: 171-181. doi: 10.1038/nrmicro1090 [ Links ]
29. Purse BV, Brown HE, Harrup L, Mertens PP, Rogers DJ. 2008. Invasion of Bluetongue and other orbivirus infections into Europe: the role of biological and climatic processes. Rev Sci Tech OIE 27: 427-442. [ Links ]
30. Purse BV, Carpenter S, Venter GJ, Bellis G, Mullens BA. 2015. Bionomics of temperate and tropical Culicoides midges: knowledge gaps and consequences for transmission of Culicoidesborne viruses. Annu Rev Entomol 60: 373-392. doi: 10.1146/annurev-ento-010814-020614 [ Links ]
31. Ratinier M, Caporale M, Golder M, Franzoni G, Allan K, Nunes SF, Armezzani A, et al. 2011. Identification and characterization of a novel nonstructural protein of Bluetongue virus. Plos Pathog 7: e1002477. doi: 10.1371/journal.ppat.1002477 [ Links ]
32. Reddington JJ, Reddington GM, MacLachlan NJ. 1991. A competetive ELISA for detecction of antibodies to the group antigen of Bluetongue virus. J Vet Diagn Invest 3: 144-147. doi: 10.1177/104063879100300207 [ Links ]
33. Rivera H, Madewell BR, Ameghino E. 1987. Serological survey of viral antibodies in the Peruvian alpaca (Lama vicugna). Am J Vet Res 48: 189-191. doi: 10.1046/j.1439-0450.1999.00215.x [ Links ]
34. Rivera H, Cárdenas L, Ramírez M, Manchego A, More J, Zúñiga A, Romero M. 2013. Infección por orbivirus en huanganas (Tayassu pecari) de Madre de Dios. Rev Inv Vet Perú 24: 544550. doi: 10.15381/rivep.v24i4.2738 [ Links ]
35. Ronderos MM, Greco NM, Spinelli GR. 2003. Diversity of biting midges of the genus Culicoides Latreille (Diptera: Ceratopogonidae) in the area of the Yacyreta Dam Lake between Argentina and Paraguay. Mem I Oswaldo Cruz 98: 19-24. doi: 10.1590/S0074-0276200-3000100003 [ Links ]
36. Rosadio RH, Evermann JF, DeMartini JC. 1984. A preliminary serological survey of viral antibodies in Peruvian sheep. Vet Microbiol 10: 91- 96. doi: 10.1016/0378-1135(84)90059-2 [ Links ]
37. Schulz C, Bréard E, Sailleau C, Jenckel M, Viarouge C, Vitour D, Palmarini M, et al. 2016. Bluetongue virus serotype 27: detection and characterization of two novel variants in Corsica, France. J Gen Virol 97: 20732083. doi: 10.1099/jgv.0.000557 [ Links ]
38. Silva FS, Carvalho LPC. 2013. A population study of the Culicoides biting midges (Diptera: Ceratopogonidae) in urban, rural, and forested sites in a cerrado area of northeastern Brazil. Ann Entomol Soc Am 106: 463-470. doi: 10.1603/AN12047 [ Links ]
39. Tabachnick WJ. 1996. Culicoides variipennis and Bluetongue-virus epidemiology in the United States. Annu Rev Entomol 41: 23-43. doi: 10.1146/annurev.en.41.010196.000323 [ Links ]
40. Tabachnick WJ. 2013. Changes in predicting climate and environment effects on vector-borne disease episystems in a changing word. J Exp Biol 213: 946-954. doi: 10.1242/jeb.037564 [ Links ]
41. Tanya VN, Greiner EC, Gibbs EPJ. 1992. Evaluation of Culicoides insignis (Diptera: Ceratopogonidae) as a vector of Bluetongue virus. Vet Microbiol 32: 114. doi: 10.1016/0378-1135(92)90002-B [ Links ]
42. Verdezoto J, Breard E, Viarouge C, Quenault H, Lucas P, Sailleau C, Zientera S, et al. 2018. Novel serotype of Bluetongue virus in South America and first report of epizootic haemorrhagic disease virus in Ecuador. Transbound Emerg Dis 65: 244-247. doi: 10.1111/tbed.12625 [ Links ]
43. Wilson AJ, Mellor PS. 2009. Bluetongue in Europe: past, present and future. Philos TR R Soc B 364: 26692681. doi: 10.1098/rstb.2009.0091 [ Links ]
Recibido: 6 de junio de 2018
Aceptado para publicación: 2 de noviembre de 2018