Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.30 no.2 Lima abr./jun. 2019
http://dx.doi.org/10.15381/rivep.v30i2.15407
ARTÍCULOS PRIMARIOS
Caracterización molecular ómica de una cepa de Bacillus amyloliquefaciens aislada de la microbiota del paiche Arapaima gigas con actividad antagonista contra bacterias patógenas de peces
Omic molecular characterization of a Bacillus amyloliquefaciens strain isolated from the paiche Arapaima gigas microbiota with antagonistic activity against fish pathogenic bacteria
Manuel Feria1,5, Arnaldo Castañeda1, Odalis Toledo1, Deysy Castillo3 Mario Cueva3, Virna Cedeño2,4
1 Universidad Nacional de Tumbes, Perú
2 Instituto de Investigación Inca’Biotec S.A.C, Tumbes, Perú
3 Cooperativa de Trabajadores BioteCoop, Tumbes, Perú
4 Concepto Azul, Guayaquil, Ecuador
5 E-mail: manuelfezevallos@gmail.com
RESUMEN
Se analizaron las características de una cepa de Bacillus amyloliquefaciens aislada del intestino de Arapaima gigas, seleccionada por su capacidad para generar inhibición de múltiples bacterias patógenas de peces, mediante el uso de herramientas moleculares como PCR y espectrometría de masas MALDI TOF/TOF. Los resultados mostraron a través de PCR que esta cepa cuenta con genes claves para la generación de péptidos antimicrobianos como bmyB (bacillomycin L synthetase B), fenD (fengycin sintetasa), srfAA (subunidad 1 surfactin sintetasa), bacA (proteína de biosíntesis de bacilysin) e iturin (iturin A). Además, mediante el análisis por espectrometría de masas se detectaron bacteriocinas (plipastatin, gramicidin, fengycin, surfactin), proteínas de fijación al intestino (like-enolase), proteinas transportadoras de péptidos antimicrobianos (ABCtransporters) y proteínas de estimulación del sistema inmunitario como flagelin. También se detectaron proteínas tipo colagenasas, chitinasas y xilosa-isomerasas que contribuyen con el proceso de digestión y asimilación. Todos estos resultados permiten considerar los múltiples beneficios de esta cepa para ser utilizada como probiótico en el cultivo de peces.
Palabras clave: Bacillus amyloliquefaciens; MALDI TOF; probióticos; Arapaima gigas
ABSTRACT
The characteristics of a strain of Bacillus amyloliquefaciens isolated from the intestine of Arapaima gigas, selected for its ability to generate inhibition of multiple pathogenic bacteria of fishes were analyzed by using molecular tools such as PCR and mass spectrometry MALDI TOF / TOFM. The results showed through PCR that this strain has key genes for the generation of antimicrobial peptides such as bmyB (bacillomycin L synthetase B), fenD (fengycin synthetase), srfAA (subunit 1 surfactin synthetase), bacA (bacilysin biosynthesis protein) and iturin (iturin A). In addition, by mass spectrometry analysis were detected bacteriocins (plipastatin, gramicidin, fengycin, surfactin), intestine binding proteins (like-enolase), antimicrobial peptide transport proteins (ABC-transporters) and immune system stimulation proteins like flagelin. Collagenase, chitinases and xylose-isomerases proteins were also detected that contribute to the digestion and assimilation process. All these results allow to consider the multiple benefits of this strain to be used as a probiotic in fish culture.
Key words: Bacillus amyloliquefaciens; MALDI TOF; probiotic; Arapaima gigas
INTRODUCCIÓN
La acuicultura se ha convertido en una de las principales actividades productivas a nivel mundial, incluso superando por primera vez en 2014 la contribución de la pesca extractiva para el consumo humano (FAO, 2016). La presión de la demanda obliga a aumentar las densidades de los cultivos, lo que lleva a generar enormes riesgos, en lo que se refiere a la calidad del agua y al manejo del estrés animal. Estas condiciones pueden desencadenar brotes infecciosos que comprometen los niveles productivos (Telli et al., 2014; Banerjee y Ray, 2017).
Las enfermedades causadas por bacterias son las más comunes en el cultivo de peces de agua dulce (Mukherjee y Ghosh, 2016). Convencionalmente, el uso de los antibióticos se elige para controlar las enfermedades que atacan los cultivos, pero esta estrategia se encuentra cuestionada porque se considera una fuente de contaminación ambiental (Ayandiran et al., 2014), afecta a los microorganismos beneficiosos y genera un desequilibrio en el tracto gastrointestinal de los peces (Kesarcodi-Watson et al., 2008). Además, su uso no controlado genera bacterias resistentes a los antibióticos, pudiendo convertirse en un riesgo para la salud pública (Banerjee y Ray, 2017).
Estratégicamente, se propone el uso de probióticos, que se definen como organismos vivos que al ser administrados en cantidades adecuadas confieren beneficios a la salud del huésped (Araya et al., 2002). Estos microorganismos tienen múltiples ventajas para el animal, como promover el crecimiento (Midhun et al., 2017), mejorar la respuesta inmune, aumentar la actividad digestiva (Nayak, 2010), modular la colonización microbiana, controlar enfermedades y mejorar la calidad del agua (Chi et al., 2014).
Entre los géneros bacterianos con mayor atención para la investigación y desarrollo de probióticos, se encuentras especies del género Bacillus (Silva et al., 2015). Entre ellas, la especie Bacillus amyloliquefaciens es un antagonista de amplio espectro (Kaewklom et al., 2013) y tiene la capacidad de producir enzimas con actividad digestiva (Wang et al., 2008).
Actualmente, a través del uso de herramientas genómicas y enfoques dirigidos al análisis funcional, mediante la caracterización de las proteinas (proteómica, MALDI TOF TOF), se pueden identificar determinadas características que contribuyan a la selección de bacterias para su uso como probióticos en la acuicultura (Lippolis et al., 2011; Vinusha et al., 2018).
El objetivo del presente estudio fue conocer las características funcionales de mayor relevancia en una cepa de Bacillus amyloliquefaciens aislada a partir del tracto digestivo de paiche Arapaima gigas, mediante el uso de herramientas como la espectrometría de masas (shotgun MALDI TOF TOF) para conocer las principales proteinas producidas por esta cepa. Además, a través de técnicas como PCR, determinar la presencia de genes clave para la biosíntesis de péptidos antimicrobianos.
MATERIALES Y MÉTODOS
Cepa Bacteriana y Condiciones de Cultivo
Bacillus amyloliquefaciens 11CI-2 fue aislada del intestino de Arapaima gigas «paiche» en un estudio previo (Castillo, 2017). La cepa almacenada en caldo soya tripticaseína (TSB) con 18% de glicerol a -20 °C fue subcultivada en medio solido agar de soja tríptico (TSA) a 37 °C durante 18 horas, y posteriormente transferida a caldo TSB a 37 °C en tubos de 1.5 ml, para su posterior utilización en los ensayos.
Actividad Antimicrobiana
Bacillus amyloliquefaciens 11CI-2 fue enfrentada a bacterias patógenas de peces tales como Aeromonas hydrophila, Pseudomonas aeruginosa, Plesiomonas shigelloides, Vibrio sp, Aeromonas veronii, Klebsiella sp, Citrobacter freundii. Se realizaron ensayos de enfrentamiento célula vs. célula, con extracto de proteínas del sobrenadante libre de células de un cultivo de la cepa B. amyloliquefaciens. Los ensayos fueron realizados mediante el método de difusión en agar modificado de Balcázar et al. (2008). El agar se perforó con una pipeta Pasteur estéril, generando un pocillo de 0.6 mm, donde se añadieron 20 µl del cultivo bacteriano o del extracto de proteína del sobrenadante libre de células (dependiendo del ensayo) a las placas previamente inoculadas con las bacterias patógenas. Se determinó la capacidad de inhibición a través de la medición del diámetro de la zona de inhibición. Se consideró como resultado negativo una capacidad de inhibición de 6 mm, como leve de 6 a 11 mm, como moderada entre 11 y 16 mm y como fuerte mayor a 16 mm.
Sensibilidad Antibiótica
La susceptibilidad se evaluó con seis antibióticos comerciales: vancomicina (30 µg), gentamicina (10 µg), eritromicina (15 µg), cloranfenicol (30 µg), amikacina (30 µg), amoxicilina + ácido clavulánico (30 µg). El procedimiento fue adaptado de (Ramesh et al., 2014). Para esto, se inoculó la cepa de B. amyloliquefaciens 11CI-2 (108 UFC/ml) en medio solido agar de soja tríptico (TSA) con un asa de Drigalski; posteriormente se colocaron los discos con antibióticos sobre el medio y se incubaron a 37 °C por 24 horas. La zona de inhibición fue registrada en milímetros (mm).
Actividad Proteolítica
Este ensayo se realizó para demostrar la producción de componentes enzimáticos con actividad proteolítica. En agar Skim Milk (10%) se realizaron orificios de 6 mm de diámetro y se inocularon 30 µl de la cepa bacteriana. Se incubó por 24 horas a 37 °C. La actividad proteolítica se vio revelada en la aparición de zonas claras alrededor de la zona de inhibición.
Análisis Genómico
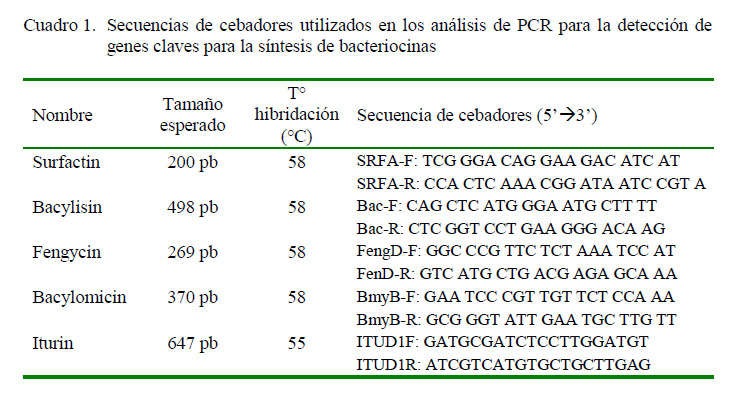
La cepa bacteriana fue cultivada en caldo soya tripticaseína (TSB) durante 24 horas a 30 °C. Posteriormente se hizo la extracción de ADN con el método de bromuro de cetil-trimetil amonio (CTAB) basado en el protocolo de Worden (2009). La amplificación de los genes claves para la biosíntesis de péptidos antimicrobianos (surfactin, bacylisin, bacylomicin, fengycin e iturin A) fue realizada mediante la técnica de reacción en cadena de la polimerasa (PCR) de punto final. Para cada reacción se utilizó 2.5 µl de buffer 10X, 1 µl de cloruro de magnesio (MgCl ) a 50 mM, 0.1 unidad de Taq polimerasa recombinante (Gen-On), 0.5 µl de una mezcla de dNTPs a una concentración de 10 mM, 0.6 µl de cada cebador a 15 pmol, en un volumen final de 25 µl y 100 ng de ADN genómico molde. La detección de los principales genes de biosíntesis de péptidos antimicrobianos fue realizada con iniciadores específicos, los cuales son descritos por Mora et al. (2011) y Ramarathnam et al. (2007). Las secuencias elegidas correspondieron a bmyB (bacillomycin L synthetase B), fenD (fengycin sintetasa), ituA (iturin A), srfAA (subunidad 1 surfactin sintetasa), bacA (proteína de biosíntesis de bacilysin). Las condiciones de ciclos para la amplificación de todos los genes fueron las siguientes: 95 °C por 5 min, 35 ciclos de 95 °C por 30 s, temperatura de alineamiento por 45 s y 72 °C por 45 s. Un paso de extensión final a 72 °C por 5 min seguido por uno de conservación a 4 °C. La temperatura de alineamiento fue a 58 °C para bmyB, fenD, srfAA, y bacA, mientras que para ituA fue 55 °C. La amplificación de los genes estudiados se hizo con los cebadores detallados en el Cuadro 1. Los productos obtenidos por PCR fueron secuenciados por la empresa MACROGEN (https://www.macrogenusa.com, Rockville, USA) y las secuencias obtenidas fueron alineadas en la base de datos libre NCBIBLAST.
Extracción de Proteínas Celulares
Las proteínas celulares fueron obtenidas siguiendo las indicaciones de Sharifuzzaman et al. (2011) con algunas modificaciones. El cultivo bacteriano se realizó añadiendo 150 µl de un precultivo de 18 horas a 37 °C de la cepa de Bacillus amyloliquefaciens 11CI-2 en un matraz de 50 ml que contenía 15 ml de caldo TSB y se incubó a 37 °C a 4 g y fue cosechado cuando alcanzó su fase de crecimiento exponencial en 0.D 600 de 1.0. El componente celular fue recuperado mediante centrifugación a 1792 g a 4 °C por 20 min. La extracción de proteínas se realizó con el Kit QIAGEN, 2011, siguiendo las indicaciones del fabricante.
Extracción de Proteínas Extracelulares
Siguiendo el método de Sharifuzzaman et al. (2011) y Shankar et al. (2012) modificado, se extrajeron las proteínas presentes en el sobrenadante de un cultivo de 24 h a 4 g y 37 °C de la cepa de B. amyloliquefaciens 11CI-2 en un matraz de 250 ml que contenía 50 ml de caldo TSB. Los sobrenadantes se separaron de los sedimentos bacterianos mediante centrifugación (1792 g, 15 min, 4 °C) y pasaje a través de un filtro Milipor de 0.22 ìm. Las proteínas de la fracción libre de células se precipitaron mediante la adición de ácido tricloacético (TCA) al 100%, helado (concentración final 10% w/v) y fenil metilsulfonil fluoruro (PMSF), un inhibidor de proteasa a una concentración final de 1 mM. La precipitación se realizó durante la noche a 4 ºC. Los sedimentos de proteína se suspendieron y se lavaron dos veces mediante centrifugación metanol y fueron secados al ambiente. Finalmente, el sedimento se resuspende en agua de grado HPLC con TFA al 0.1% y se almacena a -20 ° C para su posterior uso.
Separación por SDS PAGE y Digestión Enzimática Directa
Las muestras de proteínas, tanto celulares como extracelulares, fueron sometidas a dos procesos. En uno se realizó la separación de las proteínas totales mediante un gel de poliacrilamida (SDSPAGE) al 15% con un campo eléctrico de 90 V por 2-3 horas. Las bandas seleccionadas se cortan manualmente con ayuda de un bisturí estéril, y son sometidas al tratamiento de tripsinización, siguiendo el protocolo de Shevchenko et al. (2007). Por otro lado, los extractos totales de las proteínas celulares y extracelulares fueron además digeridos de manera directa mediante un tratamiento con tripsina, siguiendo el protocolo de Schmidt et al. (2009).
Procesamiento y Análisis de Datos
El análisis de proteínas se obtuvo a partir de un sistema analizador proteómico MALDITOF/TOF (5800, AB SCIEX System). Los espectros fueron captados en modo MS Reflector Positivo con un láser de Nd: YAD de 349 nm de longitud de onda, con una intensidad de laser de 2800 y una velocidad de 600 µm/ segundo y 750 disparos. Para el análisis de doble tiempo de vuelo (TOF/TOF), los precursores fueron acelerados a 8 kV y se seleccionaron en la puerta de entrada de iones. Todos los fragmentos peptídicos generados por la colisión de los precursores en la cámara de disociación inducida (CID) se aceleraron a 15 kV en la fuente 2 y las masas se analizaron después de pasar por el reflector de iones.
El análisis de los datos se realizó de manera automática por el programa 4000 Series Explorer v. 3.5.3 (Applied Biosystem). Todos los datos obtenidos por MS/MS fueron procesados y analizados por el software ProteinPilotTM 4.5, basado en el algoritmo Paragon™ 4.5.0.0 (Matrix Science, Boston, MA), usando el GPS Explorer software v. 3.6, que permite la búsqueda no redundante de proteínas. Para los análisis se consideró las condiciones de alquilación con iodoacetamida y digestión con tripsina. Las bases de datos asociadas al género Bacillus fueron descargados de los repositorios virtuales libres como UniProt (http://www.uniprot.org/) y de la base virtual del Centro Nacional de Información Biotec-nológicaNCBI (http://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE=Proteins)
RESULTADOS
Actividad Antimicrobiana
La cepa B. amyloliquefaciens demostró tener capacidad de inhibir la mayoría bacterias patógenas de peces seleccionadas de estudios anteriores y que pertenecen al cepario de bacterias de IncaBiotec S.A.C. La cepa solo no mostró inhibición contra Pseudomonas putida (Cuadro 2.)
Sensibilidad Antibiótica y Actividad Proteolítica
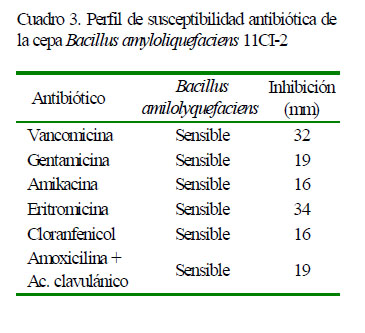
La cepa fue susceptible a todos los antibióticos contra los que fue evaluada. Los antibióticos con mayor inhibición fueron eritromicina y vancomicina (Cuadro 3). Por otro lado, la cepa B. amyloliquefaciens 11CI2 mostró una fuerte capacidad proteolítica, mostrada en los halos claros formados en el medio sólido alrededor del pocillo donde fue inoculada la cepa (19 mm).
Identificación de Genes de Biosíntesis
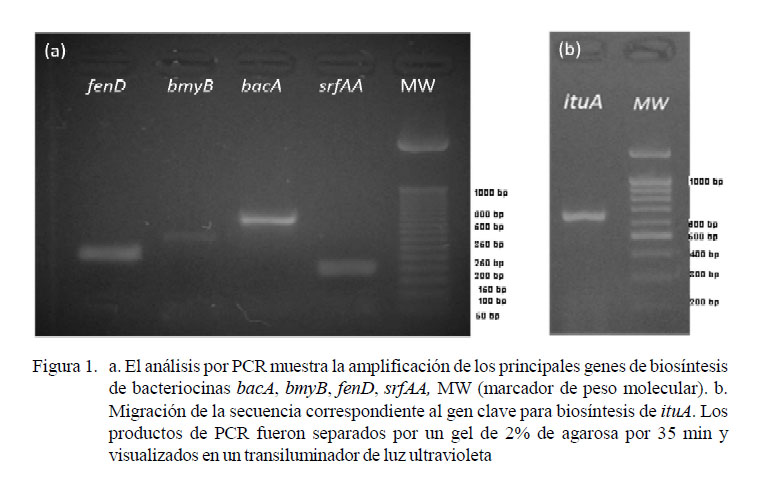
Se obtuvieron cinco productos correspondientes a los tamaños de ituA (647 bp), bacA (498 bp), bmyB (370 bp), fenD (269 bp), srfAA (200 pb) (Figura 1). Las secuencias analizadas por BLAST obtuvieron más del 90% de cobertura e identidad en la búsqueda de homología con sus respectivos genes de biosíntesis; por lo que se determinó la presencia de genes clave para la producción de péptidos antimicrobianos como iturin A (código accesión GenBank KP967589.1), bacillomicina L sintetasa B (bmyB) (código accesión GenBank KY111359.1), la proteína de biosíntesis de bacilisina (bacA) (código accesión GenBank MF098752.1), fengycin sintetasa (fenD) (código accesión GenBank KP453873.1) y la subunidad 1 de la surfactin sintetasa (srfAA) (código accesión GenBank KY051728.1).
Análisis MALDI TOF-TOF
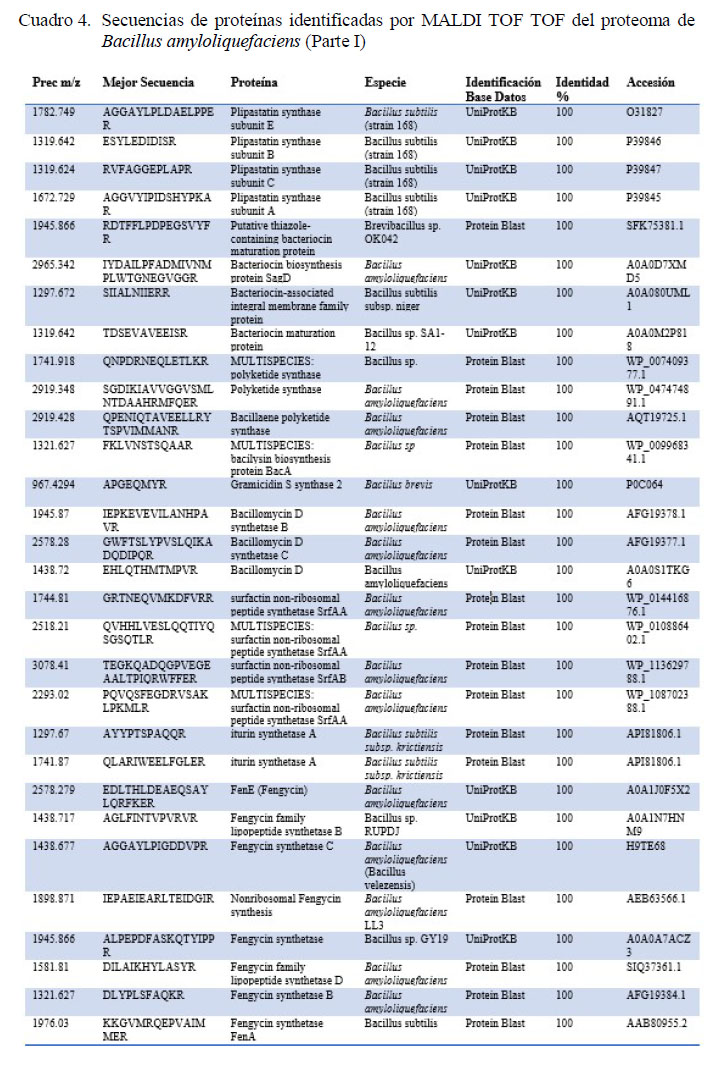
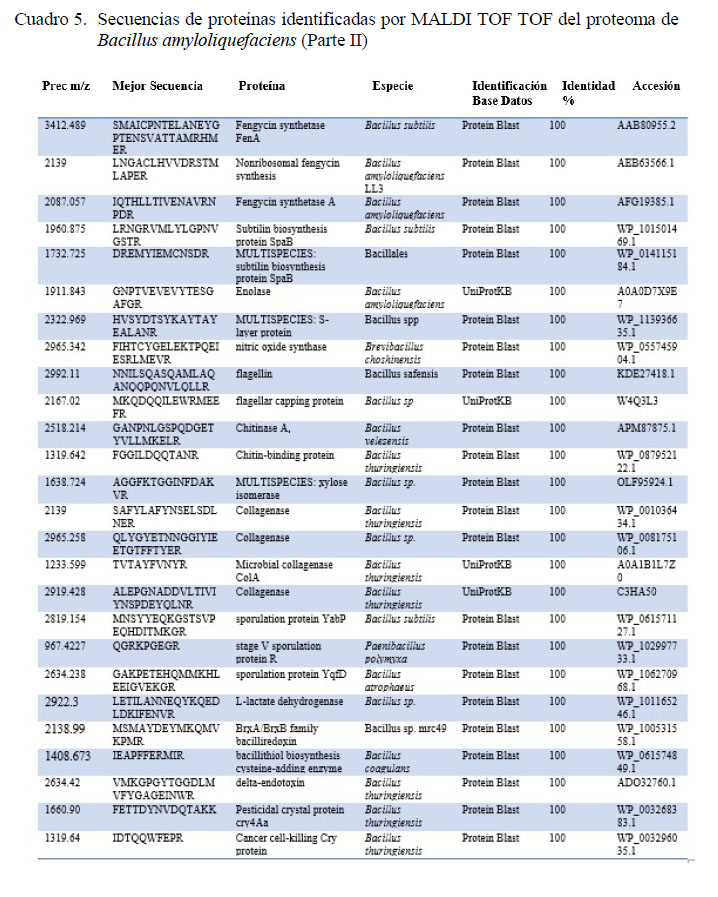
Mediante espectrometría de masas doble TOF se pudo identificar secuencias de las proteínas claves involucradas en funciones como la biosíntesis de péptidos antimicrobianos no ribosomales del grupo de lipopéptidos antimicrobianos como surfactin, bacillomycin D, fengycin e iturin; del grupo de policétidos antibióticos como bacillaene y el dipeptido antimicrobiano bacylisin; y de bacteriocinas como gramicidin, bacteriocina tipo streptolysin (SagD) y lantibióticos como subtilin (Cuadros 4 y 5). Por otro lado, se encontraron además proteínas relacionadas a otras características como la capacidad de la cepa bacteriana a fijarse en el epitelio intestinal como enolase, y proteina de capa S (S-layer). También se encontraron proteínas relacionadas a la digestión de chitina (chitinasa), colagenasa y del aprovechamiento de azucares a través de xilosa isomerasa (Cuadros 4 y 5).
La esporulación es un mecanismo clave para que una bacteria sea considerada para su uso en la alimentación animal. Mediante el uso de la espectrometría de masas MALDI TOF TOF se reconocieron proteínas claves para realizar procesos celulares como la proteína de esporulación YqfD, YabP y proteína R del estado V. Además, se pudieron detectar componentes esenciales para el microorganismo y su supervivencia en ambientes ácidos como bacillithiol (Cuadros 4 y 5). Así mismo, con esta técnica se pudieron identificar moléculas claves en la estimulación del sistema inmunitario del hospedero como flagellin, así como una sintasa de óxido nítrico, la cual funciona como una principal molécula de respuesta celular y protección de la mucosa intestinal. Además, se identificaron secuencias correspondientes a proteínas cristalinas, clave para la inhibición de insectos patógenos (Cuadros 4 y 5).
DISCUSIÓN
Diversas características son tomadas en cuenta para la selección de una cepa como probiótica. Entre estas, la capacidad para generar inhibición contra bacterias patógenas (Thankappan et al. 2015; Nandi et al., 2017). El aislado bacteriano identificado como Bacillus amyloliquefaciens 11CI-2 fue probado in vitro contra bacterias patógenas de importancia en la piscultura, como Aeromonas hydrophila, Pseudomonas aeuruginosa y Citrobacter freundii (Eissa et al., 2010; Thanigaivel et al., 2015; Thankappan et al., 2015), demostrando tener capacidad antagónica frente a todas las cepas patógenas.
La sensibilidad frente a antibióticos es una característica resaltante en cepas probióticas, pues evita la posible transferencia de genes de resistencia entre cepas probióticas y patógenas que podrían habitar al hospedero; considerando además a la cadena alimentaria como una vía de transferencia de bacterias patógenas antibióticos resistentes a los humanos (do Vale Pereira et al., 2017). Los resultados demostraron que la cepa Bacillus amyloliquefaciens 11CI-2 es susceptible a todos los antibióticos contra la que fue probada, semejante a lo mostrado por Kadaikunnan et al. (2015).
Los componentes antimicrobianos producidos por bacterias son abundantes y diversos, y la capacidad de producirlos es considerada una característica principal cuando se trata de selección de bacterias probióticas. Estas bacteriocinas favorecen el establecimiento del probiótico dentro del tracto intestinal, la inhibición de cepas patógenas invasoras y la modulación de la microbiota hospedera. La determinación de los genes claves para la biosíntesis de bacteriocinas como surfactin, fengycin, bacyllomicin, bacylisin e iturin A, mediante la prueba de PCR, al igual que en los estudios con Bacillus spp de Koumoutsi et al. (2004), Ramarathnam et al. (2007) y Athukorala et al. (2009) permitieron confirmar la presencia de estos genes en la cepa aislada B. amyloliquefaciens 11CI-2.
La capacidad de las bacterias para producir enzimas proteolíticas es muy importante desde el punto de vista nutricional. Estas proteasas potencian la digestión, y están involucradas en los mecanismos de defensa contra agentes patógenos (Thankappan et al., 2015); por lo tanto, mediante el crecimiento sobre agar Skim Milk se demostró la capacidad proteolítica de la cepa B. amyloliquefaciens 11CI-2 coincidiendo con múltiples antecedentes sobre la evaluación de bacterias del género Bacillus (Bairagi et al., 2002; Ray et al., 2012; Banerjee et al., 2013; Nandi et al. 2017; Kavitha et al., 2018).
Actualmente, las comunidades bacterianas que componen la microbiota asociada al tracto digestivo de animales y humanos se encuentra bastante estudiada a partir de herramientas de secuenciamiento masivo del ADN (metagenómica); sin embargo, la información obtenida no ayuda a caracterizar la funcionalidad microbiana (Vinusha et al., 2018), la cual podría ser descrita haciendo uso de herramientas de análisis de transcritos o de proteínas; así pues, el análisis proteómico de cepas potencialmente probióticas puede dar información sobre los potenciales beneficios de una cepa de este tipo (Ruíz et al., 2016). En esta investigación, a través del uso de la espectrometría de masas MALDI TOF TOF se identificaron las características de mayor importancia en las cepas potencialmente probióticas a través del análisis de su proteoma celular y extracelular.
Debido a la producción de péptidos con capacidad de inhibir cepas patógenas, diversos trabajos se han centrado en la investigación de cepas de Bacillus capaces de sintetizar lipopéptidos antimicrobianos y antivirales como surfactin (Athukorala et al., 2009; Sajitha et al., 2016); iturin y fengycin (Kim et al., 2010), bacillomycin D (Ramarathnam et al., 2007) y el dipéptido bacilysin (Wu et al., 2014). Aparte de estos péptidos identificados por MALDI TOF TOF y PCR, se pudo demostrar la presencia de proteínas claves para la biosíntesis de bacilleane, gramicidin y subtilin, componentes antimicrobianos previamente descritos (Moldenhauer et al. 2007; Liou et al. 2015; Chen et al., 2018).
Otras moléculas fueron además detectadas mediante espectrometría de masas como las sintasas de óxido nítrico, moléculas clave para la generación de óxido nítrico (NO), el cual está relacionado a la activación del sistema inmunitario a través de la regulación de la comunicación de células de defensa del huésped (Wang et al., 2010). Así mismo, se identificó flagellin, una proteína capaz de incrementar la producción de péptidos defensinas (β-defensin 2) (Schlee et al., 2007).
La capacidad de aprovechar y degradar componentes como la chitina, es una característica muy útil para mejorar el proceso de digestión y asimilación en peces, a través de su microbiota asociada (Dutta et al., 2015). En el presente estudio, se logró identificar mediante proteómica la presencia de enzimas chitinasas de la cepa de B. amyloliquefaciens 11 CI-2. Además, se identificaron enzimas colagenasas y xilosa isomerasa, involucradas en la nutrición de los peces (Ray et al., 2012).
Para que una bacteria con características probióticas pueda prosperar en un ambiente difícil como el tracto digestivo, es necesario que resista las condiciones de estrés, y para esto, mecanismos como la esporulación son muy importantes (Elshaghabee et al., 2017). En el presente estudio se identificaron proteínas claves para la esporulación como YqfD (Wemhoff y Meinhardt, 2013) y proteínas claves para la biosíntesis de bacillithiol y bacilliredoxin, que sirven como mecanismos de resistencia a estrés ácido (Gaballa et al., 2010; Chi et al., 2011; Chandrangsu et al., 2017). Por otro lado, la cepa probiótica debe poseer características que le permitan fijarse al tracto digestivo y prosperar, como por ejemplo proteínas de anclaje y comunicación celular (Castaldo et al., 2009), para interactuar con proteínas como la fibronectina de la matriz extracellular y proteínas de capa S (S-layer) (Sánchez et al., 2009).Ambas proteínas pudieron ser identificadas en este estudio a través del análisis por MALDI TOF TOF.
CONCLUSIONES
Los datos obtenidos de la cepa Bacillus amyloliquefaciens 11 CI-2 aislada de tracto digestivo de paiche Arapaima gigas, ponen en evidencia las buenas características de esta cepa para ser considerada como probiótico para su empleo en la acuicultura de peces.
Agradecimiento
«Círculo de Investigación en biotecnología molecular para el desarrollo y sostenibilidad de los sectores acuícolas del Perú». Código 132-2015.
Literatura citada
1. Araya M, Morelli L, Reid G, Sanders ME, Stanton C, Pineiro M, Ben Embarek P. 2002. Guidelines for the evaluation of probiotics in food. Joint FAO/WHO Working Group report on drafting guidelines for the evaluation of probiotics in food. Ontario, Canada. [Internet]. Available in: https://www.who.int/foodsafety/fs_management/en/probiotic_-guidelines.pdf [ Links ]
2. Athukorala SNP, Fernando WGD, Rashid KY. 2009. Identification of antifungal antibiotics of Bacillus species isolated from different microhabitats using polymerase chain reaction and MALDI-TOF mass spectrometry. Can J Microbiol 55: 1021-1032. doi: 10.1139/W09-067 [ Links ]
3. Ayandiran TA, Ayandele AA, Dahunsi SO, Ajala OO. 2014. Microbial assessment and prevalence of antibiotic resistance in polluted Oluwa River, Nigeria. Egyptian J Aquatic Res 40: 291- 299. doi: 10.1016/j.ejar.2014.09.002 [ Links ]
4. Bairagi A, Ghosh KS, Kumar S, Sen R, Kumar A. 2002. Enzyme producing bacterial ûora isolated from ûsh. Aquacult Int 10: 109-121. doi: 10.1023/A:1021355406412 [ Links ]
5. Balcázar JL, Vendrell D, de Blas I, Ruiz-Zarzuela I, Muzquiz JL, Girones O. 2008. Characterization of probiotic properties of lactic acid bacteria isolated from intestinal microbiota of fish. Aquaculture 278: 188-191. doi: 10.1016/ j.aquaculture.2008.03.014 [ Links ]
6. Banerjee G, Ray AK. 2017. The advancement of probiotics research and its application in fish farming industries. Res Vet Sci 115: 66-77. doi: 10.1016/j.rvsc.2017.01.016 [ Links ]
7. Banerjee G, Ray AK, Askarian F, Ringø E. 2013. Characterisation and identification of enzyme-producing autochthonous bacteria from the gastrointestinal tract of two Indian airbreathing fish. Benef Microbes 4: 277- 284. doi: 10.3920/BM2012.0051 [ Links ]
8. Castaldo C, Vastano V, Siciliano RA, Candela M, Vici M, Muscariello L, Marasco R, et al. 2009. Surface displaced alfa-enolase of Lactobacillus plantarum is a fibronectin binding protein. Microb Cell Fact 8: 14. doi: 10.1186/1475-2859-8-14 [ Links ]
9. Castillo D. 2017. Caracterización molecular de la microbiota intestinal de alevines de paiche Arapaima gigas y selección de cepas potencialmente probióticas. Tesis de Maestría. Tumbes, Perú: Univ. Nacional de Tumbes. 38 p. [ Links ]
10. Chandrangsu P, Loi VV, Antelmann H, Helmann JD. 2017. The role of bacillithiol in gram-positive firmicutes. Antioxid Redox Sign 28: 445-462. doi: 10.1089/ars.2017.7057 [ Links ]
11. Chen L, Gu W, Xu HY, Yang GL, Shan XF, Chen G, Wang CF, Qian AD. 2018. Complete genome sequence of Bacillus velezensis 157 isolated from Eucommia ulmoides with pathogenic bacteria inhibiting and lignocellulolytic enzymes production by SSF. 3 Biotech 8: 114. doi: 10.1007/s13205-018-1125-2 [ Links ]
12. Chi BK, Gronau K, Mäder U, Hessling B, Becher D, Antelmann H. 2011. S-bacillithiolation protects against hypochlorite stress in Bacillus subtilis as revealed by transcriptomics and redox proteomics. Mol Cell Proteomics 10: M111.009506. doi: 10.1074/mcp.M111.009506 [ Links ]
13. Chi C, Jiang B, Yu XB, Liu TQ, Xia L, Wang GX. 2014. Effects of three strains of intestinal autochthonous bacteria and their extracellular products on the immune response and disease resistance of common carp, Cyprinus carpio. Fish Shellfish Immun 36: 9-18. doi: 10.1016/j.fsi.2013.10.003 [ Links ]
14. do Vale-Pereira G, da Cunha DG, Pedreira-Mourino JL, Rodiles A, Jaramillo-Torres A, Merrifield DL. 2017. Characterization of microbiota in Arapaima gigas intestine and isolation of potential probiotic bacteria. J Appl Microbiol 123: 1298-1311. doi: 10.1111/jam.13572 [ Links ]
15. Dutta D, Banerjee S, Mukherjee A, Ghosh K. 2015. Selection and probiotic characterization of exoenzyme-producing bacteria isolated from the gut of Catla catla (Actinopterygii: Cyprini-formes: Cyprinidae). Acta Ichthyol Piscat 45: 373-384. doi: 10.3750/AIP2015.45.4.05 [ Links ]
16. Eissa NME, El-Ghiet EA, Shaheen AA, Abbass A. 2010. Characterization of Pseudomonas species isolated from tilapia «Oreochromis niloticus» in Qaroun and Wadi-El-Rayan lakes, Egypt. Global Veterinaria 5: 116-121. [ Links ]
17. Elshaghabee FMF, Rokana N, Gulhane RD, Sharma C, Panwar H. 2017. Bacillus as potential probiotics: status, concerns, and future perspectives. Front Microbiol 8: 1490. doi: 10.3389/fmicb.2017.01490 [ Links ]
18. [FAO] Organización de las Naciones Unidas para la Alimentación y la Agricultura. 2016. El estado mundial de la pesca y la acuicultura 2016. Contribución a la seguridad alimentaria y la nutrición para todos. Roma. [Internet]. Disponible en: http://www.fao.org/3/ai5555s.pdf [ Links ]
19. Silva TA, Petrillo TR, Yunis-Aguinaga J, Marcusso P, Claudiano G, Moraes F, Moraes JR. 2015. Effects of the probiotic Bacillus amyloliquefaciens on growth performance, hematology and intestinal morphometry in cage-reared Nile tilapia. Lat Am J Aquat Res 43: 963971. doi: 10.3856/vol43-issue5-fulltext-16 [ Links ]
20. Gaballa A, Newton GL, Antelmann H, Parsonage D, Upton H, Rawat M, Claiborne A, et al. 2010. Biosynthesis and functions of bacillithiol, a major lowmolecular-weight thiol in Bacilli. P Natl Acad Sci Usa 107: 6482-6486. doi: 10.1073/pnas.1000928107 [ Links ]
21. Kadaikunnan S, Rejiniemon SS, Khaled JM, Alharbi NS, Mothana R. 2015. In-vitro antibacterial, antifungal, antioxidant and functional properties of Bacillus amyloliquefaciens. Ann Clin Microb Anti 14: 9. doi: 10.1186/s12941015-0069-1 [ Links ]
22. Kaewklom S, Lumlert S, Kraikul W, Aunpad R. 2013. Control of Listeria monocytogenes on sliced bologna sausage using a novel bacteriocin, amysin, produced by Bacillus amyloliquefaciens isolated from Thai shrimp paste (Kapi). Food Control 32: 552-557. doi: 10.1016/j.foodcont.2013.01.012 [ Links ]
23. Kavitha M, Raja M, Perumal P. 2018. Evaluation of probiotic potential of Bacillus spp isolated from the digestive tract of freshwater fish Labeo calbasu (Hamilton, 1822). Aquaculture Reports 11: 59-69. doi: 10.1016/j.aqrep.2018.07.001 [ Links ]
24. Kesarcodi-Watson A, Kaspar H, Lategan MJ, Gibson L. 2008. Probiotics in aquaculture: the need, principles and mechanisms of action and screening processes. Aquaculture 274: 1-14. doi: 10.1016/j.aquaculture.2007.11.019 [ Links ]
25. Kim P I, Ryu J, Kim YH, Chi YT. 2010. Production of biosurfactant lipopeptides iturin A, fengycin, and surfactin A from Bacillus subtilis CMB32 for control of Colletotrichum gloeosporioides. J Microbiol Biotechn 20: 138-145. doi: 10.4014/jmb.0905.05007 [ Links ]
26. Koumoutsi A, Chen XH, Henne A, Liesegang H, Hitzeroth G, Franke P, Vater J, et al. 2004. Structural and functional characterization of gene clusters directing nonribosomal synthesis of bioactive cyclic lipopeptides in Bacillus amyloliquefaciens Strain FZB42. J Bacteriol 186: 1084-1096. doi: 10.1128/JB.186.4.1084-1096.2004 [ Links ]
27. Liou JW, Hung YJ, Yang CH, Chen YC. 2015. The antimicrobial activity of gramicidin a is associated with hydroxyl radical formation. Plos One 10: 5-7. doi: 10.1371/journal.pone.0117065 [ Links ]
28. Lippolis R, Gnoni A, Abbrescia A, Panelli D, Maiorano S, Paternoster MS, Sardanelli AM, et al. 2011. Comparative proteomic analysis of four Bacillus clausii strains: proteomic expression signature distinguishes protein profile of the strains. J Proteomics 74: 2846-2855. doi: 10.1016/j.jprot.2011.-06.032 [ Links ]
29. Midhun SJ, Neethu S, Vysakh A, Sunil MA, Radhakrishnan EK, Jyothis M. 2017. Antibacterial activity of autochthonous bacteria isolated from Anabas testudineus (Bloch, 1792) and it’s in vitro probiotic characterization. Microb Pathogenesis 113: 312-320. doi: 10.1016/j.micpath.2017.10.058
30. Moldenhauer J, Chen XH, Borriss R, Piel J. 2007. Biosynthesis of the antibiotic bacillaene, the product of a giant polyketide synthase complex of the trans-AT family. Angew Chem Int Edit 46: 8195-8197. doi: 10.1002/anie.200703386 [ Links ]
31. Mora I, Cabrefiga J, Montesinos E. 2011. Antimicrobial peptide genes in Bacillus strains from plant environments. Int Microbiol 14: 213-223. doi: 10.2436/20.1501.01.151 [ Links ]
32. Mukherjee A, Ghosh K. 2016. Antagonism against fish pathogens by cellular components and verification of probiotic properties in autochthonous bacteria isolated from the gut of an Indian major carp, Catla catla (Hamilton).Aquac Res 47: 2243-2255. doi: 10.1111/are.12676 [ Links ]
33. Nandi A, Banerjee G, Dan SK, Ghosh K, Ray AK. 2017. Probiotic efficiency of Bacillus sp in Labeo rohita challenged by Aeromonas hydrophila: assessment of stress profile, haematobiochemical parameters and immune responses. Aquac Res 48: 4334-4345. doi: 10.1111/are.13255 [ Links ]
34. Nandi A, Dan SK, Banerjee G, Ghosh P, Ghosh K, Ringø E, Ray AK. 2017. Probiotic potential of autochthonous bacteria isolated from the gastrointestinal tract of four freshwater teleosts. Probiotics Antimicro 9: 12-21. doi: 10.1007/s12602-016-9228-8 [ Links ]
35. Nayak SK. 2010. Role of gastrointestinal microbiota in fish. Aquac Res 41: 1553-1573. doi: 10.1111/j.13652109.2010.-02546.x [ Links ]
36. Ramarathnam R, Bo S, Chen Y, Fernando WG, Xuewen G, de Kievit T. 2007. Molecular and biochemical detection of fengycinand bacillomycin D-producing Bacillus spp, antagonistic to fungal pathogens of canola and wheat. Can J Microbiol 53: 901-911. doi: 10.1139/W07-049 [ Links ]
37. Ramesh D, Vinothkanna A, Rai AK, Vignesh VS. 2014. Isolation of potential probiotic Bacillus spp and assessment of their subcellular components to induce immune responses in Labeo rohita against Aeromonas hydrophila. Fish Shellfish Immunol 45: 268-276. doi: 10.1016/j.fsi.2015.04.018 [ Links ]
38. Ray AK, Ghosh K, Ringø E. 2012. Enzyme-producing bacteria isolated from fish gut: a review. Aquacult Nutr 18: 465492. doi: 10.1111/j.1365-2095.2012.00943.x [ Links ]
39. Ruiz L, Hidalgo C, Blanco-Míguez A, Lourenço A, Sánchez B, Margolles A. 2016. Tackling probiotic and gut microbiota functionality through proteomics. J Proteomics 147: 28-39. doi: 10.1016/j.jprot.2016.03.023 [ Links ]
40. Sajitha KL, Dev SA, Maria Florence EJ. 2016. Identification and characterization of lipopeptides from Bacillus subtilis b1 against sapstain fungus of rubberwood through MALDI-TOF-MS and RT-PCR. Curr Microbiol 73: 46-53. doi: 10.1007/s00284-016-1025-9 [ Links ]
41. Sánchez B, Arias S, Chaignepain S, Denayrolles M, Schmitter JM, Bressollier P, Urdaci MC. 2009. Identification of surface proteins involved in the adhesion of a probiotic Bacillus cereus strain to mucin and fibronectin. Microbiology 155: 1708-1716. doi: 10.1099/mic.0.025288-0 [ Links ]
42. Schlee M, Wehkamp J, Altenhoefer A, Oelschlaeger TA, Stange EF, Fellermann K. 2007. Induction of human β-defensin 2 by the probiotic Escherichia coli Nissle 1917 is mediated through flagellin. Infect Immun 75: 23992407. doi: 10.1128/IAI.01563-06 [ Links ]
43. Schmidt F, Fiege T, Hustoft HK, Kneist S, Thiede B. 2009. Shotgun mass mapping of Lactobacillus species and subspecies from caries related isolates by MALDI-MS. Proteomics 9: 1994- 2003. doi: 10.1002/pmic.200701028 [ Links ]
44. Shankar J, Walker RG, Ward D, Horsburgh MJ. 2012. The Enterococcus faecalis exoproteome: identification and temporal regulation by fsr. Plos One 7: e33450. doi: 10.1371/journal.pone.0033450 [ Links ]
45. Sharifuzzaman SM, Abbass A, Tinsley JW, Austin B. 2011. Subcellular components of probiotics Kocuria SM1 and Rhodococcus SM2 induce protective immunity in rainbow trout (Oncorhynchus mykiss, Walbaum) against Vibrio anguillarum. Fish Shellfish Immun 30: 347-353. doi: 10.1016/j.fsi.2010.11.005 [ Links ]
46. Shevchenko A, Tomas H, Havlis J, Olsen JV, Mann M. 2007. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nat Protoc 1: 2856-2860. doi: 10.1038/nprot.2006.468 [ Links ]
47. Telli GS, Ranzani-Paiva MJ, Dias Dde C, Sussel FR, Ishikawa CM, Tachibana L. 2014. Dietary administration of Bacillus subtilis on hematology and non-specific immunity of Nile tilapia Oreochromis niloticus raised at different stocking densities. Fish Shellfish Immun 39: 305-311. doi: 10.1016/j.fsi.2014.05.025 [ Links ]
48. Thanigaivel S, Vijayakumar S, Gopinath S, Mukherjee A, Chandrasekaran N, Thomas J. 2015. In vivo and in vitro antimicrobial activity of Azadirachta indica (Lin) against Citrobacter freundii isolated from naturally infected tilapia (Oreochromis mossambicus).Aquaculture 437: 252-255. doi: 10.1016/j.aquacul-ture.2014.-12.008 [ Links ]
49. Thankappan B, Ramesh D, Ramkumar S, Natarajaseenivasan K, Anbarasu K. 2015. Characterization of Bacillus spp from the gastrointestinal tract of Labeo rohita towards to identify novel probiotics against fish pathogens. Appl Biochem Biotech 175: 340-353. doi: 10.1007/s12010-014-1270-y [ Links ]
50. Vinusha KS, Deepika K, Johnson TS, Agrawal GK, Rakwal R. 2018. Proteomic studies on lactic acid bacteria: a review. Biochem Biophysics Reports 14: 140-148. doi: 10.1016/j.bbrep.2018.04.009 [ Links ]
51. Wang YB, Li JR, Lin J. 2008. Probiotics in aquaculture: challenges and outlook. Aquaculture 281: 1-4. doi: 10.1016/j.aquaculture.2008.06.002 [ Links ]
52. Wang Y, Osatomi K, Yoshida A, Liang X, Kanai K, Oda T, Hara K. 2010. Extracellular products from virulent strain of Edwardsiella tarda stimulate mouse macrophages (RAW264.7) to produce nitric oxide (NO) and tumor necrosis factor (TNF)-á. Fish Shellfish Immun 29: 778-785. doi: 10.1016/j.fsi.2010.07.014 [ Links ]
53. Wemhoff S, Meinhardt F. 2013. Generation of biologically contained, readily transformable, and genetically manageable mutants of the biotechnologically important Bacillus pumilus. Appl Microbiol Biot 97: 7805-7819. doi: 10.1007/s00253-013-4935-5 [ Links ]
54. Worden A. 2009. CTAB extraction buffer. Cold Spring Harbor Protocols. [Internet]. Available in: http://cshprotocols.cshlp.org/content/2009/10/pdb.rec11984 [ Links ]
55. Wu L, Wu H, Chen L, Xie S, Zang H, Borriss R, Gao X. 2014. Bacilysin from Bacillus amyloliquefaciens FZB42 has specific bactericidal activity against harmful algal bloom species. Appl Environ Microb 80: 7512-7520 doi: 10.1128/AEM.02605-14 [ Links ]
Recibido: 6 de noviembre de 2018
Aceptado para publicación: 2 de mayo de 2019