Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista de Investigaciones Veterinarias del Perú
Print version ISSN 1609-9117
Rev. investig. vet. Perú vol.30 no.3 Lima July/Set 2019
http://dx.doi.org/10.15381/rivep.v30i3.16598
ARTÍCULOS PRIMARIOS
Evaluación de modelos de predicción de composición química y energía bruta de kikuyo (Pennisetum clandestinum) usando espectroscopía en infrarrojo cercano (NIRS)
Combined treatment of chemotherapy and surgery in the transmissible venereal tumor in canines
Flor Mejía1,4, Ives Yoplac2, Wilmer Bernal2, Wilson Castro3
1 Escuela de Post Grado, Universidad Nacional Toribio Rodríguez de Mendoza de Amazonas, Chachapoyas, Perú
2 Facultad de Ingeniería Zootecnista, Agronegocios y Biotecnología, Universidad Nacional Toribio Rodríguez de Mendoza de Amazonas, Chachapoyas, Perú
3 Facultad de Ingeniería, Universidad Privada del Norte, Cajamarca, Perú
3 E-mail: flor.mejia@untrm.edu.pe
RESUMEN
El objetivo de este estudio fue evaluar modelos de predicción de la composición química del pasto kikuyo (Pennisetum clandestinum) usando espectroscopía en el infrarrojo cercano (NIRS). Se recolectaron muestras de P. clandestinum del distrito de Florida-Pomacochas, Amazonas, Perú, en tres edades de la planta (45, 60 y 75 días después del corte) y dos épocas del año (lluviosa y seca). Se determinó el contenido de humedad (H), proteína cruda (PC), extracto etéreo (EE), fibra cruda (FC), cenizas y energía bruta (EB). Se obtuvieron los espectros de absorbancia en el rango de longitud de onda de 1100-2500 nm. Mediante funciones y scripts de Matlab 2015ª se implementaron modelos de predicción, completos y optimizados, mediante redes neuronales (ANN) y regresión por mínimos cuadrados parciales (PLSR). Los modelos optimizados usaron 18 longitudes de onda relevantes, determinadas para ambos tipos de modelos de acuerdo a la matriz de beta coeficientes del modelo PLSR. Los modelos PLSR vs ANN, en la etapa de validación, mostraron mejores ajustes (R2>0.70) en H, PC, EE, cenizas y EB con un R2 de 0.74, 0,89, 0.79, 0.74 y 0.87, respectivamente. Por tanto, el modelo NIRS-PLSR tiene potencial en la predicción de la composición del pasto kikuyo (P. clandestinum).
Palabras clave: análisis químico; NIRS; PLSR; ANN; validación; Pennisetum clandestinum
ABSTRACT
The aim of this study was to evaluate predictive models of the chemical composition of kikuyo grass (Pennisetum clandestinum) using near infrared spectroscopy (NIRS). Samples of P. clandestinum were collected from the district of Florida-Pomacochas, Amazonas, Peru, in three stages of the plant (45, 60 and 75 days after cutting) and two seasons (rainy and dry). The moisture content (H), crude protein (PC), ether extract (EE), crude fibre (FC), ash and gross energy (EB) were determined. The absorbance spectra were obtained in the wavelength range of 1100-2500 nm. Through Matlab 2015ª functions and scripts, complete and optimized prediction models were implemented using neural networks (ANN) and regression by partial least squares (PLSR). The optimized models used 18 relevant wavelengths, determined for both types of models according to the matrix of beta coefficients of the PLSR model. The models PLSR vs ANN, in the validation stage, showed better fit (R2>0.70) in H, PC, EE, ash and EB with an R2 of 0.74, 0.89, 0.79, 0.74 and 0.87, respectively. Therefore, the NIRS-PLSR model has potential in the prediction of the composition of the kikuyo grass (P. clandestinum).
Key words: chemical analysis; NIRS; PLSR; ANN; validation; Pennisetum clandestinum
INTRODUCCIÓN
El análisis químico de los forrajes permite conocer su composición nutricional, facilitando la formulación de raciones que cubran los requerimientos energéticos y nutricionales de los animales (Molano et al., 2016). Sin embargo; el alto costo de los reactivos químicos y su escasa disponibilidad en la región Amazonas (Asekova et al., 2016) resalta la importancia de contar con tecnologías económicas para ampliar el uso de estos análisis (Oliva et al., 2015).
Una tecnología emergente, con aplicación en el análisis de la composición química y nutricional es la espectroscopía en el infrarrojo cercano (Near-Infrared Spectroscopy, NIRS). Esta se ha usado con relativo éxito en la determinación de la composición nutricional de forrajes (Valenciaga et al., 2006; Sandoval et al., 2008; Asekova et al., 2016; Bezada et al., 2017). Las principales ventajas del NIRS son su bajo coste, reducido tiempo de análisis y carácter no destructivo (Cen y He, 2007; Zhang y Su, 2014); no obstante, la técnica requiere generar mode los de calibración para relacionar los perfiles espectrales NIR y valores de composición obtenidos por métodos tradicionales, como el método proximal (Dykes et al., 2014, Guindo et al., 2016; Rushing et al., 2016; Gatius et al., 2017).
Por tanto, debido a que no se cuenta con modelos de predicción de la composición en pastos de la región Amazonas se planteó como objetivo principal de este trabajo evaluar modelos de predicción de la composición química del pasto kikuyo (Pennisetum clandestinum) usando NIRS.
MATERIALES Y MÉTODOS
Lugar de Estudio y Muestras
Las muestras se recolectaron en el distrito de Florida-Pomacochas, provincia de Bongará, Amazonas y los análisis se realizaron en el Laboratorio de Nutrición Animal y Bromatología de Alimentos de la Universidad Nacional Toribio Rodríguez de Mendoza de Amazonas (Chachapoyas, Perú).
Se trabajó con 42 muestras (30 para calibración y 12 para validación externa) de P. clandestinum recogidas de dos épocas del año: lluviosa (marzo-junio) y seca (agostooctubre), en tres edades de la planta (45, 60 y 75 días después del corte). Las muestras fueron secadas en una estufa a 60 °C por 48 horas y, posteriormente, molidas y envasadas en bolsa de polietileno de alta densidad.
Análisis Químico de Referencia
El análisis químico se realizó según el protocolo de la AOAC (1990) para las variables nutricionales proteína cruda (AOAC 976.05), extracto etéreo (AOAC 920.39), fi bra cruda (AOAC 978.10), cenizas totales (AOAC 942.05) y humedad (AOAC 925.45A, AOAC, 1995). En el caso de energía bruta se utilizó la metodología de calorimetría.
Obtención de Perfiles Espectrales
Los perfiles espectrales se obtuvieron mediante la metodología descrita por Asekova et al. (2016) y Molano et al. (2016), con las siguientes modificaciones: el escaneo de los espectros de absorbancia NIRS (SpectraStar 2500XL, EEUU), con lámpara halógena de tungsteno como fuente de luz y detector InGaAs (indio – galio – arsénico) se hicieron en el rango de longitud de onda 1100-2500 nm, y los espectros de muestra cada se tomaron por quintuplicado.
Modelado
Para modelar las relaciones entre perfiles espectrales y parámetros nutricionales se usaron funciones implementadas en los scripts de Matlab 2015ª. Asimismo, la regresión por mínimos cuadrados parciales (Partial Least Squares Regression, PLSR) y las redes neuronales (Artificial Neural Networks, ANN) hicieron uso de los toolboxs de Matlab.
PLSR
PLSR es uno de los métodos más utilizados y se ha aplicado para predecir propiedades de los forrajes (Ibáñez y Alomar, 2008; Andueza et al., 2011; Shetty et al., 2012; Reddersen et al., 2013; Bezada et al., 2017). En esencia, utiliza los valores de absorbancia dentro de un rango de longitud de onda determinado, extrae las características del espectro y luego establece la correlación entre las mediciones instrumentales y los valores de la propiedad de interés (Wold et al., 2001; Zhang et al., 2017).
Para desarrollar y validar el modelo PLSR se realizaron modelos completos con t od as l a s lo ngi tu de s d e onda de c ada parámetro y optimizados con las longitudes de onda relevantes de cada uno de los parámetros mediante el procedimiento descrito por Vásquez et al. (2017).
ANN
Las ANN son técnicas de aprendizaje supervisadas que se utilizan para predicción y clasificación. Los modelos ANN consisten en una capa de entrada, una o más capas ocultas y una capa de salida (Afandi et al., 2016). La capa de entrada recibe los valores de entrada y, por medio de una función de transferencia, distribuye los valores de atributo de entrada en las siguientes capas. En las capas ocultas, los datos se calculan utilizando funciones de transferencia sigmoidal no lineal. En la capa de salida, la cantidad de elementos de procesamiento depende de las categorías de clasificación (Vásquez et al., 2017; Zeng et al., 2017), y el número de neuronas en la capa de entrada y en la capa de salida corresponden a las longitudes de onda y los parámetros que se modelan (Vásquez et al., 2017).
Se construyeron los modelos ANN completos y optimizados en forma análoga al modelo PLSR. El modelo optimizado usó las mismas longitudes de onda seleccionadas para el modelo PLSR.
Validación
Para la validación del modelo se siguió el procedimiento propuesto por Vásquez et al. (2017). Un conjunto de datos, denominado de validación, de 12 muestras se usaron para determinar el error entre los valores reales de cada parámetro nutricional y los predichos usando los modelos.
RESULTADOS Y DISCUSIÓN
Análisis Químico de Referencia
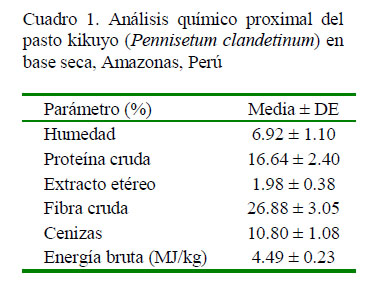
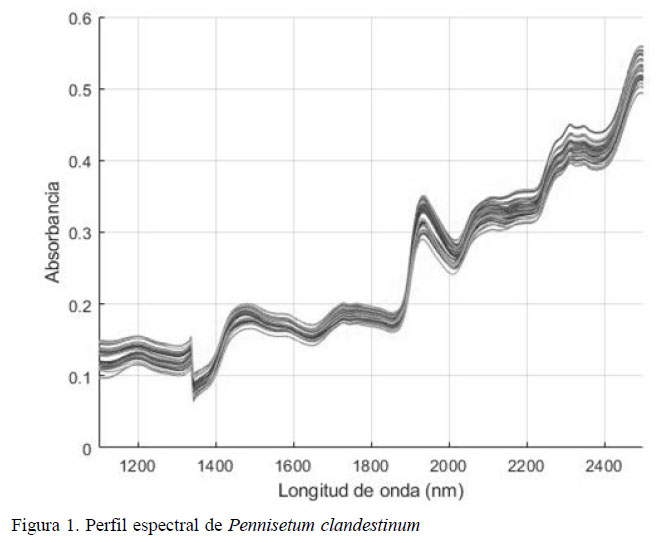
Los resultados del análisis químico proximal del pasto kikuyo se presentan en el Cuadro 1. Los valores fueron inferiores a los reportados por Soto et al. (2005), Aguilar et al. (2009) y Jaimes et al. (2015) para proteína cruda (19.04, 20.6 y 17.8%, respectivamente) y extracto etéreo (4.05, 2.26 y 2.10%; respectivamente). Así mismo, el porcentaje de cenizas en este estudio fue superior al 9.53 y 10.0% encontrados por Soto et al. (2005) y Jaimes et al. (2015), respectivamente. El perfil espectral se encuentra en el rango de longitud de onda de 1100-2500 nm (Figura 1).
Longitudes de Ondas Relevantes
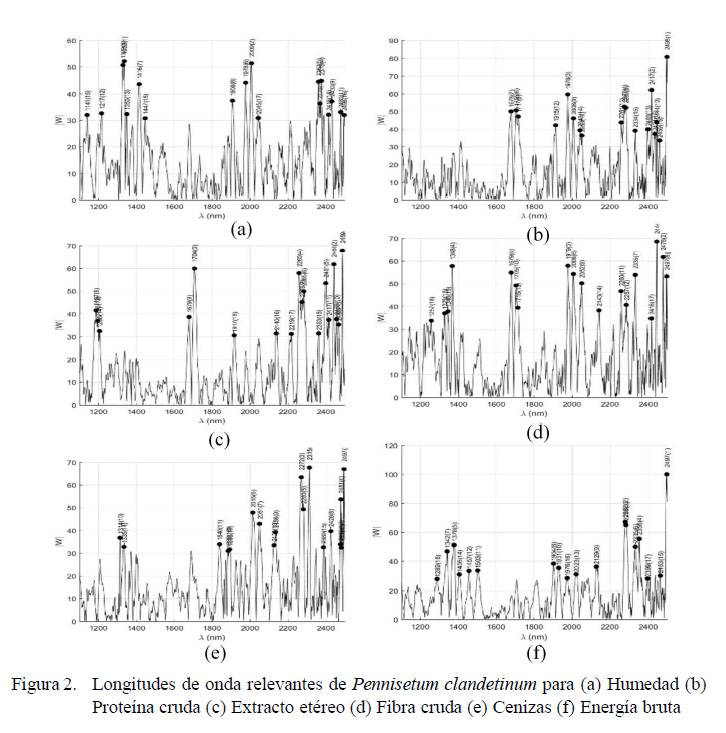
Las longitudes de onda relevantes para construir el modelo optimizado se distribuyeron a lo largo del rango evaluado (Figura 2). Se obtuvieron 18 longitudes de onda relevantes, estando para humedad en el rango de 1100-1400, 1600-2087 y 2170-2489 nm, para proteína de 1678-2496 nm, para extracto etéreo de 1187-1266, 1679, 1708 y 1917-2489 nm, para fibra cruda de 1257-1396, 1666-1730, 1880-2143 y 2238-2497 nm, para cenizas de 1211-1240, 1840-2136 y 2272-2497 nm y para energía bruta de 1289-1503, 1904-2129 y 2262-2497 nm. Los resultados concuerdan con Osborne (2006) y Sandoval et al. (2008), quienes indican que cada longitud de onda tiene una diferente banda de absorción el cual indica un nutriente diferente en cada longitud de onda.
Modelado y Validado con PLSR y ANN
El efecto del número de variables latentes explicado en el porcentaje de Y y el error cuadrático medio (RMSE) fue evalua do para determinar el número óptimo de variables latentes en el modelo PLSR, los cual nos determina la cantidad de longitudes de onda relevantes a utilizar.
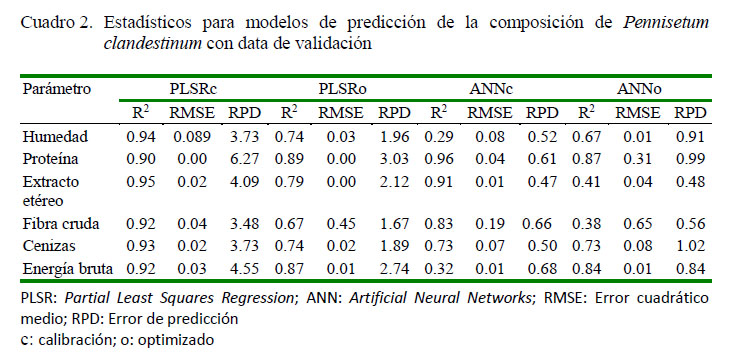
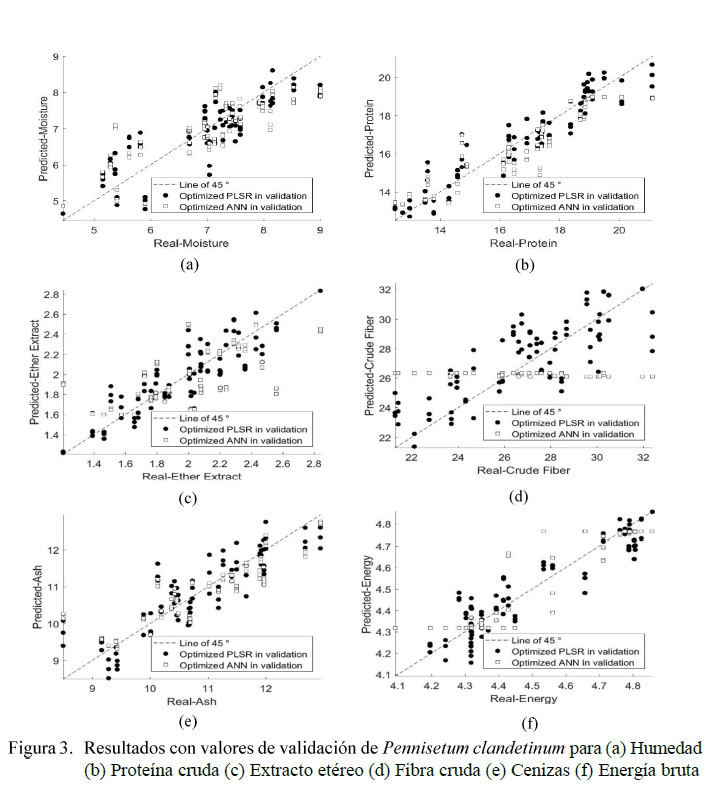
Se generaron dos modelos de predicción, los cuales fueron validados y los resultados se muestran en el Cuadro 2 y Figura 3. El modelo PLSR produjo resultados aceptables con altos valores de R2 y el modelo ANN presentó valores bajos no aceptables, a excepción de PC y EB. En PC reporto un R2 superior a 0.90 y para los demás parámetros nutricionales evaluados se obtuvo un R2 inferior a 0.90, pero mayor que 0.70, lo cual indica un R2 aceptable en PLSR.
Estos resultados fueron similares a los obtenidos por Bezada et al. (2017), quienes estudiaron el rye-grass italiano mediante el modelo de mínimos cuadrados parciales (PLSR) usando NIRS en el rango de longitud de onda de 400-2500 nm, obteniendo un R2 de modelado en PC, EE, cenizas y FC de 0.94, 0.71, 0.74 y 0.71 respectivamente. De igual manera, Asekova et al. (2016) determinaron la calidad de forraje de soya mediante NIRS, usando el método mínimos cuadrados parciales modificados (MPLS) en el rango de longitud de onda de 400-2500 nm obteniendo un R2 de 0.91 para PC y 0.93 EE. Asimismo, Zamudio (2016) evaluó la composición química del rye-grass mediante el uso de NIRS con el modelo PLSR , donde deter minaron el R2 para PC, FC, EE y cenizas de 0.96, 0.90, 0.83 y 0.95, respectivamente. Las diferencias entre estudios se podrían deber a los equipos utilizados para la predicción y al número de muestras utilizadas.
CONCLUSIONES
-
Los modelos NIRS-ANN mostraron valores de ajuste inferiores a los modelos NIRS-PLSR en la predicción de la composición del pasto kikuyo.
-
El modelo NIRS-PLSR es capaz de realizar la predicción de humedad, proteína cruda, extracto etéreo, cenizas y energía bruta con valores de ajuste (R2) superiores al 0.74 y mínima correlación de R2 de 0.55 para fibra.
Agradecimientos
Al Programa de Becas de CONCYTEC-CIENCIACTIVA y a la Universidad Nacional Rodríguez de Mendoza de Amazonas por el financiamiento de la investigación.
Literatura citada
1 . Afandi SD, Herdiyeni Y, Prasetyo LB, Hasbi W, Arai K, Okumura, H. 2016. Nitrogen content estimation of rice crop based on near infrared (NIR) reflectance using artificial neural network (ANN). Procedia Environ Sci 33: 63-69. doi: 10.1016/j.proenv.2016.03.057 [ Links ]
2 . Aguilar OX, Moreno BM, Pabón ML, Carulla JE. 2009. Effect of kikuyo (Pennisetum clandestinum) or ryegrass (Lolium hibridum) intake on conjugated linoleic acid concentration and fatty acids composition of milk fat. Livestock Res Rural Develop 21(4). [Internet]. Disponible en: http://www.lrrd.org/lrrd21/4/agui21049.htm [ Links ]
3 . Andueza D, Picard F, Jestin M, Andrieu J, Baumont R. 2011. NIRS prediction of the feed value of temperate forages: efficacy of four calibration strategies. Animal 5: 1002-1013. doi: 10.1017/S1751731110002697 [ Links ]
4 . [AOAC] Association of Official Analytical Chemists. 1990. Official Methods of Analysis. 15th ed. Vol 1. Washington, USA. [ Links ]
5 . [AOAC] Association of Official Analytical Chemists. 1995. Official Methods of Analysis. 16th ed. Vol 2. Washington, USA. [ Links ]
6 . Asekova S, Han SI, Choi HJ, Park SJ, Shin DH, Kwon CH, Lee JD. 2016. Determination of forage quality by near-infrared reflectance spectroscopy in soybean. Turk J Agric For 40: 45-52. doi: 10.3906/tar-1407-33 [ Links ]
7 . Bezada S, Arbaiza T, Carcelén F, San Martín F, López C, Rojas J, Vélez V. 2017. Prediction of chemical composition and neutral detergent fibre of Italian ryegrass (Lolium multiflorum Lam) by near infrared spectroscopy (NIRS). Rev Inv Vet Perú 28: 538-548. doi: 10.15381/rivep.v28i3.13357 [ Links ]
8 . Cen H, He Y. 2007. Theory and application of near infrared reflectance spectroscopy in determination of food quality. Trends Food Sci Tech 18: 72-83. doi: 10.1016/j.tifs.2006.09.003 [ Links ]
9 . Dykes L, Hoffmann L, Portillo Rodriguez O, Rooney WL, Rooney L. W. 2014. Prediction of total phenols, condensed tannins, and 3-deoxyanthocyanidins in sorghum grain using nearinfrared (NIR) spectroscopy. J Cereal Sci 60: 138-142. doi : 10.1016/j.jcs.2014.02.002 [ Links ]
10. Gatius F, Miralbés C, David C, Puy J. 2017. Comparison of CCA and PLS to explore and model NIR data. Chemometr Intell Lab 164: 76-82. doi: 10.1016/j.chemolab.2017.03.011 [ Links ]
11. Guindo D, Davrieux F, TemeN , Vaksmann M, Bastianelli D, Verdeil J, Rami J. 2016. Pericarp thickness of sorghum whole grain is accurately predicted by NIRS and can affect the prediction of other grainquality parameters. J Dairy Sci 69: 218-227. doi: 10.1016/j.jcs.2016.03.008 [ Links ]
12. Ibáñez L, Alomar D. 2008. Prediction of the chemical composition and fermentation parameters of pasture silage by near infrared reflectance spectroscopy (NIRS). Chil J Agr Res 68: 352-359. doi: 10.4067/S0718-58392008000400005 [ Links ]
13. Jaimes LJ, Cerón JM, Correa HJ. 2015. Efecto de la época del año y la etapa de lactancia sobre el consumo alimenticio de vacas Holstein pastoreando kikuyo (Cenchrus clandestinus) en Colombia. Livestock Res Rural Develop 27(12). [Internet]. Disponible en: http://www.lrrd.org/lrrd27/12/jaim27244.html [ Links ]
14. Mol ano ML, Cortés ML, Ávila P, Martens SD, Muñoz S. 201 6. Ecuaciones de calibración en espectroscopía de reflectancia en el infrarrojo cercano (NIRS) para predicción de parámetros nutritivos en forrajes tropicales. Forrajes Tropicales 4: 139-145. doi: 10.17138/TGFT(4)139-145 [ Links ]
15. Oliva M, Rojas D, Morales A, Oliva C, Oliva M. 2015. Contenido nutricional, digestibilidad y rendimiento de biomasa de pastos nativos que predominan en las cuencas ganaderas de Molinopampa, Pomacochas y Leymebamba, Amazonas, Perú. Scientia Agropec 6: 211-215. doi: 10.17268/sci.agropecu.2015.03.07 [ Links ]
16. Osborne BG. 2006. Near-infrared spect roscopy in food analysis. In: Encyclopedia of analytical chemistry. John Wiley & Sons. p 1-14. doi: 10.1002/9780470027318.a1018 [ Links ]
17. Reddersen B, Fricke T, Wachendorf M. 2013. Effects of sample preparation and measurement standardization on the NIRS calibration quality of nitrogen, ash and NDFom content in extensive experimental grassland biomass. Anim Feed Sci Tech 183: 77-85. doi: 10.1016/j.anifeedsci.2013.04.016 [ Links ]
18. Rushing JB, Saha UK, Lem us R, Sonon L, Baldwin BS. 2016. Analysis of some important for age quality attributes of sou theast ern wil drye (El ymus glabriflorus) usingne arinfrared reflectance spectroscopy. Am J Analytical Chem 7: 642-662. doi: 10.4236/ajac.2016.79060 [ Links ]
19. Sandoval LA, Bueso FJ, Vélez M. 2008. Predicción nutricional para pastos tropicales por espectroscopía de reflectancia en el infrarrojo cercano. Agronomía Mesoamericana 19: 221-225. doi: 10.15517/AM.V19I2.5003 [ Links ]
20. Shetty N, Gislum R, Mette A, Jensen D, Boelt B. 2012. Development of NIR calibra-tion models to assess year-toyear variation in total non-structural carbohydrates in grasses using PLSR. Chemometr Intell Lab 111: 34-38. doi: 10.1016/j.chemolab.2011.11.004 [ Links ]
21. Soto C, Valencia A, Galvis RD, Correa HJ. 2005. Efecto de la edad de corte y del nivel de fertilización nitrogenada sobre el valor energético y proteico del pasto kikuyo (Pennisetum clandestinum). Rev Colomb Cienc Pec 18: 17-26. [ Links ]
22. Valenciaga D, Oliveira E, Saliba S. 2006. Near infrared reflectance spectroscopy (NIRS) and its potentials for forage evaluation. Cuban J Agr Sci 40: 245-252. [ Links ]
23. Vásquez N, Magan C , Oblitas J , Chuquizuta T, Castro W. 2017. Comparison between artificial neural net work and partialleast squares regression models for hardness modelling during the ripening process of Swiss-type cheese using spectral profiles. J Food Eng 219: 8-15 . doi: 10.1016/j.jfoodeng.2017.09.008 [ Links ]
24. Wold S, Sjostrom M, Eriksson L. 2001. PLS-regression: a basic tool of chemometrics. Chemometr Intell Lab 58: 109-130. doi: 10.1016/S0169-7439(01)-00155-1 [ Links ]
25. Zamudio BS. 2016. Predicción de la composición química de rye grass mediante el uso de espectroscopia de reflectancia en el infrarrojo cercano (NIRS). Tesis de Médico Veterinario. Lima, Perú: Univ. Nacional Mayor de San Marcos. 53 p. [ Links ]
26. Zeng W, Xu C, Zhao G, Wu J, Huang J. 2017. Estimation of sunflower seed yield using partial least squares regression and artificial neural network models. Pedosphere 28: 764-774. doi: 10.1016/S1002-0160(17)60336-9 [ Links ]
27. Zhang C, Su J. 2014. Application of near infrared spectroscopy to the analysis and fast quality assessment of traditional Chinese medicinal products. Acta Pharmaceutica Sinica B 4: 182-192. doi: 10.1016/j.apsb.2014.04.001 [ Links ]
28. Zhang H, Li Z, Chen T, Qin B. 2017. Quantitative determination of auramine obytera hertz spectros copy with 2DCOS-PLSR model. Spectrochim Acta A 184: 335-341. doi: 10.1016/j.saa.2017.05.017 [ Links ]
Recibido: 21 de agosto de 2018
Aceptado para publicación: 15 de abril de 2019