Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista de Investigaciones Veterinarias del Perú
versão impressa ISSN 1609-9117
Rev. investig. vet. Perú vol.30 no.4 Lima out./dez. 2019
http://dx.doi.org/10.15381/rivep.v30i4.14733
Artículos primarios
Diversidad genética en ovinos (Ovis aries) Asblack de Lima, Perú, utilizando marcadores microsatélites
Genetic diversity in Asblack sheep (Ovis aries) of Lima, Peru by microsatellite markers
J.A. Morón1,3, C. Yalta2, G. Gutiérrez1, E. Veli2
1 Departamento de Producción Animal, Facultad de Zootecnia, Universidad Nacional Agraria La Molina, Lima, Perú
2 Laboratorio de Biología Molecular y Genómica, Dirección de Recursos Genéticos y Biotecnología, Instituto Nacional de Innovación Agraria, Lima, Perú.
3 E-mail: jmoron@lamolina.edu.pe
RESUMEN
El ovino Asblack es el resultado del cruce de los ovinos Assaf y Blackbelly con fines de formar una raza sintética. El objetivo del estudio fue analizar la diversidad genética de los ovinos Asblack y su relación con los ovinos Assaf y Blackbelly. Se colectaron muestras de sangre a 103 ovinos no emparentados de la región Lima, Perú. Los ovinos fueron genotipados para 17 marcadores microsatélites. Se halló una alta diversidad en las poblaciones, identificándose un total de 146 alelos, donde la población de ovinos Asblack tuvo el mayor número de alelos. La heterocigosidad esperada fue mayor a la observada en los ovinos Asblack con respecto al ovino Assaf y Blackbelly, lo que indica una tendencia al déficit de heterocigotos en el cruce. Además, se observó nueve locus que no están en equilibrio de Hardy-Weinberg, bajo la hipótesis nula de unión aleatoria de gametos en el cruce Asblack. La variación molecular entre las poblaciones fue 11.4% y la variación entre individuos dentro de las poblaciones fue de 6.8%; sin embargo, al analizar la estructura poblacional, el flujo genético fue de 0.03, lo que indica una diferenciación genética. Los valores de FIS (0.077) reflejaron bajo niveles de endogamia y el FST (0,115) indicó una moderada diferenciación genética entre las poblaciones. Las poblaciones se separaron en tres grupos (K=3), resultado soportado por el análisis factorial de correspondencia. En conclusión, se evidencia una alta diversidad genética en las poblaciones estudiadas por raza e individuos, con una moderada diferenciación de ovinos Asblack con respecto a sus razas progenitoras.
Palabra clave: ovinos; microsatélites; diversidad genética; estructura poblacional
ABSTRACT
The Asblack sheep is the cross of Assaf and Blackbelly to obtain a composite breed. The aim of this study was to determine the genetic diversity of the Asblack and its relationship with the parental breeds. Blood samples were taken from 103 unrelated sheep from the Lima, Peru region. Sheep samples were genotyped for 17 microsatellite markers. High diversity was found in the populations and a total of 146 alleles were detected. Asblack had the highest number of alleles. The expected heterozygosity was greater than the observed in Asblack sheep with regards to Assaf and Blackbelly which indicates a trend for a deficit of heterozygotes in the cross. In addition, nine loci were not in HardyWeinberg equilibrium, under the null hypothesis of random union of gametes in the cross. The molecular variation among the populations was 11.4% and the variation among individuals within the populations was 6.81% of the total; however, when analyzing the population structure, the genetic flow was 0.03, which indicates a lower genetic differentiation. The values of FIS (0.077) reflected low levels of inbreeding, and FST (0.115) indicates a moderate genetic differentiation. On the other hand, the populations were separated in the three clusters (K=3), a result supported by the factorial correspondence analysis. In conclusion, a high genetic diversity was found in the populations studied by breed and individuals, with a moderate differentiation of Asblack sheep with its parental breeds.
Key words: sheep; microsatellites; genetic diversity; population structure
INTRODUCCIÓN
Ovinos Assaf y Blackbelly, procedentes de Israel y la Isla de Barbados, respectivamente, fueron introducidos al Perú entre los años 80 y 90. El cruzamiento de estas razas tenía como objetivo la formación de una raza rústica denominada Asblack, capaz de ser utilizada en la producción intensiva para la obtención de carne de cordero, con un grado de sangre que se buscaba estabilizar en ¼ Blackbelly y ¾ Assaf. Este cruce busca aprovechar las cualidades genéticas de la raza Assaf (mayor peso corporal y mayor producción de leche) y de la raza Blackbelly (prolificidad) (Aliaga, 2006).
A partir de 1991, el Ing. Rigoberto Calle comenzó a hacer apareamientos en la Granja de ovinos «Rigoranch» (Cieneguilla, Perú) con el objetivo de formar una nueva raza ovina (cruzamiento de ovejas Blackbelly con carneros Assaf) denominada «Asblack», con la finalidad de reunir la habilidad materna, producción de leche y conformación cárnica del ovino Assaf con la prolificidad del ovino Blackbelly (BB). El resultado sería un ovino capaz de ser utilizado en la producción intensiva de carne de cordero, tratando de estabilizar el cruce en ¼ BB y ¾ Assaf (Aliaga, 2006).
Llaxacóndor (1995) reportó que el Asblack, en sus diferentes grados de cruzamiento, estaba siendo usado en el Programa de Mejoramiento Genético de ovinos criollos de la zona de Cori (Provincia de Aija, Ancash, Perú), con la finalidad de fusionar la precocidad, prolificidad y producción lechera de los Asblack con la rusticidad de los criollos. Así mismo, señala que el ovino Asblack había demostrado una notoria adaptación a la zona de Coris.
Los ovinos Asblack han sido poco estudiados desde el punto de vista genético, motivo por el cual, no se le puede considerar como una raza. Actualmente, el uso de las herramientas moleculares permite analizar estructuras genéticas de las poblaciones y empleando análisis matemáticos y estadísticos se logra examinar procesos y patrones evolutivos que pueden haber influido, o aún seguir influyendo, en la estructura y distribución de estas especies (Toro et al., 2009).
Una de las herramientas moleculares utilizadas a nivel mundial en especies animales son los microsatélites. Diversos autores reportan el uso de marcadores microsatélites en ovinos locales y adaptados para estimar la estructura y diversidad genética (Neubauer et al., 2015; Salamon et al., 2014; Yilmaz et al., 2014; Ghazy et al., 2013; Pablo et al., 2013; Bauer et al., 2010, Steliana et al., 2009; Blackburn et al., 2011). El presente estudio tuvo como objetivo determinar la diversidad genética de los ovinos Asblack y su relación con los ovinos Assaf y Blackbelly usando marcadores microsatélites en la Región de Lima, Perú. Los resultados de este estudio permitirán establecer al ovino Asblack como una raza oriunda del país, permitiendo elaborar criterios de mejora genética (ovinos con dos crías mínimo y corderos con mayor peso) para mejorar la rentabilidad y facilidad de manejo de los animales.
MATERIALES Y MÉTODOS
Muestras y Extracción de ADN
Se recolectaron muestras de sangre en tubos de 10 ml que contenían 500 µl de EDTA a partir de la vena yugular de ovinos no emparentados y de mayor edad. Las muestras se colectaron de 47 ovinos Asblack y 26 de ovinos Assaf de la Universidad Nacional Agraria La Molina Granja «Rigoranch», y de 30 ovinos Blackbelly del fundo «El Cortijo» en Chilca, Lima (Perú). Las muestras fueron llevadas para su procesamiento al Laboratorio de Biología Molecular y Genómica del Instituto Nacional de Innovación Agraria (INIA), Lima.
El proceso de extracción de ADN se realizó utilizando el método de Sambrook y Russel (2001) con algunas modificaciones: a) aislamiento de glóbulos blancos mediante lavados repetitivos con fuffer Tris EDTA, b) lisis de glóbulos blancos con detergente SDS, enzima proteinasa K y acetato de K, c) extracción orgánica mediante dos lavados con solventes orgánicos (cloroformo: alcohol isoamílico), d) precipitación y lavado del ADN a partir de NaCl y etanol absoluto y, e) resuspensión del ADN con Buffer TE.
Amplificación de Microsatélites
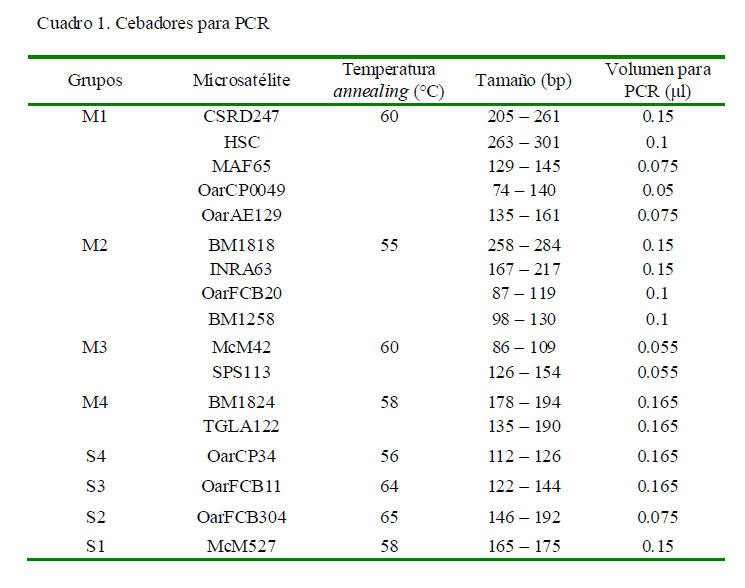
Se seleccionaron 17 marcadores microsatélites fluoromarcados con FAM, HEX y NED descritos en estudios previos (Georges y Massey, 1992; Montgomery et al., 1993; Bishop et al., 1994; Hulme et al., 1994; Ede y Crawford, 1995; de Gortari et al., 1998; Di Stasio, 2001; Alfonso et al., 2006). Para ello, el volumen de cada marcador en la amplificación fue estandarizado y agrupado para ser utilizados en sistemas multiplex (es decir, que en una misma reacción se amplifican varios marcadores, de acuerdo con su tamaño de pares de bases y a su temperatura de anillamiento) y en sistemas simplex (Cuadro 1).
Las condiciones de PCR fueron las siguientes: 95 ºC por 5 min, seguida de 30-40 ciclos a 95 °C por 45 s, 55 °C y 65 °C por 60 s y 72 °C por 1 min; extensión final a 72 °C por 15 min. Los productos amplificados de PCR fueron separados mediante electroforesis capilar utilizando un analizador genético ABI 3130XL (Applied Biosystems).
La asignación de alelos se realizó con el programa Genemapper 4.0 (Applied Biosystems). El software visualiza los fragmentos en forma de picos que se miden en unidades de fluorescencia. Luego se formó una base de datos con los genotipos de cada una de las muestras.
Análisis de Datos
Para el análisis de la diversidad genética se utilizó el programa GENEPOP v. 4.1 (Rousset, 2008) y Cervus v.3.0 (Kalinowski, et al., 2007). Además se realizó un análisis de varianza molecular (AMOVA) con el programa Arlequin v. 3.1 (Excoffier et al., 2006). El flujo genético en los ovinos estudiados se determinó mediante el modelo de alelos privados de Barton y Slatkin (1986) con el programa Genepop v. 4.1 (Rousset, 2008). Los índices de fijación (FIS, FST, FIT) fueron calculados usando el programa FSTAT v. 2.9.3 (Goudet, 2002).
Finalmente, para el análisis de la estructura de la población se realizó un análisis factorial de correspondencia, el cual describe la asociación de variables cualitativas, en la que cada individuo está representado solo una vez por el valor de cada modalidad (locus) y variable (alelos para cada locus) con el programa Genetix v. 4.05 (Belkhir et al., 2003) y el programa Structure v. 2.3.2.1 (Pritchard et al., 2000), asumiendo un rango de k = 1 al k = 3.
RESULTADOS
Diversidad Genética
Diversidad genética por marcador
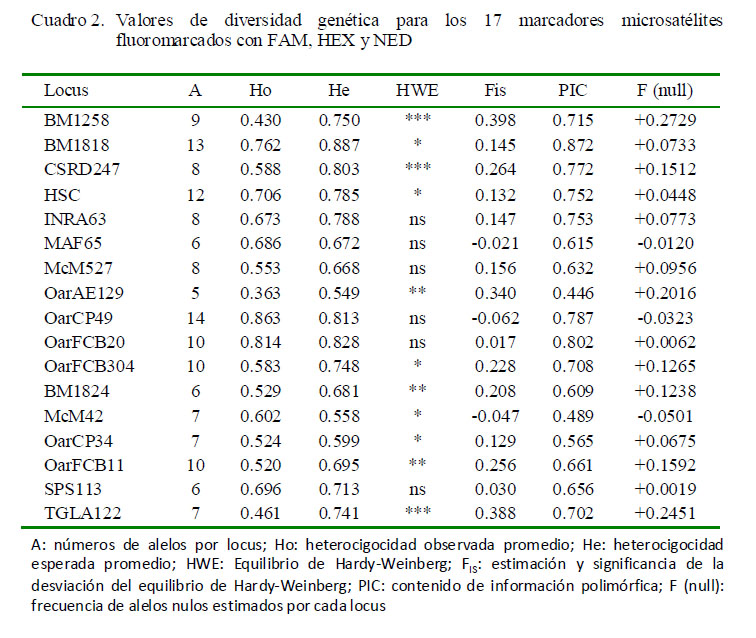
Se identificaron 146 alelos en los 17 microsatélites (Cuadro 2). El mayor número de alelos se encontró en el locus OarCP49 (n=14), mientras que el menor número de alelos se encontró en el locus OarAE129 (n=5). Los valores del contenido de información polimórfica (PIC) fueron altamente polimórficos en 14 microsatélites, siendo el locus BM1818 quien presentó un PIC de 0.872, mientras que en el locus OarAE129 se reportó un bajo polimorfismo (PIC: 0.446).
Los valores de heterocigosidad esperada (HE) fueron más elevados que la observada (HO) en 14 microsatélites. Los locus BM1818, BM1824 y SPS113 presentaron valores de HE mayor que la HO en las tres poblaciones, indicando que las poblaciones de ovinos son estables genéticamente, por lo cual, tienen una alta diversidad genética (Cuadro 2). Las diferencias de heterocigosidad evidencian desviaciones del equilibrio de HardyWeinberg (p<0.001), que se atribuyen al exceso de homocigotos; encontrando 11 marcadores que no se encuentran en equilibrio de Hardy-Weinberg.
Diversidad genética por población
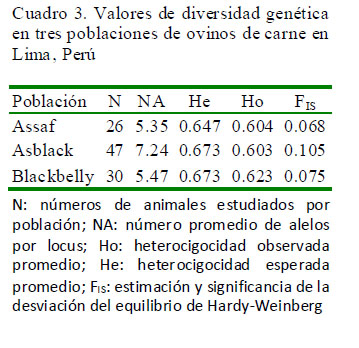
Los ovinos Asblack presentaron un promedio de alelos mayor con respecto a la de sus progenitoras. Asimismo, se identificaron 11 alelos exclusivos entre las poblaciones estudiadas, con un rango de frecuencias de 0.05,0.15%; de los cuales el alelo 150 (OarAE129) fue reportado para los ovinos Asblack; los alelos 90 (OarFCB20), 120 (BM1258), 161 (TGLA122), 220 (CSRD247), 272 (BM1818) y los alelos 265, 285, 289, 291 >(HSC) para los ovinos Blackbelly, y el alelo 106 (OarFCB20) para los ovinos Assaf.
Los ovinos Asblack presentan un déficit de heterocigotos a diferencia de sus progenitoras quienes presentaron un exceso de heterocigotos. Además, se encontraron cuatro marcadores en los ovinos Assaf (BM1258, OarFCB304, OarFCB11, TGLA122) que no están en equilibrio de Hardy-Weinberg. Así mismo, cuatro marcadores (BM1258, BM1818, HSC, BM1824) para los ovinos Blackbelly y ocho marcadores para los ovinos Asblack (BM1258, CSRD247, HSC, OarAE129, McM42, OarCP34, SPS113, TGLA122) que no están en equilibrio de Hardy-Weinberg debido a un déficit de heterocigotos. En consecuencia, se aprecia un coeficiente de consanguinidad (FIS=0.105) ligeramente alto para la población Asblack con respecto a sus progenitores quienes presentaron un valor positivo (Cuadro 3).
También se puede observar una alta diversidad genética en las poblaciones estudiadas, por la alta variación molecular entre individuos (81.74%); mientras que la variación (6.81%) entre individuos dentro de cada población fue bajo a consecuencia de un déficit de heterocigotos y, la variación reportada entre las poblaciones es de 11.46%.
Diferenciación Genética
Para las tres poblaciones de ovinos se encontró una frecuencia media de alelos privados de 0.031 y un número de migrantes por generación igual a 0.03, el cual es un valor muy bajo e indica que por cada 100 generaciones migran 3 individuos. De esta forma se puede enfatizar que existe una diferenciación genética entre las poblaciones.
El valor obtenido de FIS fue 0.08 mostrando un déficit de heterocigotos. La baja reducción en la heterocigosidad individual indica un apareamiento controlado. El valor obtenido del FIT fue de 0.18 y el valor obtenido de FST fue 0.11, lo cual indica una moderada diferenciación entre las poblaciones (Cornell University, 2004); es decir, aún hay cercanía entre las poblaciones de Assaf y Asblack a causa de cruzamiento entre estas poblaciones.
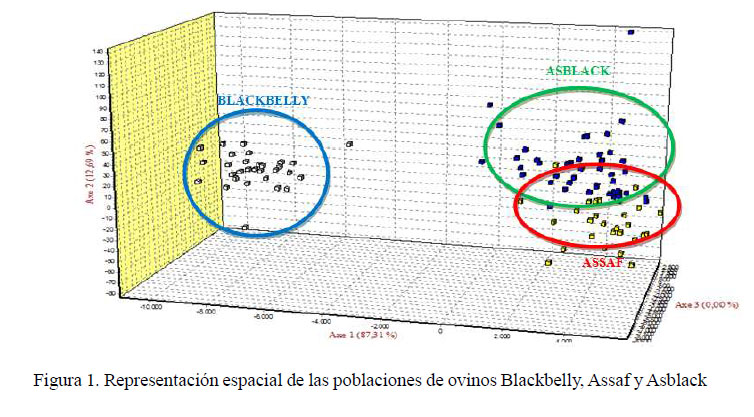
El grado de diferenciación en la estructura genética que existe entre individuos de la misma población y las tres poblaciones en estudio es concordante al Análisis Factorial de Correspondencia (Figura 1), donde el primer componente está explicando el 87.3% de la variación total, separando moderadamente las poblaciones de Assaf y Asblack. El segundo componente representa el 12.7% del total de la variación y separa la población Blackbelly.
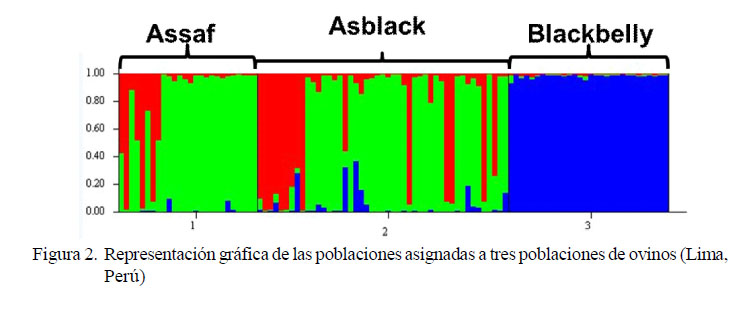
Las relaciones genéticas entre las poblaciones de ovinos evaluadas sobre la base de 17 marcadores microsatélites demostraron que las poblaciones se diferencian genéticamente. En función de los resultados (K=3), se agrupan las poblaciones en dos grandes grupos (Figura 2).
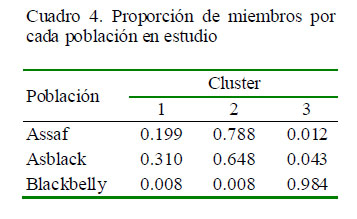
En el Cuadro 4 se observa la proporción de miembros para cada grupo identificado, utilizando 100000 repeticiones y 500 Burn. El análisis asignó a la población Blackbelly a un grupo independiente (cluster 3 por ser la más diferenciada. El cluster 2 tuvo mayor proporción de miembros de la población Assaf con una influencia similar de la población Asblack. Por último, en el cluster 1 se observa la población Asblack con una ligera influencia de los ovinos Assaf.
DISCUSIÓN
La granja Rigoranch a través de un programa de mejora genética tuvo como finalidad la formación de ovinos Asblack a partir de ovinos Assaf y Blackbelly, orientada a un incremento en la producción de carne de cordero (Aliaga, 2006). Este cruce genera una reducción del tamaño de la población produciendo la disminución del número de alelos, especialmente la pérdida de aquellos que se encuentran en frecuencias muy bajas debido al efecto de la deriva génica. Sin embargo, el número promedio de alelos en los ovinos Asblack es mayor con relación a sus progenitores como consecuencia de alelos exclusivos debido al efecto de una posible mutación.
Los ovinos Blackbelly fueron estudiados por Paiva et al. (2011) utilizando cinco (INRA63, MAF65, OarAE129, OarFCB20, OarFCB304) de los 17 microsatélites mencionados en este trabajo, obteniendo un número de alelos promedio (n=4.45) inferior a la reportada en este trabajo (n=5.47), pero con una heterocigosidad observada superior a la esperada. Por otro lado, los ovinos Assaf fueron analizados por 14 microsatélites obteniendo un número de alelos promedio de 7 y una heterocigosidad esperada de 0.670 (Legaz et al., 2008); sin embargo, solo el locus BM1818 fue utilizado en ambos trabajos, obteniéndose en el presente estudio un menor número de alelos promedio (n=5.47) y una heterocigosidad esperada de 0.651.
Al comparar cada una de las poblaciones por marcador, el contenido de información polimórfica resultó altamente significativo en todos los valores de probabilidades; es decir, que estos loci son muy buenos para diferenciar genéticamente las poblaciones entre sí; sin embargo, dos microsatélites presentaron valores que no eran significativos para la diferenciación genética, y esto podría ser a un sesgo en los resultados. Asimismo, se observa un déficit de heterocigotos, sobre todo en ovinos Asblack, como consecuencia de su manejo por un proceso de selección; donde se han fijado alelos en las tres poblaciones, probablemente debido a una deriva génica.
El coeficiente de endogamia obtenido para los ovinos progenitores (Assaf y Blackbelly) fueron bajas por ser razas establecidas y estar en manejo productivo y reproductivo constante. Por otro lado, la población Asblack presentó una endogamia ligeramente alta (FIS=0.105), aunque lo esperado era obtener una endogamia elevada como consecuencia de los cruces entre sus progenitores durante el proceso de formación de este grupo genético y el manejo que se viene realizando con los ovinos.
Los ovinos Asblack de la granja Rigoranch evaluados en trabajos anteriores presentan características productivas y reproductivas similares (Aliaga, 2006), siendo este un rebaño homogéneo. No obstante, a nivel molecular (Fst=0.11) se aprecian diferencias moderadas en la estructura genética entre las tres poblaciones, lo que indica que hay una migración recíproca entre poblaciones (Chikhi et al., 2004). También se puede observar que las tres poblaciones tienen estructuras similares, donde la mayor variabilidad se da a nivel intra-poblacional (81.7%) que a nivel inter-poblacional (11.5%).
Asimismo, para estabilizar y generar una raza de ovino Asblack se buscaba el grado de sangre ¾ Assaf y ¼ Blackbelly (Aliaga, 2006); pero al observar la representación espacial de las poblaciones por el análisis factorial por correspondencia se aprecia que la población de ovinos Asblack está más cercano a la proporción de la población de ovinos Assaf, lo cual indica que el grado de sangre no es necesariamente la mencionada. Esto se confirma al evaluar la representación gráfica de las poblaciones con el programa Structure, donde se encuentra la población de ovinos Blackbelly bien diferenciada del resto y una moderada diferencia entre las otras dos poblaciones (Assaf y Asblack) como consecuencia de apareamientos aleatorios generados entre estas poblaciones.
CONCLUSIONES
-
Las poblaciones de ovinos Blackbelly, Assaf y Asblack presentaron una alta diversidad genética al obtener una heterocigosidad observada superior a la esperada, indicando un exceso de heterocigotos.
-
Se observa la presencia de un alelo exclusivo en su población como consecuencia de un proceso de selección, pudiendo haber ocurrido una mutación dando origen a nuevos alelos.
-
El grado de diferenciación genética entre las poblaciones estudiadas fue moderada.
Agradecimientos
Los autores agradecen al fundo «El Cortijo» y la granja «Rigoranch» por facilitar el manejo de las poblaciones de ovinos. También agradecen al Instituto Nacional de Innovación Agraria (INIA) y al Programa de Ovinos y Camélidos Americanos UNALM por el apoyo financiero en la presente investigación.
LITERATURA CITADA
Alfonso L, Parada A, Legarra A, Ugarte E, Arana A. 2006. The effects of selective breeding against scrapie susceptibility on the genetic variability of the Latxa Black-Faced sheep breed. Genet Sel Evol 38: 495-511. doi: 10.1051/gse:2006017 [ Links ]
Aliaga J. 2006. Producción de ovinos. Lima, Perú; Juan Gutemberg. 299 p. [ Links ]
Barton NH, Slatkin M. 1986. A quasiequilibrium theory of the distribution of rare alleles in a subdivided population. Heredity 56: 409-415. doi: 10.1038/hdy.1986.63 [ Links ]
Bauer M, Margetin M, Bauerova M, Oravcova M, Peškovièová D. 2010. Microsatellite analysis of valachian sheep population in Slovakia using 16-plex PCR. Acta Fitotec Zootec 2010: 55-58.
Belkhir K, Borsa L, Chikhi N, Bonhomme F. 2003. Genetix: 4.05 Logiciel sous WindowsTM pour la genetique des populations. Laboratoire Genoma Populations, Interactions, Adaptations. Montpellier. France. [ Links ]
Bishop MD, Kappes SM, Keele JW, Stone RT, Sunden SL, Hawkins GA, Toldo SS, et al. 1994. A genetic linkage map for cattle. Genetics 136: 619-639. [ Links ]
Blackburn H, Paiva S, Wildeus S, Getz W, Waldron D, Stobart R, Bixby D, et al. 2011. Genetic structure and diversity among sheep breeds in the United States: identification of the major gene pools. J Anim Sci 89: 2336-2348. doi: 10.2527/jas.2010-3354 [ Links ]
Chikhi L, Goossens B, Treanor A, Bruford MW. 2004. Population genetic, structure of and inbreeding in an insular cattle breed, the jersey, and its implications for genetic resource management. Heredity 92: 396-401. doi: 10.1038/sj.hdy.6800433 [ Links ]
Cornell University, IPGRI. 2004. Medidas de la diversidad genética. [Internet]. Disponible en: https://www.bioversityinternational. org/fileadmin/user_upload/online_library/publications/pdfs/Molecula_Markers_Volume_2_es.pdf [ Links ]
de Gortari MJ, Freking BA, Cuthbertson RP, Kappes SM, Keele JW, Stone RT, et al. 1998. A secondgeneration linkage map of the sheep genome. Mamm Genome 9: 204-209. doi: 10.1007/s003359900726 [ Links ]
Di Stasio L. 2001. ISAG Standing Committee on «Applied genetics in sheep and goats. Panels of markers for parentage verification tested at the 2001/ 02 ISAG comparison test». [Internet]. Available in: https://www.isag.us/Docs/consignmentforms/2005_PanelsMarkersSheepGoats.pdf [ Links ]
Ede A, Crawford A. 1995. Mutations in the sequence flanking the microsatellite at the KAP8 locus prevent the amplification of some alleles. Anim Genet 26: 43-44. doi: 10.1111/j.13652052.1995.tb02619.x [ Links ]
Excoffier L, Laval G, Schneider S. 2006. An integrated software package for population genetics data analysis (Arlequin v.3.1). University of Berne, Switzerland. [Internet]. Available in: http://cmpg.unibe.ch/software/arlequin3/ [ Links ]
Georges M, Massey J. 1992. Polymorphic DNA markers in Bovidae. Patent WO 92/13102. [Internet]. Available in: https://patents.google.com/patent/WO1992013102A1/en [ Links ]
Ghazy A, Mokhtar S, Eid M, Amin A, Elzarei M, Kizaki K, Hashizume K. 2013. Genetic diversity and distance of three Egyptian local sheep breeds using microsatellite markers. Res Zool 3: 1-9. 10.5923/j.zoology.20130301.01 [ Links ]
Goudet J. 2002. FSTAT, a program to estimate and test gene diversities and fixation indices (version 2.9.3). University of Lausanne. [Internet]. Available in: https://www2.unil.ch/popgen/softwares/fstat.htm [ Links ]
Hulme DJ, Silk JP, Redwin JM, Barendse W, Beh KJ. 1994. Ten polymorphic ovine microsatellites. Anim Genet 25: 434-435. doi: 10.1111/j.13652052.1994.tb00543.x [ Links ]
Kalinowski ST, Taper ML, Marshall TC. 2007. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Mol Ecol 16: 1099-1106. doi: 10.1111/j.1365-294X.2007.-03089.x [ Links ]
Legaz E, Álvarez I, Royo LJ, Fernández I, Gutiérrez JP, Goyache F. 2008. Genetic relationships between Spanish Assaf (Assaf.E) and Spanish native dairy sheep breeds. Small Ruminant Res 80: 3944. doi: 10.1016/j.smallrumres.2008.09.001 [ Links ]
Montgomery GW, Crawford AM, Penty JM, Dodds KG, Ede AJ, Henry HM, Pierson CA, et al. 1993. The ovine Booroola fecundity gene (FecB) is linked to markers from a region of human chromosome 4q. Nat Genet 4: 410 414. doi: 10.1038/ng0893-410 [ Links ]
Neubauer V, Vogl C, Seregi J, Sáfár L, Brem G. 2015. Genetic diversity and population structure of Zackel sheep and other Hungarian sheep breeds. Arch Anim Breed 58: 343-350. doi: 10.5194/aab-58343-2015 [ Links ]
Pablo M, Landi V, Martínez A, Lara C, Delgado JV. 2013. Caracterización genética de la oveja lojeña mediante marcadores microsatélites. Actas Iberoam Conserv Anim 3: 194-200. [ Links ]
Paiva S, Mariante A, Blackburn H. 2011. Combining US and Brazilian microsatellites data for a meta-analysis of sheep (Ovis aries) breed diversity: facilitating the FAO global plan of action for conserving animal genetic resources. J Hered 102: 697-704. doi: 10.1093/jhered/esr101 [ Links ]
Pritchard JK, Stephens M, Donnelly P. 2000. Inference of population structure using multilocus genotype data. Genetics 155: 945-959. [ Links ]
Rousset F. 2008. Genepop´007: a complete reimplementation of the Genepop software for windows and linux. Mol Ecol Res 8: 103-106. doi: 10.1111/j.14718286.2007.01931.x [ Links ]
Salamon D, Gutierrez-Gil B, Arranz JJ, Barreta J, Batinic V, Dzidic A. 2014. Genetic diversity and differentiation of 12 eastern Adriatic and western Dinaric native sheep breeds using microsatellites. Animal 8: 200-207. doi: 10.1017/S1751731113002243 [ Links ]
Sambrook J, Russell D. 2001. Cold Spring Harbor Laboratory Press. Vol. 1: 2344. [ Links ]
Steliana E, Georgescu S, Manea M, Zaulet M, Hermenean A, Costache M. 2009. Genetic diversity using microsatellite markers in four Romanian autochthonous sheep breeds. Rom Biotech Lett 15: 5059-5065. [ Links ]
Toro MA, Fernández J, Caballero A. 2009. Molecular characterization of breeds and its use in conservation. Livest Sci 120: 174-195. doi: 10.1016/j.livsci.2008.07.003 [ Links ]
Yilmaz O, Cemal I, Karaca O. 2014. Genetic diversity in nine native Turkish sheep breeds based on microsatellite analysis. Anim Genet 45: 604-608. doi: 10.1111/age.12173 [ Links ]
Recibido: 15 de junio de 2018
Aceptado para publicación: 15 de agosto de 2019