Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.30 no.4 Lima oct./dic. 2019
http://dx.doi.org/10.15381/rivep.v30i4.17148
Artículos primarios
Genotipificación por RAPD-PCR de aislados de Yersinia ruckeri procedentes de truchas arcoíris (Oncorhynchus mykiss) de la sierra del Perú
Genotyping by RAPD-PCR of Yersinia ruckeri isolates from rainbow trout (Oncorhynchus mykiss) of the highlands of Peru
Fernando Mesías V.1, Enrique Serrano-Martínez1,2, Luis Llanco A.1
1 Grupo SANIVET, Facultad de Medicina Veterinaria y Zootecnia, Universidad Peruana Cayetano Heredia, Lima, Perú
2 E-mail: enrique.serrano@upch.pe
RESUMEN
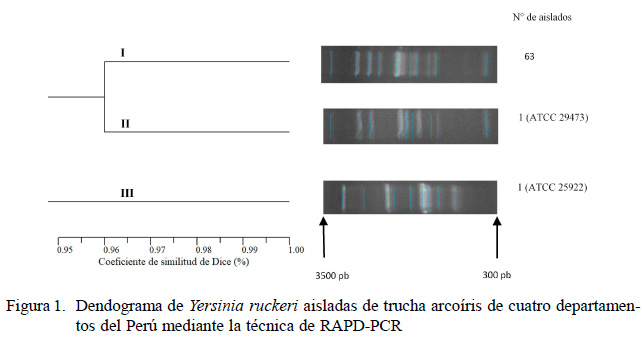
El objetivo del presente estudio fue identificar genotipos de Y. ruckeri, según su procedencia, mediante RAPD-PCR. Se tomaron muestras asépticas de bazo y riñón de truchas arcoíris (Oncorhynchus mykiss) con signos clínicos de yersiniosis, provenientes de piscigranjas de cuatro departamentos (Junín, Lima, Ancash y Puno) del Perú. Las cepas fueron cultivadas en agar tripticasa de soya a 25 °C por 24 horas y la identificación presuntiva de las colonias sospechosas de corresponder a Y. ruckeri se hizo mediante tinción Gram y pruebas bioquímicas de oxidasa y catalasa. Luego, se confirmó la identidad de Y. ruckeri por PCR. Se obtuvieron 63 aislados de Y. ruckeri y se analizaron mediante RAPD-PCR. El dendograma de diversidad genética agrupó a todos los aislados en un clúster, estrechamente relacionadas con la cepa control Y. ruckeri ATCC 29473 (96% de similitud). También se observó que todos los aislados eran genéticamente idénticos (clones). Se sugiere que las cepas podrían tener un origen común debido a que tuvieron diferentes procedencias geográficas y elevada similitud entre ellos.
Palabras clave: sanidad acuícola; RAPD-PCR; yersiniosis; aislamiento; Yersinia ruckeri
ABSTRACT
The aim of this study was to identify Y. ruckeri genotypes, according to their origin, by RAPD-PCR. Aseptic samples of spleen and kidney were taken from rainbow trout (Oncorhynchus mykiss) with clinical signs of yersiniosis in fish farms of four departments (Junín, Lima, Ancash and Puno) of Peru. The strains were cultivated on trypticase soy agar at 25 °C for 24 hours and the presumptive identification of the colonies suspected of being Y. ruckeri was made by Gram stain and biochemical tests of oxidase and catalase. Then, the identity of Y. ruckeri was confirmed by PCR. In total, 63 isolates of Y. ruckeri were obtained and analyzed by RAPD-PCR. The dendogram of genetic diversity grouped all the isolates in one cluster, closely related to the control strain Y. ruckeri ATCC 29473 (96% similarity). It was also observed that all the isolates were genetically identical (clones). It is suggested that the strains could have a common origin because they had different geographical origins and high similarity between them.
Key words: fish health; RAPD-PCR; yersiniosis; isolation; Yersinia ruckeri
INTRODUCCIÓN
La enfermedad de la boca roja que afecta a los salmónidos, principalmente a la trucha arcoíris, es producida por Yersinia ruckeri. Esta bacteria gramnegativa causa infecciones sistémicas de curso agudo a crónico y actualmente tiene una distribución mundial (AustinB yAustin D,2016).La enfermedad se caracteriza por ocasionar altas mortalidades y pérdidas económicas en las piscigranjas, junto con la presencia de hemorragias alrededor de la boca y el ano, base de las aletas y en la superficie de los órganos internos de los peces (Barnes, 2011; Kumar et al., 2015). Fue aislada por primera vez en una piscigranja de los EEUU a comienzos de la década de 1950 (Rucker, 1966). El primer reporte en Perú de este agente infeccioso fue en Junín (Bravo y Kojagura, 2004) y, posteriormente, en otras regiones como Lima, Huancavelica y Áncash (Sirvas et al., 2011; Flores, 2013).
Si bien Y. ruckeri ha sido descrita como una especie altamente homogénea respecto de sus características fenotípicas y bioquímicas (Altun et al., 2013; Sierralta et al., 2013; Calvez et al., 2014); el desarrollo de métodos de tipificación molecular ha permitido profundizar los conocimientos sobre la variabilidad genética de este patógeno. Entre estos métodos se incluyen la ribotipificación (García et al., 1998), ERIC-PCR y REPPCR (Bastardo et al., 2012a; Duman et al., 2017), electroforesis en gel de campo pulsado (PFGE) (Huang et al., 2013) y tipificación de secuencias en múltiples locus (MLST) (Bastardo et al., 2012b; Calvez et al., 2015). Hay estudios que describen la diversidad genética de Y. ruckeri como elevada (Bastardo et al., 2012b; Duman et al., 2017), o estrechamente relacionada (Huang et al., 2013; Calvez et al., 2015), independiente del origen geográfico, hospedero y la elección del marcador para evaluar su variabilidad. Entre otros métodos de tipificación molecular basado en PCR, el ADN polimórfico amplificado al azar (RAPD-PCR) (Williams et al., 1990), ha sido utilizado con éxito para estudiar la diversidad genética microbiana con fines epidemiológicos, así como establecer asociaciones con características fenotípicas como virulencia y resistencia a antimicrobianos, entre otros. Lamentablemente, y a pesar de la importancia de Y. ruckeri para la acuicultura nacional, son pocos los estudios que han intentado caracterizar molecular-mente esta bacteria. Es así que el objetivo de este estudio fue caracterizar la variabilidad genotípica de aislados de Y. ruckeri de truchas arcoíris procedentes de cuatro departamentos del Perú mediante RAPD-PCR (Random Amplification of Polymorphic DNA, por sus siglas en inglés).
MATERIALES Y MÉTODOS
El estudio fue de tipo observacional, analítico y transversal. El aislamiento e identificación bioquímica, así como la caracterización molecular y análisis genético de Y. ruckeri se realizó en el Laboratorio de Parasitología de la Facultad de Medicina Veterinaria y Zootecnia (FAVEZ) de la Universidad Peruana Cayetano Heredia (UPCH), Lima, Perú. El estudio fue aprobado por el comité de ética de la FAVEZ-UPCH.
Se muestrearon 240 truchas arcoíris (30 peces por piscigranja) con signos clínicos de yersiniosis, colectadas entre febrero y agosto de 2018, procedentes de ocho piscigranjas de Junín, Ancash, Lima y Puno, ubicadas entre los 3200 y 4400 msnm. Los peces fueron llevados en cajas de tecnopor hasta el laboratorio dentro de bolsas plásticas con agua y oxígeno, en relación de 2 a 1, y cubiertas con gel packs para mantener la temperatura durante el transporte.
Los peces se anestesiaron mediante sobredosis de eugenol (3 ml/l de agua) y se procedió con la eutanasia por corte medular (CCAC, 2010), para realizar la necropsia, según la metodología descrita por Meyers (2009). Se tomaron muestras de bazo y riñón anterior de cada individuo, la cuales se sembraron por estriado en placas con agar tripticasa de soya (TSA) (Liofilchem, Italia) e incubados a 25 °C por 24 h. La identificación presuntiva de las colonias sospechosas de ser Y. ruckeri fue realizada mediante tinción de Gram (bacilos gramnegativos) y las pruebas bioquímicas de oxidasa y catalasa. A continuación, se realizó PCR convencional para confirmar la identidad de Y. ruckeri. Para esto, se extrajo el ADN bacteriano de colonias crecidas en caldo infusión cerebro corazón (BHI), utilizando un kit comercial (DNeasyBlood &Tissuekit, QIAGEN,Alemania). Los cebadores, para detectar el gen rRNA 16S, y las condiciones de la reacción de PCR fueron realizadas según el protocolo de Gibello et al. (1999). El producto fue analizado en gel de agarosa con TBE al 1%, corrido a 90v durante 1 h, teñido con bromuro de etidio (1 µg/ml) y digitalizado. La cepa ATTC 29473 de Y. ruckeri fue utilizada como control positivo y agua libre de DNAsas como control negativo. El 100 bp DNA ladder (Invitrogen, Brasil) fue utilizado como marcador de peso molecular. El resultado positivo se dio por la observación de un amplicón de 575 pares de bases (pb).
Para la tipificación molecular por RAPD-PCR se utilizaron los iniciadores arbitrarios: OPA 3, 9, 12, 17 y 18. En un termociclador (Eppendorf, Mastercycler), en volumen final de 20 µl se adicionó 50 ng de ADN bacteriano, 10 pmol del cebador, 2 mM de cada desoxinucleósido trifosfato (dNTP), 5 µl de buffer de Taq polimerasa y 5 U/µl de Taq polimerasa. Se utilizaron como control externo el ADN de E. coli ATCC 25922 y como control interno el de Y. ruckeri ATCC 29473. La reacción de PCR se realizó siguiendo un paso inicial de desnaturalización (94 °C durante 5 min), luego 35 ciclos de desnaturalización (94 °C durante 1 min), hibridación (42 °C durante 2 min) y extensión (72 °C durante 2 min), seguidos por una extensión final a 72 °C durante 10 min. Los productos amplificados se separaron electroforéticamente a 80 v durante 90 min en geles de agarosa al 1.2%, con solución amortiguadora TAE 1X. Los geles fueron teñidos con solución de bromuro de etidio (1 µg/ml) durante 15 min y posteriormente visualizados con luz UV.
Los patrones de bandas generados por RAPD-PCR fueron inspeccionados de manera visual y se generó una matriz binaria (presencia/ausencia). El análisis se realizó con base al coeficiente de similitud de Dice y el dendograma se generó con el software NTSYSpc v. 2.21, utilizando el método de agrupación UPMGA (unweighted pair group method with arithmetic mean, por sus siglas en inglés) (Applied Biostatistics, USA).
RESULTADOS Y DISCUSIÓN
Se obtuvieron 63 aislados de Y. ruckeri de 43 truchas, siendo 432 de bazo y 31 del riñón anterior, los cuales fueron confirmados molecularmente mediante PCR convencional al obtener una banda única del tamaño esperado. En el Perú se han reportado varios brotes de yersiniosis en piscigranjas de Lima, Áncash, Huancavelica y Puno (Sirvas et al., 2011; Flores; 2013; Sandoval et al., 2016), y más recientemente en Pasco, Cajamarca y Amazonas (Cerro et al., 2017; Castro et al., 2017). En el presente estudio, se aisló Y. ruckeri de bazo y riñón de truchas arcoíris son signos clínicos de enfermedad, procedentes de Puno, Áncash, Junín y Lima. Esto demostraría la persistencia de esta bacteria en centros productivos de truchas arcoíris.
De los cinco cebadores probados, solo OPA-18, generó perfiles RAPD en los 63 aislados de Y. ruckeri y en la cepa tipo de Y. ruckeri ATCC 29473. Se generaron 11 bandas en el rango de 300-3500 pb (Figura 1). El dendograma agrupó a las Y. ruckeri en un eran genéticamente idénticos y ligeramente distintos de la cepa tipo de Y. ruckeri ATCC 29473 (similitud de 96%) y muy distantes de la cepa de E. coli ATCC 25922 (similitud de 33%, no mostrada). Este resultado es similar al de investigaciones realizadas por Onuk et al. (2011) y Altun et al. (2013), quienes al analizar 97 y 15 aislados de Y. ruckeri, respectivamente, usando RAPD-PCR, encontraron pocos, pero homogéneos grupos clonales.
En otros estudios, donde se utilizó la técnica de RAPD-PCR, pero con otras especies del género Yersinia, se encontró tanto una baja (Leal et al., 1999;Akhila et al., 2013; Shanmugapriya et al., 2014) como alta homogeneidad entre los diversos aislados (Franzin y Cabodi, 2003; Soto et al., 2013). La mayoría de estos estudios recomiendan el uso de RAPD-PCR sobre otros métodos de tipificación, debido a su rapidez, simplicidad y confiabilidad al utilizar reactivos estandarizados.
Los 63 aislados de Y. ruckeri se agruparon en un solo genogrupo, resultado similar al de Bastardo et al. (2011b), quienes mediante el uso de REP-PCR y ERIC-PCR, obtuvieron un solo clúster al analizar 30 aislados de Y. ruckeri procedentes de Junín. En otro estudio similar con 11 aislados de Y. ruckeri procedentes del salmón del atlántico (Salmo salar L.) se obtuvieron dos y tres genogrupos mediante ERIC-PCR y REPPCR, respectivamente; encontrándose una homogeneidad mayor del 85% en estos grupos clonales (Bastardo et al., 2011a). Adicionalmente, Duman et al. (2017) estudiaron la relación genética mediante ERICPCR de 141 aislados de Y. ruckeri, hallando la presencia de cuatro genogrupos (72% similitud), donde el 82% de estos aislados mostraron un patrón similar (clones).
La baja diversidad genética y gran homogeneidad intraespecífica encontrada por Huang et al. (2013) y Calvez et al. (2015) entre los aislados de Y. ruckeri podría haberse debido a que se originaron de un mismo ancestro en común, indicando una estructura de población clonal, producto de la influencia del origen geográfico, diferentes presiones de selección, nichos ecológicos (hospederos) específicos y movimientos de los peces de cultivo, desempeñando todos un papel importante en la difusión y evolución de Y. ruckeri. Por otro lado, Bastardo et al. (2012b) y Gulla et al. (2018), analizaron aislados de Y. ruckeri de diversas partes del mundo, incluyendo el Perú, mediante tipificación multilocus de secuencias (MLST) y análisis multi-locus de número variable de repeticiones en tándem (MLVA), encontrando elevada diversidad genética entre los diferentes complejos clonales, asociándolos al serotipo, biotipo, hospedero, origen geográfico, tipo de ambiente y cantidad de aislados. Los autores sugieren que los aislados procedentes de Perú se originaron de América del Norte, los cuales podrían haberse diseminado por causas antropogénicas al no ser la trucha arcoíris una especie nativa de nuestro país.
CONCLUSIONES
-
Se identifica a Yersinia ruckeri como el microrganismo causante de brotes de enfermedad en piscigranjas de la Sierra central y sur del Perú.
-
Mediante el análisis RAPD-PCR se determinó que los 63 aislados son genéticamente muy similares entre sí, sugiriéndose que estos pueden tener un mismo origen y que exista clonalidad.
Agradecimientos
Los autores agradecen a FONDECYTCONCYTEC por el apoyo financiero brindado al Programa de Maestría en Sanidad Acuícola de la UPCH (Convenio de Gestión N.° 230-2015 FONDECYT–De-PROMOCIÓN 2), que permitió los análisis y la capacitación técnica de los investigadores para la ejecución del estudio. Del mismo modo, se agradece a los piscicultores y tesistas que colaboraron durante la recolección de muestras.
LITERATURA CITADA
1. Akhila S, Shanmuga S, Senthil T, Thayumanavan T. 2013. Molecular diversity analysis of Yersinia enterocolitica isolated from marine marketed fish. Int J Curr Microbiol App Sci 2: 204-214. [ Links ]
2. Altun S, Onuk EE, Ciftci A, Duman M, Buyukekiz AG. 2013. Determination of phenotypic, serotypic and genetic diversity and antibiotyping of Yersinia ruckeri isolated from rainbow trout. Kafkas Univ Vet Fak 19: 225-232. doi: 10.9775/kvfd.2012.7606 [ Links ]
3. Austin B, Austin D. 2016. Bacterial fish pathogens: diseases of farmed and wild fish. 6th ed. Switzerland: Springer International. 366 p. [ Links ]
4. Barnes AC. 2011. Enteric red mouth disease (Yersinia ruckeri). In: Woo P, Bruno D (eds). Fish diseases and disorders. Vol 3: Viral, bacterial and fungal infections. UK: CABI. p 484-511. [ Links ]
5. Bastardo A, Bohle H, Ravelo C, Toranzo AE, Romalde JL. 2011a. Serological and molecular heterogenecity among Yersinia ruckeri strains isolated from farmed Atlantic salmon Salmo salar in Chile. Dis Aquat Organ 93: 207- 214. doi: 10.3354/dao02296 [ Links ]
6. Bastardo A, Sierralta V, León J, Ravelo C, Romalde JL. 2011b. Phenotypical and genetic characterization of Yersinia ruckeri strains isolated from recent outbreaks in farmed rainbow trout Oncorhynchus mykiss (Walbaum) in Peru. Aquaculture 317: 229-232. [ Links ]
7. Bastardo A, Ravelo C, Romalde JL. 2012a. A polyphasic approach to study the intraspecific diversity of Yersinia ruckeri strains isolated from recent outbreaks in salmonid culture. Vet Microbiol 160: 176-182. doi: 10.1016/ j.vetmic.2012.05.024 [ Links ]
8. Bastardo A, Ravelo C, Romalde JL. 2012b. Multilocus sequence typing reveals high genetic diversity and epidemic population structure for the fish pathogen Yersinia ruckeri. Environ Microbiol 14: 1888-1897. doi: 10.1111/ j.1462-2920.2012.02735.x [ Links ]
9. Bravo S, Kojagura V. 2004. First isolation of Yersinia ruckeri from rainbow trout (Oncorhynchus mykiss) in Peru. Bull Eur Assn Fish P 24: 104- 108. [ Links ]
10. Calvez S, Gantelet H, Blanc G, Douet DG, Daniel P. 2014. Yersinia ruckeri biotypes 1 and 2 in France: presence and antibiotic susceptibility. Dis Aquat Organ 109: 117-126. doi: 10.3354/dao02725 [ Links ]
11. Calvez S, Fournel C, Douet DG, Daniel P. 2015. Pulsed-field gel electrophoresis and multi locus sequence typing for characterizing genotype variability of Yersinia ruckeri isolated from farmed fish in France. Vet Res 46: 73. doi: 10.1186/s13567-015-0200-5 [ Links ]
12. Castro G, Ramos F, Uribe J, Manchego A, Sandoval N. 2017. Aislamiento de Yersinia ruckeri en truchas arcoiris (Oncorhynchus mykiss) provenientes de una piscigranja de la ciudad Chachapoyas, Amazonas. En: IV Conferencia Internacional CISIPA 2017. Lima: SANIPES-PRODUCE. [ Links ]
13. Cerro L, Manchego A, Ñuñure J, Pihue J, Sandoval N. 2017. Estudio de sensibilidad antibiótica de diferentes cepas de Yersinia ruckeri procedentes de diferentes regiones del país En: IV Conferencia Internacional CISIPA 2017. Lima: SANIPES-PRODUCE. [ Links ]
14. [CNC] Canadian Council on Animal Care. 2010. CCAC guidelines on euthanasia of animals used in science. Ottawa: CNC. 36 p. [ Links ]
15. Duman M, Altun S, Cengiz M, Saticioglu IB, Buyukekiz AG, Sahinturk P. 2017. Genotyping and antimicrobial resistance genes of Yersinia ruckeri isolates from rainbow trout farms. Dis Aquat Org 125: 31-44. doi: 10.3354/dao03132 [ Links ]
16. Flores K. 2013. Determinación de la diversidad fenotípica de Yersinia ruckeri en aislados de trucha arcoíris (Oncorhynchus mykiss) de cultivo de las regiones de Junín, Ancash y Huancavelica. Tesis de Médico Veterinario. Lima: Univ. Nacional. Mayor de San Marcos. 66 p. [ Links ]
17. Franzin L, Cabodi D. 2004. Molecular typing of Yersinia strains by pulsed-field gel electrophoresis and RAPD-PCR. In: Skurnik M, Bengoechea JA, Granfors K (eds). The genus Yersinia. Springer. p 349-352. [ Links ]
18. García JA, Dominguez L, Larsen JL, Pedersen K. 1998. Ribotyping and plasmid profiling of Yersinia ruckeri. J Appl Microbiol 85: 949-955. [ Links ]
19. Gibello A, Blanco MM, Moreno MA, Cutuli MT, Domenech A, Domínguez L, Fernández-Garayzábal JF. 1999. Development of a PCR assay for detection of Yersinia ruckeri in tissues of inoculated and naturally infected trout. Appl Environ Microb 65: 346-350. [ Links ]
20. Gulla S, Barnes AC, Welch TJ, Romalde JL, Ryder D, Ormsby MJ, Carson J, et al. 2018. Multilocus variable- number tandem-repeat analysis of Yersinia ruckeri confirms the existence of host specificity, geographic endemism, and anthropogenic dissemination of virulent clones. Appl Environ Microb 84: e00730-18. doi: 10.1128/AEM.00730-18 [ Links ]
21. Huang Y, Michael G, Becker R, Kaspar H, Mankertz J, Schwarz S, Runge M, Steinhagen D. 2013. Phenoand genotypic analysis of antimicrobial resistance properties of Yersinia ruckeri from fish. Vet Microbiol 171: 406-412. doi: 10.1016/j.vetmic.2013.10.026 [ Links ]
22. Kumar G, Menanteau-Ledouble S, Saleh M, El-Matbouli M. 2015. Yersinia ruckeri, the causative agent of enteric redmouth disease in fish. Vet Res 46: 2-6. doi: 10.1186/s13567-015-0238-4 [ Links ]
23. Leal T, Leal N, Paiva de Almeida A. 1999. RAPD-PCR typing of Yersinia enterocolitica (Enterobacteriaceae) O: 3 serotype strains isolated from pigs and humans. Genet Mol Biol 22: 315-319. doi: 10.1590/S1415-47571999000300005 [ Links ]
24. Onuk E, Alper C, Arzu F, Gülay C, Altun S, Balta F, Ozer S, Coban A. 2011. Phenotypic and molecular characterization of Yersinia ruckeri isolates from rainbow trout (Oncorhynchus mykiss, Walbaum, 1792) in Turkey. Berl Munch Tierarztl 124: 320-328. doi: 10.2376/0005-9366-124-320 [ Links ]
25. Meyers TR. 2009. Standard necropsy procedures for finfish. In: Heil N (ed). NWFHS Laboratory Procedures Manual. 5th ed. Washington: US Fish and Wildlife Service. p 64-74. [ Links ]
26. Rucker RR. 1966. Redmouth disease of rainbow trout (Salmo gairdneri). Bull Off Int Epizoot 65: 825-830. [ Links ]
27. Sandoval N, Llanco L, Manchego A, Mesías F. 2016. Determinación de Yersinia ruckeri y susceptibilidad antimicrobiana en alevines de truchas arcoíris cultivadas en el lago Titicaca-Puno. In: LACQUA - SARA 2016. Lima: World Aquaculture Society. [ Links ]
28. Shanmugapriya S, Senthilmurugan T, Thayumanavan T. 2014. Genetic diversity among Yersinia enterocolitica isolated from chicken and fish in and around Coimbatore City, India. Iran J Public Health 43: 835-844. [ Links ]
29. Sierralta V, León J, De Blas I, Bastardo A, Romalde J, Castro T, Mateo E. 2013. Patología e identificación de Yersinia ruckeri en trucha arcoíris (Oncorhynchus mykiss) en piscigranjas de Junín, Perú. AquaTIC 38: 28-45. [ Links ]
30. Sirvas S, Sánchez C, Peña C. 2011. Diagnóstico e identificación rápidos por PCR de Yersinia ruckeri aislada de Oncorhynchus mykiss procedentes de Canta, Lima, Perú. Rev Peru Biol 18: 349-353. doi: 10.15381/rpb.v18i3.451 [ Links ]
31. Soto E, Griffin M, Verma A, Castillo- Alcala F, Beierschmitt A, Beeler- Marfisi J, Arauz M, et al. 2013. An outbreak of Yersinia enterocolitica in a captive colony of African green monkeys (Chlorocebus aethiops sabaeus) in the Caribbean. Comparative Med 63: 439-444. [ Links ]
32. Williams JGK, Kubelik AR, Livak KJ, Rafalski JA, Tingey V. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res 18: 6531-6535. doi: 10.1093/nar/18.22.6531 [ Links ]
Recibido: 19 de noviembre de 2018
Aceptado para publicación: 31 de mayo de 2019