INTRODUCCIÓN
Las infecciones por Salmonella son consideradas como el principal problema sani- tario en sistemas de producción de cuyes (Ca- via porcellus), siendo el serovar Typhimurium el causante de la infección. La alta tasa de morbilidad (53%) y mortalidad (95%) (Mora- les et al., 2007), la presencia de portadores asintomáticos, los gastos de tratamiento y la posibilidad de contaminación de la carne o de sus subproductos durante el proceso de eviscerado pueden provocar grandes pérdi- das económicas al productor (Kabir, 2010).
El creciente número de reportes de ce- pas de Salmonella Typhimurium resistentes a antimicrobianos constituye un serio proble- ma epidemiológico en el control de la salmonelosis humana y animal (Kabir, 2010; Shu-Kee et al., 2015). La resistencia reportada frente a la familia de tetraciclinas en Salmonella enterica es mediada por los genes tetA, tetB, tetC, tetD, tete y tetG, que le confieren la capacidad de generar bombas de flujo, protección ribosomal e inactivación enzimática del fármaco (Faldynova et al., 2003); así mismo, es el caso de la resistencia a sulfonamidas mediada por los genes sul1, sul2 y sul3 que codifican isoformas del enzima blanco del fármaco (Antúnes et al., 2005).
El genoma de Salmonella enterica presenta una plasticidad que le permite adaptarse fácilmente a cambios en su medio ambiente gracias a un incremento en la variabilidad genética y la generación de nuevos serovares o genotipos (Sun et al., 2014). La plasticidad genética presente en cepas de Salmonella Typhimurium dificulta el desarrollo de estudios epidemiológicos, más aún considerando las limitaciones de los métodos microbiológicos y serológicos para la identificación y caracterización de las cepas circulantes (Kabir, 2010; Shu-Kee et al., 2015). No obstante, en la actualidad se cuentan con técnicas de tipificación molecular basadas en PCR como REP-PCR, ERIC-PCR (Enterobacterial Repetitive Intergenic Consensus - PCR) (Albufera et al., 2009), AFLP o basadas en secuenciamiento como el MLST (Achtman et al., 2012) que permiten detectar la variabilidad y polimorfismos genéticos presentes en Salmonella enterica (Prasertsee et al., 2016).
Una gran cantidad de estudios sobre tipificación molecular en Salmonella se han realizado en animales domésticos; sin embargo, los estudios en cuyes son insuficientes. Evaluaciones genéticas sumadas a la determinación de patrones de resistencia antimicrobiana en cepas de Salmonella Typhimurium involucradas en uno o más brotes de salmonelosis en cuyes permitiría establecer correlaciones epidemiológicas que ayuden a establecer la causa o posible origen de las infecciones, así como los mecanismos de transferencia de la resistencia antimicrobiana en esta especie, similar a lo realizado en otras especies domésticas (Shu-Kee et al., 2015). El presente estudio reporta una caracterización preliminar de perfiles fenotípicos y genéticos de resistencia a antibióticos y su relación con la variabilidad genética en cepas de Salmonella Typhimurium de cuyes.
MATERIALES Y MÉTODOS
Material Biológico
Treinta y cinco cepas provenientes de aislados clínicos de Salmonella enterica subs. enterica serovar Typhimurium (Salmonella Typhimurium) en cuyes obtenidos en 2016 (n=9) y 2018 (n=26) provenientes de la costa central de Lima, Perú, fueron utilizadas para la identificación de perfiles de resistencia antimicrobiana y caracterización genética.
Tres cepas de referencia: Salmonella enterica serovar Enteritidis (ATCC® 13076), Staphylococos aureus (ATCC® 29213), Salmonella enterica serovar Typhimurium (ATCC® 14028) fueron utilizadas como controles positivos y negativo. Las cepas de referencia fueron reactivadas siguiendo las instrucciones de uso del fabricante (Microbiologics PI.194.SPAN.LA Rev. A).
Todos los aislados clínicos y las cepas de referencia fueron transferidos a caldos enriquecidos Rappaport Vassiliadis y MullerKauffmann Tetrathionate e incubados a 37 °C por 24 horas, y finalmente sembradas en agar selectivo XLD (Xilosa Lisine Deoxycholate). Las colonias sospechosas a Salmonella sp fueron subcultivadas en agar no selectivo y posteriormente sometidas al análisis bioquímico, usando una batería de siete agares TSI (Triple Sugar Iron), LIA (Lysine iron agar), citrato de Simon´s, urea, rojo de metilo, Voges-Proskauer y SIM (sulfide, indole, motility) y confirmadas por sistema de identificación rápida API (Analytical Profile Index) 20E (BioMérieux). La interpretación de los resultados fue realizada siguiendo lo indicado por la Norma Española UNE-EN ISO 65791 (UNE, 2017).
Perfil de Resistencia Antimicrobiana
Una suspensión bacteriana en NaCl 0.8% con una turbidez de 0.5 unidades de escala de McFarland fue utilizada para evaluar la sensibilidad a siete familias de antibióticos: fenicoles (florfenicol, 30 µg), sulfonamidas (sulfametoxazol, 20 µg), tetraciclinas (doxiciclina, 30 µg; oxitetraciclina, 30 µg), penicilinas (amoxicilina, 20 µg), quinolonas (enrofloxacina, 10 µg; norfloxacina, 10 µg; levofloxacina, 5 µg; ciprofloxacina, 5 µg), polimixinas (colistina, 10 µg) y fosfonatos (fosfomicina, 200 µg) mediante el método de disco difusión en agar Mueller Hinton y una lectura de los halos de inhibición dentro de las 18 a 20 horas de incubación a 37 °C, siguiendo lo descrito por el Comité Nacional de Estándares Clínicos de Laboratorio (CLSI, 2017).
Extracción de ADN de Salmonella Typhimurium
ADN genómico y plasmídico de 35 cepas de Salmonella Typhimurium fue obtenido a partir de una colonia resuspendida en buffer fosfato salino (PBS) y extraído mediante el método de membranas de sílica utilizando el kit de extracción de ADN GF-1 Tissue DNA Extraction (Vivantis), siguiendo las indicaciones del fabricante. El ADN genómico fue almacenado a -20 °C hasta su procesamiento.
Género, Especie y Serovar
La presencia de Salmonella enterica fue confirmada en las 35 cepas mediante amplificación por PCR de un fragmento conservado del gen invA de 378 pares de bases en un volumen final de 20 µl conteniendo 1X Buffer PCR (Thermo Scientific), 2 mM dNTPs (Thermo Scientific), 2 mM MgCl (Thermo Scientific), 5 pmol de cada cebador (Macrogen), S139: 5´ GTGAAATTATCGCCACGTTCGGGCAA 3´ y S141: 5´TCAT-CGCACCGTCAAA-GGAACC 3´ (Rahn et al., 1992; Zahraei, 2005), 0.5 U de Hot Start Taq DNA polimerasa recombinante (Thermo Scientific) y 5 ng de ADN. Las condiciones termales fueron las siguientes: denaturación inicial de 95 °C por 4 min, 30 ciclos (94 °C por 30 s, 64 °C por 30 s, 72 °C por 30 s), extensión final de 72 °C por 7 min. La amplificación se llevó a cabo en un termociclador Veriti Thermal Cycler (Life Technologies). Los productos de PCR fueron separados mediante electroforesis en gel de agarosa al 1% en buffer TBE 1X (Tris 0.89 M, Borato 0.02M, EDTA 0.89 M, pH 8.3) durante 45 min a 100 v. y visualizados bajo luz UV.
Adicionalmente, la presencia de Salmonella enterica fue confirmada mediante amplificación por PCR de un fragmento de 1464 pares de bases del gen 16S rDNA (Miller et al., 2013) y su posterior secuenciamiento por ambas hebras utilizando BigDye Terminator v. 3.1 Cycle Sequencing Kit (Thermo Scientific) en el analizador genético ABI 3137 XL (Life Technologies). El servicio de secuenciamiento fue proveído por la empresa Macrogen Inc.
La presencia de Salmonella Typhimurium fue determinada mediante la amplificación de un fragmento de 559 bp del gen fliC por PCR en un volumen final de 20 µl conteniendo 1X Buffer PCR (Thermo Scientific), 2 mM dNTPs (Thermo Scientific), 1.5 mM MgCl (Thermo Scientific), 5 pmol de cada cebador (Macrogen), Fli15: 5´ GTGAAATTATCGCCACGTTCGGGCAA 3´ y Tym: 5´TCATCGCACCGTCAAA-GGAACC 3´ (Soumet et al., 1999), 0.5 U de Hot Start Taq DNA polimerasa recombinante (Thermo Scientific) y 5 ng de ADN. Las condiciones termales fueron las siguientes: denaturación inicial de 95 °C por 4 min, 30 ciclos (94 °C por 30 s, 56 °C por 30 s, 72 °C a 30 s), extensión final de 72 °C por 5 min. La amplificación se llevó a cabo en un termociclador Veriti Thermal Cycler (Life Technologies).
DNA Fingerprinting por ERIC-PCR
Perfiles de ERIC-PCR (ADN finger-printing - huella genética) fueron realizados para las 35 cepas en un volumen final de 20µl conteniendo 1X Buffer PCR (Thermo Scientific), 2 mM dNTPs (Thermo Scientific), 2.5 mM MgCl (Thermo Scientific), 5 pmol de cada cebador (Macrogen), ERIC 1R: 5´ATGTAAGCTCCTGGGGATT CAC3' y ERIC 2: 5'-AAGTAAGTGACTGGG GTGAGCG-3 (Albufera et al., 2009), 1 U de Hot Start Taq DNA polimerasa recombi-nante (Thermo Scientific) y 10 ng de ADN. Las condiciones termales fueron las siguientes: denaturación inicial de 95 °C por 4 min, 30 ciclos (90 °C por 30 s, 40 °C por 1 min, 72 °C por 1 min), extensión final de 70 °C por 8 min.
Los productos de PCR fueron separados por electroforesis en gel de agarosa al 1.2%, TBE 1X, a 100 v durante 1 h. Se realizó un score de tamaños de fragmentos mediante comparación con el marcador de peso molecular 100 bp plus (Thermo Scientific) utilizando un Fotodocumentador SmartView Pro 1200 Imager System (Major Scientific).
Perfiles de ERIC-PCR fueron transformados en una matriz de (dis)similaridad utilizando el coeficiente de Jaccard y Dice mediante el programa FAMD v. 1.25 (Schlüter y Harris, 2006), para luego generar un dendograma bajo el método UPGMA en base a una matriz de distancia genética utilizando los programas FAMD v. 1.25 (Schlüter y Harris, 2006) y Mega v. 6.0 (Tamura et al., 2013).
Detección de Genes de Resistencia
Los genes (n=14) qnrB, qnrD, qnrS, aac(62 )-Ib, tetA, tetB, tetC, tetD, cat1, cat2, cmlA, cmlB, sul1 y sul2 (Cheng et al., 2004) asociados a resistencia a quinolonas (Park et al., 2006; Cavaco et al., 2009; Jacoby et al., 2009), tetraciclinas, fenicoles y sulfamidas (Cheng et al., 2004) fueron amplificados por PCR en un volumen final de 20 µl conteniendo 1X Buffer PCR (Thermo Scientific), 2 mM dNTPs (Thermo Scientific), 2 mM MgCl2 (Thermo Scientific), 5 pmol de cada cebador (Cuadro 1), 1 U de Hot Start Taq DNA polimerasa recombinante (Thermo Scientific) y 5 ng de ADN. Las condiciones termales fueron las siguientes: denaturación inicial de 95 °C por 4 min, 30 ciclos (94 °C por 1 min, 51 °C por 1 min, 68 °C a 1 min), extensión final de 70 °C por 8 min.
Cuadro 1 Secuencias de cebadores utilizados para amplificación por PCR de 14 genes asociados con resistencia a quinolonas, tetraciclinas, fenicoles y sulfamidas
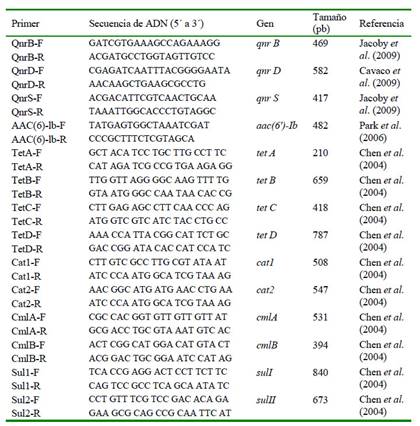
Los productos de amplificación se separaron por electroforesis en gel de agarosa al 1%, TBE 1X, a 100 v, durante 1 h. Se realizó un score de tamaños de fragmentos mediante comparación con el marcador de peso molecular 100 bp plus (Thermo Scientific) utilizando un Fotodocumentador SmartView Pro 1200 Imager System (Major Scientific).
RESULTADOS
Detección y Confirmación de Salmonella Typhimurium
Los 35 aislados presentaron un patrón morfológico en agar XLD y perfil bioquímico compatible con Salmonella enterica. La identificación molecular por PCR específica del gen invA y del fliC, así como del secuenciamiento del gen 16S rDNA confirmaron la presencia de Salmonella enterica subs. enterica serovar Typhimurium en todas las cepas.
Perfil de Resistencia Antimicrobiana
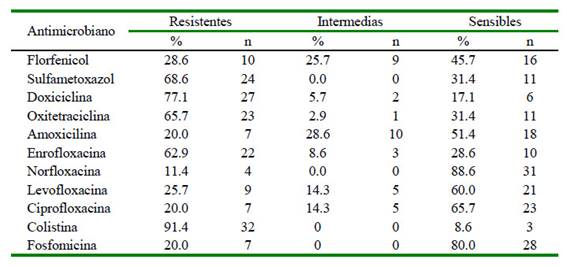
Todas las cepas (100%) mostraron resistencia antimicrobiana al menos a una familia de antibióticos y un 80% de aislados fueron multidrogo resistentes (resistencia al menos a tres familias de antibióticos). Los principales perfiles de resistencia fueron para colistina (91.4%), sulfametoxazol (68.6), enrofloxacina (62.9%) y una moderada resistencia a amoxicilina (20%), ciprofloxacina (20%) y norfloxacina (11.4%) (Cuadro 2).
Cuadro 2 Perfiles de resistencia de 35 cepas de Salmonella Typhimurium aialadas de cuyes a 11 antibióticos de siete familias (fenicoles, sulfonamidas, tetraciclinas, penicilinas, quinolonas, polimixinas y fosfonatos) por técnicas de difusión en disco de inhibición
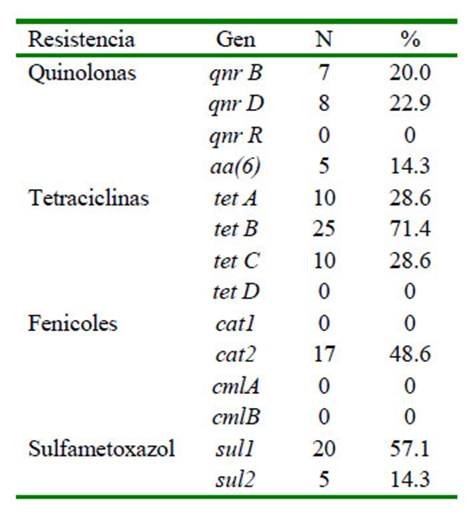
Nueve de 14 genes de resistencia analizados fueron detectados en las 35 cepas de Salmonella Typhimurium, siendo los genes tetB (71.4%), sulI (57.1%) y cat2 (48.6%) los más prevalentes (Cuadro 3).
Cuadro 3 Presencia de genes de resistencia a quinolonas (qnr), tetraciclinas (tet), fenicoles (cat, cml), sulfametoxazol (sul) de 35 cepas de Salmonella enterica serovar Typhimurium
Variabilidad Genética en Aislados de Salmonella Typhimurium
Se identificaron nueve bandas (loci) polimórficas comprendidas entre 200 y 2000-pares de bases con un promedio de 6.714 bandas y 7 perfiles genéticos de ERIC-PCR (A-G), siendo el más abundante el perfil D con 54.3% (19/35), seguido del perfil C con 28.6% (10/35) de la totalidad de cepas (Figura 1).
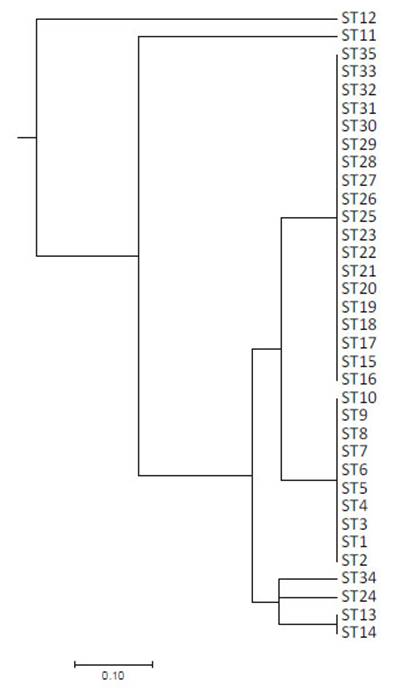
Figura 1 Dendograma basado en matriz de distancia genética utilizando el coeficiente Standard Jaccard y Dice utilizando método UPGMA para 35 cepas de Salmonella Typhimurium
La totalidad de aislados de 2016 pertenecieron al perfil C a diferencia de los aislados de 2018 donde se aprecia una mayor variación, con la presencia de siete perfiles de ERIC-PCR. Solo el 35.1% de la variación se explica por diferencias entre los años de colección de las cepas, mientras que el 64.9% de la variación se explica por diferencias intraespecíficas en las cepas analizadas mediante AMOVA (Va= 0.018, Vb= 0.034 y Vt= 0.052). Se apreció una moderada diferenciación entre las cepas de Salmonella Typhimurium por año de colección (PhiST= 0.3506), agrupándose al menos en dos clústeres genéticos (Figura 2).
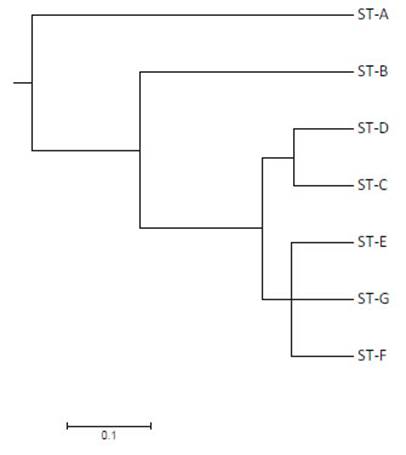
Figura 2 Dendograma basado en matriz de distancia genética utilizando el coeficiente Standard Jaccard y Dice utilizando método UPGMA para siete patrones de ERIC-PCR en Salmonella Typhimurium. Los siete genotipos en las 35 cepas han sido denominados mediante s letras (A-G) para visualizar las relaciones existentes entre los perfiles de ERIC-PCR
DISCUSIÓN
Perfiles de Resistencia a Antibióticos
Un alto porcentaje de cepas mostraron resistencia a colistina, sulfametoxazol y enrofloxacina, similar a lo reportado para otras especies animales e incluso al humano (Cabrera et al., 2004). La resistencia a las tetraciclinas fue similar a lo reportado en humanos (Onyango et al., 2008). En el presente estudio se corrobora la presencia de los genes tetA, tetB y tetC responsables de conferir el fenotipo de resistencia (Faldynova et al., 2003), pero una mayor frecuencia del gen tetB en las cepas provenientes de cuyes en Lima.
El fenotipo resistente a florfenicol presentó solo el gen cat2, presente en cepas de origen humano, a diferencia de lo reportado para otras cepas de origen animal con una mayor presencia del gen flo (Nogrady et al., 2005). Por otro lado, en contraste con el estudio de Matsuura et al. (2010) y Salvatierra et al. (2018) en cuyes de Perú, se observó la presencia de resistencia a penicilinas y sulfonamidas, con porcentajes de resistencia a amoxicilina de 20% y a sulfametoxazol de 68.6%, a pesar de que las penicilinas no se utilizan en los tratamientos médicos en cuyes (Madge, 1969; Young et al, 1987).
El fenotipo resistente al Sulfametoxazol en cuyes presentó los genes sul1 y sul2, y con mayor abundancia del primero; similar a reportes en cepas de Salmonella Typhimurium de origen animal y humano (Antúnes et al., 2005; Adesiji et al., 2014). Los fenotipos de resistencia a quinolonas (enrofloxacina y ciprofloxacina) fueron muy prevalentes, principalmente en cepas de 2018, duplicando lo reportado por Salvatierra et al. (2018). Ambos antibióticos son usados ampliamente en el control de salmonelosis en cuyes (Matssura et al., 2010). Por otro lado, se observó una discordancia entre la presencia del fenotipo de resistencia a la familia de quinolonas y la presencia de genes de resistencia, pudiendo sugerir la presencia de otros genes, mecanismos bioquímicos, o nuevas mutaciones como las producidas en los genes gyrA y parC relacionados a la resistencia a quinolonas (Nakaya et al., 2003).
Los sistemas de producción aviar constituyen una fuente para la emergencia de las cepas resistentes a antibióticos en Salmonella enterica debido al uso de antibióticos en alimentación animal y subdosificación (Verbrugghe et al., 2016). Así mismo, se ha reportado la transferencia de genes de virulencia y resistencia por medio de mecanismos de transferencia horizontal de genes como plásmidos conjugativos, integrones y transposones (Pezzella et al., 2004) entre cepas de Salmonella de diferentes orígenes. La posibilidad de contacto entre patógenos de sistemas de producción avícola hacia los sistemas de producción de cuyes es una posible explicación, ya que ambos se concentran en la costa central de Lima.
Variabilidad Genética en Aislados de Salmonella Typhimurium
Salmonella Typhimurium constituye la principal serovariedad presente en cuyes, con valores superiores al 95% en comparación con otros serovares (Chero et al., 2017). El presente estudio reporta una moderada variabilidad genética con la presencia de siete patrones de ERIC-PCR distribuidos aleatoriamente en los sitios de muestreo, a diferencia de lo reportado por Salvatierra et al. (2018) quienes encuentran un solo perfil genético acorde con una dispersión clonal en granjas de cuyes de Lima. Por otro lado, no se observa una clara correlación entre la variabilidad genética y los perfiles de resistencia antibiótica, similar a lo reportado por Salvatierra et al. (2018); sin embargo, contrasta con los resultados de Coculescu et al. (2014) sobre genotipos presentes con mayor frecuencia en casos de resistencia a betalactámicos.
La moderada variabilidad y distancia genética existente entre cepas de Salmonella Typhimurium pertenecientes a los años 2016 y 2018, sumada a los diferentes patrones de resistencia a los antibióticos podría sugerir un diferente proceso de selección al que fueron expuestas en dicho intervalo las cepas de Salmonella Typhimurium en las granjas de cuyes de Lima.
CONCLUSIONES
El 20% de las cepas de Salmonella Typhimurium fueron resistentes a antibióticos de uso humano como ciprofloxacina y amoxicilina, mientras que el 62.9% fueron resistentes a enrofloxacina.
Las cepas aisladas de Salmonella Typhimurium de cuyes presentaron moderados valores de variabilidad genética y una alta prevalencia de cepas multidrogo resistentes (80%), principalmente en los aislados pertenecientes al año 2018.












 uBio
uBio 

