INTRODUCCIÓN
La circovirosis porcina es una enfermedad de distribución mundial causada por el circovirus porcino tipo 2 (PCV2), un virus de ADN de la familia Circoviridae (Todd et al., 1991) que afecta tanto a cerdos jóvenes como adultos, produciendo un fuerte impacto económico sobre la producción porcina (Alarcon et al., 2013; Zaveckas et al., 2015). El PCV2 se ha asociado con la mayoría de los casos clínicos de cerdos, especialmente con el síndrome del desmedro multisistémico posdestete (PMWS) (Grau-Roma et al., 2008; Ellis, 2014), el síndrome de la dermatitis ynefropatía porcina (PDNS) y algunos trastornos reproductivos (Olvera et al., 2007; Zhao et al., 2014), los cuales se han nombrado de manera colectiva como enfermedades asociadas al circovirus porcino (PCVAD).
La presencia de PCV2 en células de la médula ósea ha sido relacionada con infección persistente en cerdos que permitiría el mantenimiento y propagación del virus en las granjas porcinas (Wang et al., 2013; Kweon et al., 2015). Recientemente, el PCV3 ha sido descrito en varios países asociado a inflamación multisistémica, fallas reproductivas y PDNS (Zhai et al., 2017; Hayashi et al., 2018; Klaumann et al., 2018; Sukmak et al., 2019) en algunos casos en coinfección con bacterias (Hayashi et al., 2018) y virus, incluyendo el PCV2 (Li et al., 2018).
Diversos trabajos han demostrado una amplia diversidad genética presente en el PCV2, adscrita principalmente a su naturaleza de virus de ADN de cadena simple con una alta tasa de sustitución de nucleótidos y una alta probabilidad de mutaciones (Firth et al., 2009). PCV2a fue el genotipo más prevalente en cerdos clínicamente afectados, después de lo cual el PCV2b fue el más predominante y nuevos cambios en esa predominancia han sido asociados al uso de vacunas contra PCV2 a nivel mundial (Franzo y Segalés, 2018). No obstante, Franzo et al. (2015) demostraron que ocho cepas obtenidas de cerdos silvestres en Brasil pertenecían a cuatro genotipos: PCV2a, PCV2b, PCV2c y PCV2d. Sumado a esta diversidad, se han descrito que algunas mutaciones del PCV2 han demostrado generar una mayor patogenicidad en las cepas virales (Opriessnig et al., 2014). Así, el PCV2 cuenta con genotipos a, b, c, d, e y f (Wei et al., 2013; Neira et al., 2017; Correa-Fiz et al., 2018) y recientemente estudios filogenéticos proponen los genotipos PCV2g y PCV2h, basados en criterios de distancias genéticas entre los clusters (Franzo y Segalés, 2018).
En Colombia, algunos estudios han demostrado la presencia de PCV2 en varias regiones del país (Rincón-Monroy et al., 2015). Sin embargo, se desconoce la presencia del PCV2 en las regiones del Tolima y Huila, las cuales representan parte del suroccidente y centro-oeste del país. El objetivo del presente estudio fue detectar y caracterizar de manera preliminar el circovirus porcino tipo 2 (PCV2) en granjas porcícolas de los departamentos de Huila y Tolima, Colombia.
MATERIALES Y MÉTODOS
Granjas Porcícolas
Se llevó a cabo un estudio transversal en cinco granjas porcícolas comerciales del departamento del Huila, en el suroccidente de Colombia y en otras tres granjas del departamento del Tolima en el centro-oeste del país. En estas granjas se tomaron muestras, sin tener en cuenta la línea genética. Los animales eran alimentados durante todo su ciclo productivo con concentrado comercial.
Animales y Toma de Muestras
Se tomaron muestras de sangre de 20 cerdas reproductoras de diferentes edades una semana antes del parto, de 20 lechones destetos entre 5 y 10 semanas de vida, de 20 cerdos de levante entre 11 y 16 semanas y de 20 cerdos cebados entre 17 y 23 semanas de cada granja (Figura 1). En ninguna de las granjas se realizaba la vacunación contra el circovirus porcino. Los animales fueron seleccionados aleatoriamente entre cerdos sanos y con signos clínicos relacionados con enfermedades asociadas al circovirus porcino (PCVAD). Las muestras fueron colectadas de la vena yugular mediante sistema BD Vacutainer®, siguiendo métodos de recolección estándar, minimizando el daño o estrés a los animales (Uhart et al., 2016) para la obtención de sangre completa y suero.
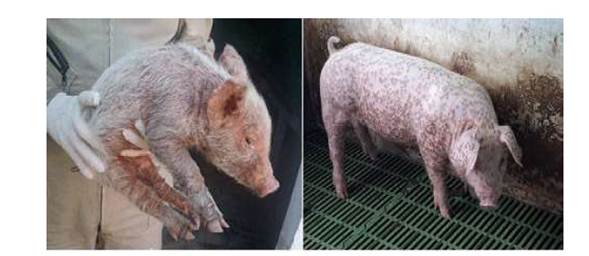
Figura 1 Animales positivos a PCV2 muestreados en granjas del Tolima y Huila. Izquierda: Lechón con signos respiratorios y nerviosos leves, con retraso en el crecimiento comparado con la camada, siendo un animal compatible con PMWS. Presentaba pelo hirsuto y costras en la piel. Derecha: Animal adulto con lesiones cutáneas compatibles con circovirosis porcina
Se crearon pooles de sueros de cinco animales (Nielsen et al., 2018), los cuales fueron enviados al Laboratorio de Diagnóstico Veterinario del Instituto Colombiano Agropecuario (ICA) para la medición de anticuerpos circulantes por medio de la técnica de ELISA (McNeilly et al., 2002). En los predios que se hallaron animales con signos clínicos compatibles con circovirosis porcina asociados a cuadros respiratorios y cutáneos severos, se realizaron necropsias y se tomaron muestras de órganos (i.e. linfonodos, pulmón, riñón) para la detección del PCV2.
Adicionalmente, se realizó una encuesta epidemiológica de aspectos básicos de manejo de la producción como la bioseguridad, nutrición, reproducción, haciendo especial énfasis en los aspectos sanitarios, con el fin de establecer los posibles factores de riesgo, mediante el análisis de tablas de contingencia y el cálculo del odds ratio. Para ello se empleó el software GraphPad Prism v. 6 para MacOS (Graph Pad software, San Diego, USA).
Detección Molecular de PCV2 mediante PCR y Secuenciamiento
Se realizó extracción de ADN total a partir de sangre completa y tejidos de diversos órganos mediante el kit Invisorb® Spin Universal (STRATEC Molecular,Alemania) siguiendo el procedimiento recomendado por el fabricante. El PCV2 fue detectado en los tejidos mediante amplificación de un fragmento de 657 pares de bases (pb) utilizando cebadores (primers) específicos (FGCCAGTTCGTCACCCTTT, R-CTCCCGCACCTTCGGATAT) para el virus (Quintana et al., 2002). Adicionalmente, se amplificó el marco de lectura abierto 2 (ORF2) completo del virus, el cual codifica para la proteína de cápside viral (Nawagitgul et al., 2000), mediante los cebadores F-GCCAGTTCGTCACCCTTT y RCAGC-GCACTTCTTTCGTT, basados en la secuencia de la cepa Zhuji2003 (número de acceso AY579893), con un tamaño de amplicón de 824 pb. La reacción de PCR se llevó a cabo en un termociclador T-100 (Bio-Rad, USA) usando un volumen final de reacción de 25 µl, compuesto por 14.87 µl de agua destiladadesionizada, 5 µl de 5x Colorless GoTaq® Flexi Buffer, 1 µl de dNTPs (1.5 mM), 1 µl de cada cebador (forward y reverse) (10 pmol/µl), 1 µl MgCl2 (25 mM), 0.125 µl de GoTaq® Flexi DNA polymerase (Promega, Madison, USA) y 1 µl de la muestra de ADN genómico.
La amplificación consistió en un ciclo de desnaturalización inicial a 95 °C por 3 min, seguida de 35 ciclos de desnaturalización a 95 °C por 30 s, anillaje a 55 °C por 30 s, extensión a 72 °C por 60 s y el último paso de extensión final a 72 °C por 7 min. Los amplicones fueron revelados mediante electroforesis horizontal en gel de agarosa al 1%, teñidos con Hydragreen® (ACTGene, USA) y visualizados bajo la luz ultravioleta mediante un documentador de geles ENDURO GDS™ (LabNet Intl, USA). Los amplicones del ORF2 completo fueron secuenciados a través del secuenciamiento de Sanger en Macrogen (Seoul, Korea), sometidos a análisis bioinformático, mediante el software Geneious v. 8.1.9 (Biomatters) (Kearse et al., 2012) y comparados con lo reportado en la base de datos del GenBank (National Center for Biotechnology Information -NCBI, USA).
RESULTADOS Y DISCUSIÓN
El análisis serológico demostró la presencia de animales en diferentes etapas de producción que se encontraban seropositivos al PCV2 (n= 25; 15 etapa de ceba provenientes del Huila, 10 etapa de levante provenientes del Tolima), con títulos de anticuerpos que demuestran una infección anterior o contacto con el virus. Todos los animales seropositivos fueron positivos a PCV2 mediante prueba de PCR. En una de las granjas, los títulos de anticuerpos demostraron la posibilidad de una infección activa. Estos hallazgos concuerdan con los reportes de seroprevalencia realizados por Liu et al. (2002) y Mesu et al. (2000) quienes demostraron que animales sanos, sin lesiones discernibles de alguna forma clínica de circovirosis, muestran títulos de anticuerpos considerados como positivos o expuestos. Así mismo, Larochelle et al. (2003) demostraron que los perfiles serológicos de animales sanos fueron iguales a los de aquellos animales con PMWS. Por otro lado, se ha descrito que esta serorreactividad está ampliamente relacionada con la cercanía a zonas o áreas donde el virus es endémico o está circulante y haya una posible transmisión (Mesu et al., 2001; Liu et al., 2002).
En el presente estudio se evaluó la presencia de nucleótidos virales en suero y tejidos de animales sanos y enfermos; no obstante, no fue posible su detección en algunas muestras de suero, por lo que se optó por el uso de sangre completa para la extracción de ADN y amplificación a través de PCR (Figura 2), lo cual arrojó algunos animales positivos en las mismas muestras detectadas como negativas previamente. En el trabajo de Liu et al. (2002) se ha descrito que cerdos infectados experimentalmente muestran niveles de nucleótidos detectables en los tejidos y que pueden no producir una señal detectable en sangre. Adicionalmente, se considera que pese a encontrarse el PCV2 en plasma/suero y a través de las células sanguíneas, su predilección por replicación en macrófagos/ monocitos (Sun et al., 2016) hace más factible utilizar fracciones celulares para la detección de los antígenos virales. En el presente estudio, la presencia de animales positivos a PCV2 sin signos clínicos compatibles con circovirosis porcina sugiere la presencia de infecciones subclínicas o variaciones en la virulencia de las cepas infectantes (Larochelle et al., 2003; Aiki-Raji et al., 2018).
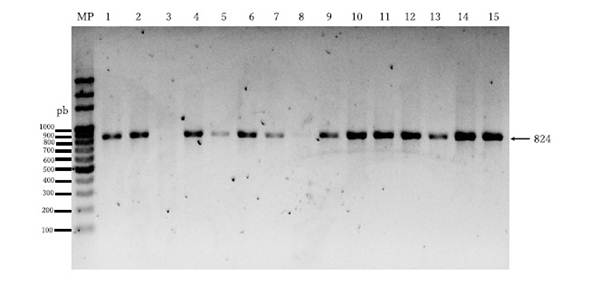
Figura 2 Gel de agarosa mostrando la amplificación del ORF2 del PCV2 mediante PCR convencional de algunas muestras representativas de tejidos de cerdos de los departamentos del Tolima y Huila, Colombia. MP: marcador de peso (100 pb, MiniPCR, Amplyus, USA), 1-15: muestras. La muestra 3 fue negativa, la muestra 8 presenta una banda tenue, confirmada con el secuenciamiento
Las muestras que resultaron positivas a la amplificación del ORF2 de PCV2 fueron secuenciadas y comparadas con secuencias de referencia depositadas en el GenBank. Todas las muestras enviadas a secuenciar fueron confirmadas como PCV2 ORF2 y correspondieron al PCV2d, mostrando un porcentaje de identidad de nucleótidos entre ellas y las depositadas en las bases de datos de un 93-99%. Estos hallazgos difieren con aquellos de Rincón-Monroy et al. (2014) para los departamentos del Cundinamarca, Valle, Antioquia y Risaralda en Colombia, donde se demostró la presencia de PCV2 de diferentes genotipos, en su mayoría el 2b.
En el presente estudio se realizaron encuestas epidemiológicas tendientes a establecer factores de riesgo asociados a la presencia del PCV2; sin embargo, no se detectaron asociaciones significativas entre la positividad al PCV2 y las características de producción y manejo de los cerdos en la granja (datos no mostrados), lo cual puede deberse al bajo número de unidades de producción muestreadas. En Colombia se han establecido las buenas prácticas de producción que deben seguir las ganaderías en general y la producción porcina en particular, siendo claro que el cumplimento del componente de bioseguridad y sanitario reduce el riesgo de transmisión de microorganismos potencialmente patógenos. Este incluye la desinfección, sistemas todo dentro todo fuera, restricción de movimiento de animales entre producciones, entre otros (ICA, 2011). Sin embargo, en países con alto nivel de tecnificación se han demostrado valores altos de prevalencia a PCV2, lo cual ha llevado a pensar que la restricción de movimiento de los animales no está reduciendo la diseminación del PCV2 (Liu et al., 2002) y se deben buscar programas de vigilancia activa que permitan un seguimiento, no solo a los animales a movilizar, sino también a la dinámica de la infección dentro de una área o región.
CONCLUSIONES
En las granjas porcícolas de las regiones del Tolima y Huila en Colombia se encuentra circulante el circovirus porcino tipo 2d (PCV2d), tanto de manera clínica como subclínica. La presencia del virus en animales clínicamente sanos y en granjas sin vacunación contra el virus denotan la circulación del virus en estas regiones y demanda estudios complementarios de vigilancia activa y caracterización epidemiológica de esta infección.












 uBio
uBio 

