INTRODUCCIÓN
Las garrapatas (Acari: Ixodidae) son ectoparásitos hematófagos de vertebrados y vectores de una variedad de patógenos causantes de enfermedades en humanos y animales.Además, existe una afinidad entre una especie de garrapata y determinados patógenos (Bowman y Nuttall, 2008). Trabajos realizados enAmérica del Sur, incluyendo al Perú, reportan a Rhipicephalus sanguineus s.l como la especie de garrapata con mayor frecuencia en caninos, basándose en diagnósticos morf.ológicos (Estares, 1999; Nunton et al., 2013).
Estudios realizados en poblaciones de R. sanguineus s.l. demuestran diversidad morfológica (de Oliveira et al., 2005), biológica e incompatibilidades reproductivas (Szabó et al., 2005), así como una amplia divergencia genética intraespecífica (Moraes-Filho et al., 2011), sugiriendo la existencia de dos variantes o genotipos denominados Linaje del Norte y Linaje del Sur bajo (Nava et al., 2012). Estas diferencias refuerzan de manera razonable la hipótesis de que existen dos especies bajo el nombre R. sanguineus s.l. enAmérica Latina (Dantas-Torres, 2008), la primera distribuida entre México y Brasil (Linaje del Norte) y la segunda ubicada en la porción Sur del continente (Linaje del Sur) (Nava et al., 2012).
En América del Sur, R. sanguineus s.l. ha sido reconocido como potencial vector de Ehrlichia canis, Anaplasma platys, Babesia canis vogeli, Hepatozoon canis, Rickettsia conorii, Rickettsia rickettsii y Rickettsia massiliae (Nava et al., 2017). Sin embargo, existen diferencias en la competencia vectorial entre los linajes del Norte y Sur, mostrando así que garrapatas pertenecientes al Linaje del Norte son vectores competentes de Ehrlichia canis (Cicuttin et al., 2015; Moraes-Filho et al., 2015).
La exacta identificación de las garrapatas es importante para el control de las enfermedades que transmiten y ha sido tradicionalmente logrado a través de criterios morfológicos llegando a la identificación de género y especie (Mangold et al., 1998). Sin embargo, la identificación de garrapatas por criterios morfológicos no permite diferenciar los diferentes linajes presentes en R. sanguineus s.l.; además, puede ser difícil cuando los especímenes se encuentran físicamente dañados (Caporale et al., 1995), falten es-tructuras importantes para la identificación o existen dificultades técnicas debidas a la falta de experiencia para realizar la identificación morfológica (Takano et al., 2014). Por otro lado, los métodos de identificación molecular que emplean marcadores moleculares de ADN han probado ser herramientas que pueden superar dichas limitaciones y han sido usados no solo para identificar especies de garrapatas (Szabó et al., 2005), sino también para realizar estudios de filogenia de garrapatas duras y blandas usando el gen 16S rADN (Blacky Piesman,1994)e identificar los linajes del Norte y Sur de R. sanguineus s.l. (Nava et al., 2012; Chitimia-Dobler et al., 2017; Dantas-Torres et al., 2017).
En el Perú la enfermedad más importante transmitida por R. sanguineus s.l. es la ehrlichiosis canina, hallándose valores altos de seropositividad y visualización de mórulas microscópicas en caninos en la región norte del país (Robles, 2008; Oliva, 2015). Por lo tanto, la correcta identificación de esta garrapata en la región norte es importante para el entendimiento de la epidemiología y el mejoramiento de los programas de control y prevención de las enfermedades que trasmiten.
MATERIALES Y MÉTODOS
Material Experimental
Se trabajó con 297 garrapatas de 74 perros en el distrito de Chiclayo (provincia de Chiclayo, departamento de Lambayeque, Perú), entre enero y abril de 2016. La zona tenía una temperatura ambiental entre 27 y 35 °C y una humedad relativa de 78%. Las garrapatas incluidas en el muestreo provenían de perros con ehrlichiosis diagnosticados mediante la evaluación de la historia clínica, signos clínicos, parámetros hematológicos, observación de mórulas microscópicas en leucocitos mononucleares y descartes serológicos.
Muestras
Las garrapatas fueron colectadas utilizando pinzas curvas de punta roma siguiendo la técnica de Needham (1985), haciendo énfasis en la oreja, axila y región interdigital (Dantas-Torres, 2010). Se conservaron en crioviales de tapa rosca con alcohol al 70% para la preservación de ADN (Mtambo et al., 2006) y remitidas a la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos, Lima.
Identificación Morfológica
Las garrapatas fueron observadas bajo lupa estereoscópica y microscópica, identificándose el género, especie, sexo y estadio evolutivo, siendo sometidas a un proceso de aclaramiento tisular con hidróxido de potasio. La identificación morfológica se realizó siguiendo las claves taxonómicas de Keirans y Litwak (1989) y Quiroz (2008), observando presencia y distribución de escudo, posición de piezas bucales, dimorfismo sexual, posición del surco anal, segundo segmento del palpo, forma de la base del capítulo, tamaño de palpos, tamaño de hipostoma y número de festones.
Identificación Molecular
Nueve especímenes identificados morfológicamente como R. sanguineus s.l. obtenidos de nueve perros (una garrapata por perro) fueron seleccionados para identificarlos mediante procedimientos moleculares. El ADN fue extraído siguiendo el protocolo elaborado por Kelehear et al. (2013) modificado. Para esto, se usó una pequeña muestra de tejido preservada en etanol absoluto o molecular, se secó al aire libre en una estufa o con ayuda de una centrífuga Vacuum, se incubó toda la noche a 56 °C (baño María) en un vial eppendorf con 100 µl de TEN buffer (404 mM Tris/ HCL; 20 mM EDTA, pH 7.2), 1% SDS y 0.2 mg/mL de proteinasa K (56 °C durante 12 h) (Buffer TEN y 10 µl proteinasa K 40 mg/µl). Posteriormente se agregó 75 µl de acetato de amonio (3M), se homogenizó en un vortex y se sedimentó en una microcentrífuga. La muestra se enfrió a -80 °C durante 10 min, se centrifugó a 14 000 g durante 5 min. El sobrenadante se pasó a un vial de 2 ml (eppendorf) y se añadió 2 volúmenes de etanol, se mezcló y se enfrió a -80 °C por 10 min, se repitió la centrifugación y se descartó el sobrenadante. El sedimento (pellet) se lavó dos veces en etanol al 70% (175 µl), centrifugando cada vez la muestra a 14 000 g durante 10 min. Luego la muestra se secó con ayuda de una centrífuga Vacuum (vacío-30 min) y se resuspendió en 100 µl de buffer TE. Finalmente, el ADN resultante se almacenó en congelación.
Las reacciones de amplificación fueron realizadas en un volumen total de 50 µl. Cada tubo de PCR contenía una mezcla de 1.3 µl de cada cebador (16S + 1 CTGCTCAATGAIIIIIITTAAATTGCTGTGG; 16S - 1 TCGGTITAAACTCAGATCATGT) (Fiorini et al., 2014), 25 µl de 2x GoTaq Green PCR master mix (2X buffer pH 8.5, 400 µM dATP, 400 µM dGTP, 400 µM dCTP, 400 µM dTTP and 3 mM MgCl2), 18.4 µl de agua molecular libre de nucleasas y 4 µl de ADN genómico para completar el volumen total de las reacciones. Se incluyó una muestra blanco, donde el ADN problema era sustituido por un volumen similar de agua, y un control positivo usando ADN de Amblyomma rotundatum. Las amplificaciones se hicieron con un termociclador SimpliAmp (Applied Biosystems) siguiendo el siguiente perfil de ciclo: 1 ciclo de 95 °C durante 5 min, 35 ciclos de desnaturalización de 95 °C durante 30 s, alineación a 50 °C durante 30 s y extensión a 72 °C durante 40 s, y una extensión final de 72 °C durante 7 min. Finalmente, los productos del PCR fueron analizados en electroforesis en agar gel y visualizados en un transiluminador (Cleaver Cientific).
Los productos de amplificación del PCR fueron purificados y enviados a una empresa privada para ser sometidos a secuenciamiento genético (Macrogen, Korea). Las secuencias obtenidas del gen 16S rADN de cada garrapata fueron alineadas usando el programa ChromasPro. Las secuencias del Perú (denominadas Sample 78, 80, 81, 82, 83, 84, 85, 86, 87) fueron comparadas con las referencias de secuencias de Brasil (A y B), Colombia, Chile, Uruguay,Argentina, España, Italia y Sudáfrica provenientes de la base de datos del GenBank (Cuadro 1). En el análisis filogenético se utilizó el Programa MEGA7, midiendo la distancia evolutiva inferida desde la secuencia de nucleótidos. Las distancias genéticas fueron calculadas a través del método «neighbor-joining»con la distancia de 2-parámetros de Kimura.
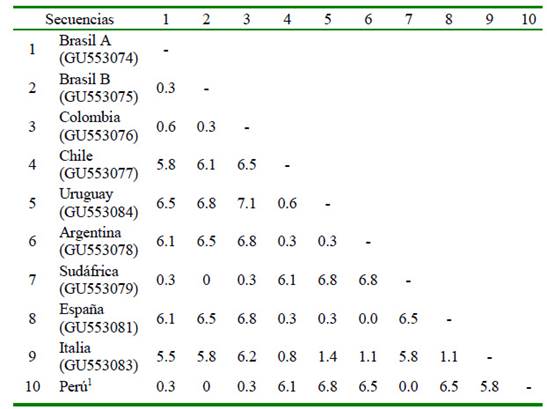
Cuadro 1 Matriz de divergencia intraespecífica absoluta (porcentaje de diferencia de nucleótidos) de secuencias parciales de 358 pares de base del gen mitocondrial 16S rADN de Rhipicephalus sanguineus s.l. obtenidas del presente estudio y referencias del GenBank
1 Consenso de las secuencias obtenidas en el presente estudio
RESULTADOS
Identificación Morfológica
Las 297 garrapatas fueron identificadas como R. sanguineus s.l. De estas, solo una estaba en estadio de ninfa y el resto era adulta (175 hembras y 121 machos). Los especímenes adultos presentaron las siguientes características morfológicas: presencia de capítulo (Figura 1A), surco anal posterior al ano (Figura 1B), segundo segmento del palpo sin proyección lateral hacia el margen del capítulo, ojos presentes, presencia de festones, base del capítulo hexagonal, piezas bucales pequeñas (Figura 1C), palpos tan o más lagos que la base del capítulo o el hipostoma, coxa I profundamente hendida (Figuras 1D,E), placa espiracular en forma de coma (Figura 1F). El escudo quitinoso en los machos se extendía en un 80-100% de la superficie dorsal, mientras que en las hembras solo cubría la mitad de la superficie (Figura 1G). Además, los machos adultos presentaban placas adanales y placas accesorias, las cuales estaban ausentes en las hembras.
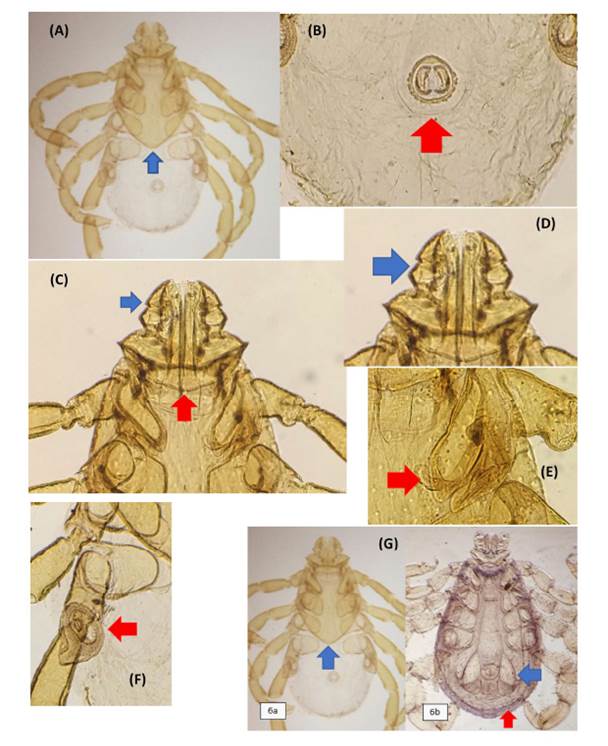
Figura 1 Garrapatas R. sanguineus s.l. (A) Presencia de capítulo y de escudo quitinoso (flecha azul); (B) Vista ventral. Presencia de surco anal posterior (flecha roja); (C) Vista dorsal. Piezas bucales pequeñas. Segundo segmento del palpo sin proyección lateral y ambos palpos no son más largos que la base del capítulo (flecha azul). Base del capítulo hexagonal (flecha roja); (D, E) Vista ventral. Palpos más largos que el hipostoma (flecha azul), presencia de coxa dividida del primer par de patas (flecha roja); (F) Vista ventral. Placa espiracular en forma de coma (flecha roja); (G) Escudo quitinoso cubriendo la superficie dorsal en un 50% en un espécimen hembra (6a, flecha azul) y 100% (6b, flecha roja) además de presentar un par de placas adanales (6b, flecha azul) en un espécimen macho de R. sanguineus s.l.
Identificación Molecular
Se amplificaron 430 pb de nucleótidos del gen 16S rADN para las nueve garrapatas identificadas morfológicamente como R. sanguineus s.l. El análisis de las secuencias confirmó el diagnóstico morfológico previo para R. sanguineus s.l. al ser comparadas con secuencias de referencia registradas en el GenBank. Las nueve secuencias obtenidas presentaron una divergencia genética intraespecífica de 0%; es decir, que se puede considerar como una sola.
Las diferencias entre secuencias de BrasilA, Brasil B, Colombia, Chile, Uruguay, Argentina, Sudáfrica, España, Italia y Perú iban desde 0 hasta 7.1%. Las diferencias entre las secuencias de Brasil A, Brasil B, Colombia (pertenecientes al conocido Linaje del Norte) fueron de 0.3-0.6%, mientras que las diferencias entre las secuencias de Chile, Uruguay, Argentina (pertenecientes al Linaje del Sur) fueron de 0.3-0.6%. De esta manera se observa que las secuencias de los países pertenecientes al Linaje del Norte y Linaje del Sur diferían de 5.8-7.1%. Por otro lado, las secuencias de Sudáfrica difieren de 0-0.3% con el Linaje del Norte y las secuencias de España e Italia difieren con el Linaje del Sur de 0-0.3 y de 0.8-1.4%, respectivamente.
La divergencia genética entre las secuencias del Perú y de países del Linaje del Norte fueron de 0-0.3%, siendo las secuencias de Perú totalmente idénticas a las de Brasil B, mientras que las diferencias con las secuencias de países del Linaje del Sur fueron de 6.1-6.8%.Además, la divergencia con las secuencias de Italia, España y Sudáfrica fueron de 5.8, 6.5 y 0%, respectivamente. Los valores absolutos de divergencia genética entre las secuencias parciales del gen mitocondrial 16S rADN se muestran en el Cuadro 1.
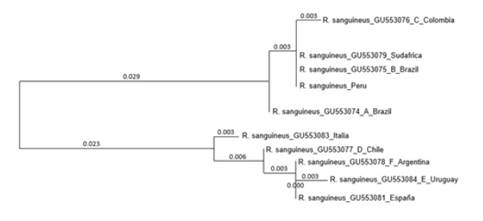
El análisis filogenético inferido por Neighbor-joining generó un árbol que segregó las secuencias de R. sanguineus s.l. en dos clases: una representada por las secuencias de Brasil A, Brasil B, Colombia (Linaje del Norte), Sudáfrica y Perú y una segunda clase representada por las secuencias de Chile, Uruguay, Argentina, España e Italia (Figura 2).
DISCUSIÓN
Las 297 garrapatas fueron identificadas morfológicamente como R. sanguineus s.l., especie ampliamente distribuida a nivel mundial (Walker et al., 2003), posiblemente debido a su relación con humanos y perros (Dantas-Torres, 2008). Esta fue la única especie de garrapata, colectada de 74 perros en el norte del Perú, a pesar de haberse reportado Amblyomma maculatum y Amblyomma ovale en la misma zona geográfica (Glenny et al., 2004), siendo fácilmente diferenciadas por el tamaño de los palpos en relación con la base del capítulo (Keirans y Litwak, 1989).
El análisis de las secuencias parciales de 16S rADN de R. sanguineus s.l. muestran nula diversidad entre ellas (0%) y muy pocas diferencias (0-0.3%) con las secuencias de Brasil A-B y Colombia (Linaje del Norte), reportadas también por Szabó et al. (2005), Moraes-Filho et al. (2011) y Nava et al. (2012). Además, el análisis filogenético agrupa las secuencias del Perú junto a las secuencias de Brasil y Colombia (Linaje del Norte), indicando su pertenencia al Linaje del Norte. Los datos climáticos de la zona del muestreo corresponden a climas tropicales y subtropicales adecuados para la distribución de R. sanguineus s.l del Linaje del Norte (Moraes-Filho et al., 2011).
Las secuencias parciales de 16S rADN de R. sanguineus s.l del Perú (Linaje del Norte) presentaron una divergencia intraespecífica de 6.1-6.8% con las secuencias de Chile, Uruguay y Argentina (Linaje del Sur), lo cual concuerda con el estudio de Nava et al. (2012). Esta divergencia, no obstante, fue mayor al 3.3% reportada entre R. sanguineus s.l. y Rhipicephalus guilhoni por Latrofa et al. (2013), mostrando de esta manera un alto porcentaje de divergencia genética entre los dos linajes.
El árbol filogenético agrupó las secuencias obtenidas con las de Sudáfrica (100% de identidad). Esto podría deberse a la introducción de R. sanguineus s.l. al Nuevo Mundo antes del siglo XV gracias a la migración humana proveniente de Asia y África (Leonard et al., 2002), además del comercio de esclavos provenientes del continente africano, permitiendo el establecimiento de R. sanguineus s.l. del Linaje del norte en Sudamérica debido a las condiciones de temperatura encontradas en dicho territorio (Burlini et al., 2009).
Los casos de ehrlichiosis canina en Chiclayo presentan una prevalencia de 75.8%, teniendo a R. sanguineus s.l. como responsable de su transmisión (Oliva, 2015). Esta capacidad vectorial fue evaluada por Cicuttin et al. (2015) entre los dos linajes mostrando la habilidad de R. sanguineus s.l. del Linaje del Norte para infectarse naturalmente con Ehrlichia canis, y su capacidad para transmitir la bacteria a la población canina con la presentación de ehrlichiosis monocítica canina (Moraes-Filho et al., 2015).
CONCLUSIONES
Las garrapatas colectadas de perros con ehrlichiosis en Chiclayo, Perú, fueron identificadas morfológicamente como Rhipicephalus sanguineus s.l.
Los análisis morfológicos y moleculares para la identificación de R. sanguineus s.l. dieron los mismos resultados.
Las secuencias de R. sanguineus s.l. colectadas en Chiclayo indican que estas garrapatas pertenecerían al Linaje del Norte.












 uBio
uBio