INTRODUCCIÓN
La industria porcícola colombiana ha evidenciado en los últimos 10 años un notable y constante crecimiento. En el primer trimestre de 2019, el sacrificio de ganado porcino fue de 1 097 420 cabezas, comparado con las 995 527 cabezas para el mismo periodo del año anterior (DANE, 2019). Asimismo, el consumo per cápita se ha incrementado hasta casi duplicarse en la última década, llegando a los 9.3 kg/habitante (Maya, 2018).
A lo largo de la cadena productiva porcícola, se debe asegurar la calidad de los productos destinados al consumo humano mediante la implementación de programas de mejoramiento y fortalecimiento de las medidas de bioseguridad, manejo y sanidad (PorkColombia, 2014). La presencia de patógenos, especialmente aquellos que producen alteraciones gastrointestinales, ocasionan importantes pérdidas económicas en la industria porcícola, además de constituir un riesgo para la salud pública dado su potencial zoonótico. Entre estos microor-ganismos se encuentran bacterias, parásitos, hongos y virus como Astrovirus, Rotavirus del grupo A, Norovirus y Virus de la Hepatitis E (Monini et al., 2015).
Rotavirus es un virus desnudo de forma icosaédrica cuya cápside está constituida por tres capas proteicas. La cápside externa está conformada por las proteínas estructurales VP7 y VP4, la intermedia por VP6 y la nucleocápside por VP2 (Long y McDonald, 2012). Su genoma se encuentra compuesto por 11 genes que forman una cadena doble de RNA (dsRNA) segmentado y que codifican seis proteínas estructurales (VP1-VP4 y VP6-VP7) y cinco proteínas no estructurales (NSP1-NSP5/NSP6), dentro de las cuales se encuentra la proteína NSP4, primera enterotoxina viral descrita, que consiste en una glicoproteína constituida por 175 residuos de aminoácidos cuyo papel es importante en la morfogénesis y la patogenicidad del virus (Saurabh et al., 2018). Ocho grupos de rotavirus (A-H) han sido caracterizados, donde el grupo A (RVA) es el más comúnmente encontrado en humanos (Bucardo y Nordgren, 2015), siendo los genotipos G1, G2, G3, G4, G9, P[8], P[4] y P[6] los más prevalentes a nivel mundial (Yang et al., 2008). En Colombia, los genotipos más frecuentemente reportados en humanos son G3P8 (33%), G2P4 (26%) y G1P8 (25%) (De la Hoz et al., 2010). Por otro lado, los rotavirus pertenecientes a los grupos A, B, C y E pueden también afectar cerdos, siendo el último detectado únicamente en porcinos (Martella et al., 2010).
Clínicamente, Rotavirus es un patógeno entérico causante de diarrea acuosa aguda en diferentes especies hospederas (Yodmeklin et al., 2017). En el humano, la enfermedad diarreica aguda (EDA) es considerada como la causante de más de 400 000 muertes y hospitalizaciones por año en niños menores de cinco años a nivel mundial (WHO, 2017). Si bien, dicha enfermedad se asocia con diferentes agentes causales, los virus son los agentes etiológicos de mayor distribución, ocasionando entre el 70 al 80% de casos en niños dentro del grupo de riesgo, siendo rotavirus el más representativo de ellos (Blanco y Reyes, 2015).
A pesar de que la información sobre la epidemiología o la diversidad antigénica de RVA en cerdos es escasa, se le ha detectado en animales entre los 7 y 60 días generalmente, con alta prevalencia entre las 3 y 5 semanas de edad. A su vez, se destacan los genotipos de mayor frecuencia G3-G5, G9 y G11 combinado con P[5]-[7], P[13], P[23], P[27] y P[34] (Vidal et al., 2018). Aunque los virus pertenecientes a este grupo son generalmente aceptados como patógenos específicos de especie, la transmisión interespecie ha sido reportada. Por ejemplo, algunas cepas de RVA pueden ser detectadas en humanos y cerdos, producto de la transmisión del virus de origen animal al humano (Midgley et al., 2012; Malasao et al., 2018).
En algunos países de Latinoamérica como Brasil, Argentina y Paraguay, cepas inusuales de rotavirus G5 han sido detectadas en niños, comprobándose mediante técnicas moleculares, que son producto de un reordenamiento entre virus humanos y porcinos (Martella et al., 2010), de allí que la determinación indirecta de la presencia del virus en granjas porcícolas permite realizar ajustes a los programas de control y prevención, con el fin de evitar los efectos negativos para la producción, derivados de las pérdidas económicas asociadas a la disminución de los índices zootécnicos así como a las posibles implicaciones en salud pública.
MATERIALES Y MÉTODOS
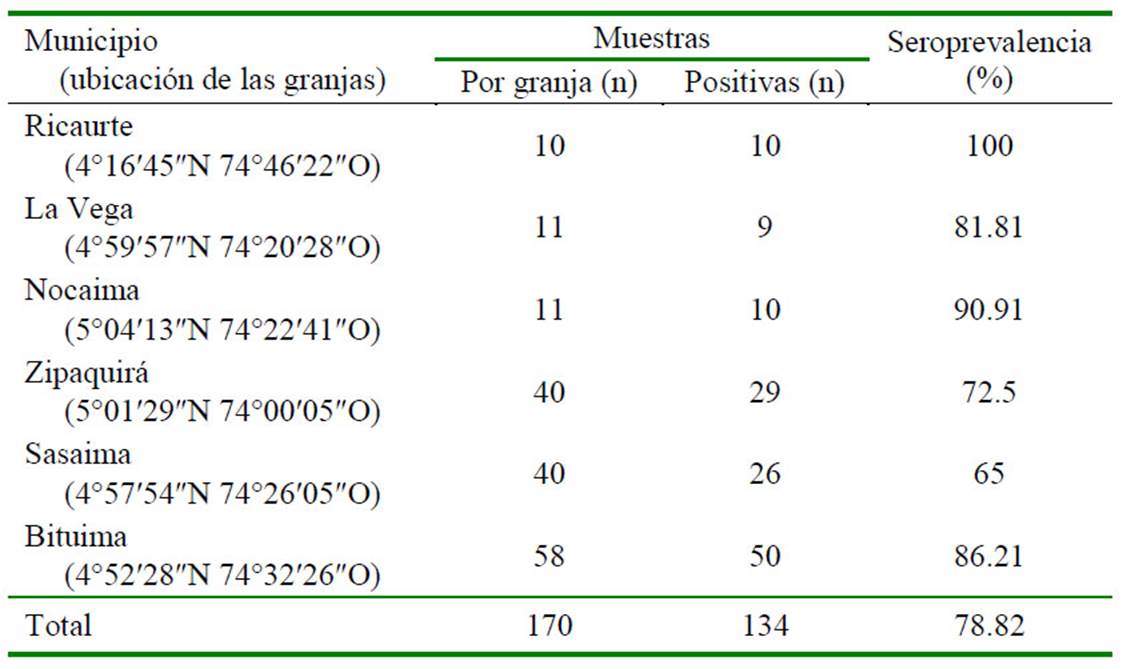
Con base a la participación voluntaria de los propietarios de las granjas porcícolas, se realizó un muestreo por conveniencia, colectándose al azar 170 muestras sanguíneas de porcinos clínicamente sanos, distribuidos en seis granjas semitecnificadas de ciclo completo. Estas granjas tienen menos de 400 hembras de cría y sus planes vacunales y sanitarios se manejaban de acuerdo con su situación y según criterio técnico-clínico veterinario. Estaban ubicadas en los municipios Ricaurte, La Vega, Nocaima, Bituima, Zipaquirá y Sasaima, del departamento de Cundinamarca, Colombia.
Las muestras de sangre fueron colectadas de la vena yugular en tubos vacutainer sin anticoagulante (Framstad et al., 2000; Casas, 2013). Las muestras fueron refrigeradas y transportadas en nevera de poliestireno expandido ("icopor") hasta los laboratorios de la Pontificia Universidad Javeriana. Las muestras fueron centrifugadas a 1500 g por 5 minutos, para la obtención del suero. Los sueros fueron procesados mediante la técnica de ELISA indirecta utilizando el estuche Ingezim Rotavirus Porcino (Ingenasa(r)), siguiendo las indicaciones de la casa comercial. Esta prueba permite detectar anticuerpos específicos frente a Rotavirus Tipo A. La sensibilidad y especificidad reportada para este estuche comercial es de 97%.
La interpretación de resultados para las pruebas de ELISA se realizó de acuerdo con las indicaciones de la casa productora, teniendo en cuenta las densidades ópticas (DO) tanto de los controles como de las muestras. Así, DO Control Positivo a 1/200: >0.7; DO Control Negativo a 1/200: <0.4; Punto de corte: 0.4, donde muestras con valores superiores al punto de corte se consideraron positivas. La seroprevalencia (SP) fue calculada teniendo en cuenta la siguiente fórmula: SP = (Total de sueros positivos / Total de muestras) * 100.
RESULTADOS
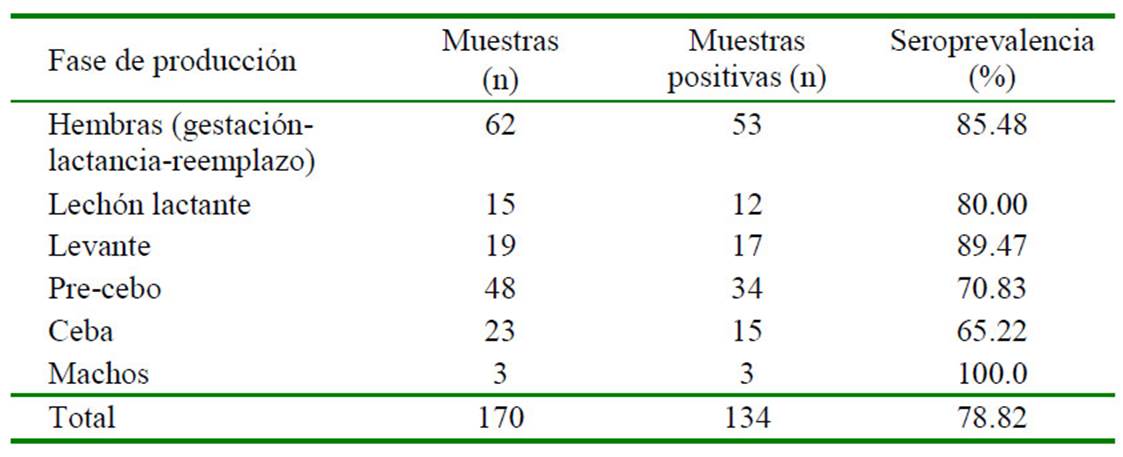
El 78.82% (n=134) de las 170 muestras analizadas fueron positivas, encontrándose una mayor seroprevalencia en los machos, mientras los valores más bajos correspondieron a cerdos en la etapa de ceba (Cuadro 1). Los resultados de seropositividad de acuerdo con la granja estuvieron en un rango de 65100% (Cuadro 2).
Cuadro 1 Seroprevalencia de rotavirus por grupo etario en granjas porcinas de ciclo completo del departamento de Cundinamarca, Colombia
DISCUSIÓN
La determinación serológica de la presencia de rotavirus en granjas porcícolas no es una práctica rutinaria, dado que no es un patógeno de reporte obligatorio. Aun así, en el presente estudio la seroprevalencia general fue de 78.82% y, aunque los reportes serológicos son escasos, los resultados obtenidos podrían ser compatibles con la prevalencia del 71.5% reportada en Brasil con la técnica de RT-PCR (Médici et al., 2011); sin embargo, también por pruebas moleculares, se reportan prevalencias más bajas, en varios países europeos como Hungría, Dinamarca, España y Eslovenia (4.2, 10, 16 y 20%, respectivamente), según Midgley et al. (2012), aunque sin hacer mención de los tipos de granja o a las medidas de bioseguridad instauradas en ellas.
Los Rotavirus se han asociado a procesos de enteritis y diarreas agudas en lechones lactantes y en destetos, e incluso en hembras lactantes (Médici et al., 2011; Vlasova et al., 2017); sin embargo, este virus también ha sido detectado en animales asintomáticos de diferentes edades (Miyazaki et al., 2013), lo que coincide con lo encontrado en el presente estudio en el que se evaluaron animales sin sintomatología gastrointestinal, a lo largo de la cadena primaria. Esta situación, no obstante, podría estar asociada con la inmunidad adquirida a través del calostro suministrado a los neonatos (Vlasova et al., 2017).
La presencia del rotavirus desencadena pérdidas económicas importantes, pues se reportan índices de mortalidad entre 7 y 20% en lechones lactantes y entre 3 y 15% en destetados (Shao et al., 2016). Por otro lado, el virus puede abrir la puerta a otros patógenos y pueden presentarse infecciones mixtas, por lo que, el monitoreo serológico contra este agente podría permitir la implementación de medidas de prevención y control tendientes al mejoramiento del estado sanitario de los animales.
Si bien, el potencial de transmisión zoonótica está reportado para los serogrupos A y C, la presencia conjunta de los serotipos G2,3,4,9,12 y P6,8 en porcinos y en humanos, hace suponer la posible rotación entre especies hospederas (Martella et al., 2010; Midgley et al., 2012; Vlasova et al., 2017), especialmente a través de consumo de agua y alimentos contaminados (Kittigul et al., 2001). Adicionalmente, la presencia del virus en el agua (residual, de fuentes hídricas naturales, subterránea y potable) se debe a la contaminación de esta con materia fecal de individuos infectados (Kittigul et al. (2001) y que podrían entrar en contacto con otras cepas de rotavirus provenientes de diferentes especies hospederas, lo que implica un riesgo, pues el virus posee diferentes mecanismos para su reordenamiento genético (Martella et al., 2010). Se ha establecido que partículas de rotavirus reordenados, de origen humano y porcino, han sido capaces de infectar de forma exitosa poblaciones humanas de Latinoamérica (Alfieri et al., 1996), India (Varghese et al., 2003), Tailandia (Okada et al., 2000) e Italia (Martella et al., 2008).
CONCLUSIONES
La presencia de anticuerpos contra rotavirus detectada en varios grupos etarios de granjas porcícolas de Cundinamarca, Colombia, sugiere que los animales evaluados pudieron estar en contacto con el virus en algún momento de su ciclo productivo, lo que no excluye la posibilidad de que los anticuerpos en cerdos lactantes y destetados se deriven de la inmunidad lactogénica transmitida por las cerdas












 uBio
uBio