INTRODUCCIÓN
Gallibacterium sp es un microorganismo patógeno primario en aves, de características Gram negativos, inmóviles, encapsulados, no esporulados, pleomórficos, mesófilos y anaerobios facultativos o microaerófilos. Pertenece al orden Pasteurellales, familia Pasteurellaceae y el género presenta tres especies: G. anatis y G. genomospecies 1 y 2. Se pueden identificar microbiológicamente por medio de cultivo, así como molecularmente mediante PCR (Bojesen et al., 2007).
Esta bacteria es altamente variable, ya que actualmente se describen 24 biovares por pruebas bioquímicas utilizando diferentes azucares (Osuna et al., 2017). Las bacterias altamente variables sufren frecuentemente nuevos cambios genéticos, ya sea por mutaciones debido a su adaptación al medio en que se desarrollan o como consecuencia del intercambio de material genético entre los mismos (Mendoza et al., 2014). Además, el abuso de antibióticos acelera las mutaciones genéticas, adaptándose a los nuevos fármacos (Mendoza et al., 2014).
Existen diversos métodos para identificar la variabilidad en Gallibacterium sp. Entre estos se tienen los ensayos bioquímicos, la susceptibilidad antimicrobiana, y las pruebas moleculares (Singh et al., 2015). Entre los métodos moleculares para tipificar bacterias, se ha encontrado que las huellas genómicas de REP-PCR, ERIC-PCR y BOX-PCR (denominadas colectivamente como rep-PCR) son extremadamente confiables, reproducibles, rápidas y altamente discriminatorias. Este método se basa en la amplificación de las secuencias de ADN mediante PCR con conjuntos de cebadores complementarios a las secuencias de ADN repetitivas naturales, altamente conservadas, presentes en múltiples copias en distintas posiciones intergénicas en los genomas de la mayoría de las bacterias Gram negativas y varias Gram positivas (Oliveira et al., 2007)
El objetivo del presente trabajo fue tipificar las cepas de Gallibacterium sp aisladas de aves comerciales con infecciones respiratorias, así como la elaboración de programas efectivos de sanidad aviar en los cuales se consideren cepas endémicas de los principales departamentos de producción avícola de Perú.
MATERIALES Y MÉTODOS
Material Biológico
Se trabajó con 60 cepas de Gallibacterium sp aisladas de muestras de pollos (BB y de carne), gallinas de postura y gallinas reproductoras con enfermedades respiratorias provenientes de granjas de los departamentos de La Libertad, Ica y Lima (Perú) entre 2014 y 2018. Las cepas se encontraban liofilizadas y guardadas en glicerol a -20 ºC y se tenía registrada la historia clínica del caso y el análisis anatomopatológico (Mendoza et al., 2014).
Análisis Microbiológico
Las aves fueron sacrificadas, se incidió en las barbillas y senos infraorbitarios, se hisoparon las secreciones nasales y tráquea. Asimismo, se ingresó en las cavidades torácica y abdominal en donde se hisoparon pulmones y sacos aéreos, y se hicieron improntas de hígado y ovario. Luego los hisopados e improntas fueron sembrados en placas de agar sangre de ovino al 5 % (las cuales fueron introducidas en campanas de microaerobiosis) y agar MacConkey. Finalmente se incubaron a 37 ºC durante 24-48 horas (Mendoza et al., 2014).
Sensibilidad Antimicrobiana
Se realizó un perfil de resistencia antimicrobiana para seis familias de antibióticos de importancia veterinaria, mediante el método de difusión en agar sangre al 5% con nueve discos de sensibilidad: Oxitetraciclina 20 µg, Florfenicol 30 µg, Fosfomicina 50 µg, Amoxicilina 20 µg, Ciprofloxacina 5 µg, Enrofloxacina 5 µg, Doxiciclina 30 µg, Norfloxacina 10 µg, Sulfatrimetropin 1.25/23.75 µg. La placa preparada con el inóculo y los discos de antibióticos se incubó a 37 ºC durante 24 a 48 horas. Los resultados se leyeron midiendo el diámetro de los halos de inhibición del crecimiento, interpretándose como sensible, intermedio o resistente, según los lineamientos del Comité Nacional de Estándares Clínicos de Laboratorio (NCCLS, 2002).
Identificación y Tipificación Molecular
Extracción de ADN
El ADN de las 60 cepas de Gallibacterium sp fue extraído mediante el método de membranas de sílica gel utilizando el GF-1 Tissue DNA Extraction kit (Vivantis, Malasia), siguiendo las indicaciones del protocolo de extracción del fabricante.
Detección molecular por PCR
Gallibacterium sp fue detectado mediante amplificación por PCR, a partir de la secuencia de los genes ribosómicos 16S y 23S. Se utilizaron los cebadores forward: 1133fgal -5´ -TAT TCT TTG TTA CCA CCG G -3´ y el reverse: 114r -5´ -GGT TTC CCC ATT CGG -3´. La reacción se realizó en un volumen final de 20 µl conteniendo 10 µl del buffer 2X HOT STAR PCR, 1 µl de ambos cebadores a 10 pmol/µl y 3 µl de ADN genómico a 5 ng/µl. Los ciclos termales fueron: 1 ciclo de desnaturalización inicial: 95 ºC por 4 min; 30 ciclos de desnaturalización a 95 ºC por 30 s; alineamiento a 55 ºC por 30 s y extensión a 72 ºC por 45 s; 1 ciclo de extensión final a 72 ºC por 5 min, realizados en un termocicladorVeriti Thermal Cycler (Life Technologies) (Bojesen et al., 2007b; Ataei et al., 2017).
Los productos de PCR para su análisis se corrieron en una cámara electroforética, cargando 3 µl de producto de PCR mezclado con 2 µl de Fluorescent DNA Loading dye en un gel de agarosa al 1% en buffer TBE 1X (Tris 0.89 M,Borato0.02M, EDTA0.89 M, pH 8.3) por 40 min a 100 v y luego visualizados bajo UV en fotodocumentador Major Science.
Tipificación molecular por ERIC-PCR
Genotipificación por Enterobacterial Repetitive Intergenic Consensus -PCR (ERIC-PCR) se realizó utilizando los cebadores: ERIC1R: 5´-ATGTAAGCTCCTGGGGATTCAC-3´ y ERIC2: 5´-AAGTAAGTGACTGGGGTGAGCG-3´) que amplifican regiones repetitivas VNTRS. El volumen final de la amplificación fue 20 µl, utilizando las concentraciones de 10 X Buffer PCR, 25 mM de dNTPs, 25 mM de MgCl2, 10 pmol de cada cebador, 1 U Taq DNA polimerasa (Fermentas) y 4 µl de 5 ng/µl de ADN genómico. Los ciclos termales utilizados fueron: 1 ciclo desnaturalización inicial de95 ºCpor4min;35ciclos:desnatu-ralización a 94 ºC por 30 s; alineamiento a 40 ºC por 3 min y extensión a 72 ºC por 2 min, y finalmente un ciclo de extensión final de 72 ºC por 7 min. Los productos de PCR de ERICPCR fueron separados mediante electroforesis en gel de agarosa al 2% en buffer TBE 1X durante 50 min a 100 v y visualizados bajo luz UV (Oliveira et al., 2007; Mendoza et al., 2014).
Análisis de patrones VNTRS para ERICPCR
Los patrones de ERIC-PCR en el gel de agarosa fueron transformados a una matriz bidimensional de ausencia (0) o presencia (1) generados por el programa Gel-Analyzer v. 2010. El análisis de la variabilidad genética intra e interespecífica para las 60 cepas de Gallibacterium sp fue realizada utilizando similarity index de Jaccard con un bootstraps de 1000 réplicas y dendogramas generados en base a una matriz de distancia genética mediante el método clásico paired groupUPGMAaplicando el programaPAST 3.23 (Hammer et al., 2001).
Tipificación molecular por REP-PCR y BOXAIR
El Repetitive Extragenic Palindromic PCR (REP-PCR) se realizó con los cebadores REP 1 (5'-IIIGCGCCGICATCAGGC-3') y REP 2 (5'-ACGTCTTATCAGGCCTAC-3'). La reacción de PCR se realizó en un volumen final de 20 µl, conteniendo 2.5 µl a 25 pmol de cada cebador, buffer de PCR 1X (tris 10 mM, KCl 50 mM; pH 8.3), dNTPs 0.2 mM, MgCl2 2.5 mM, 1U de ADN polimerasa Taq (Fermentas) y 4 µl de ADN de plantilla a 5 ng/µl. La amplificación se desarrolló en un termociclador Veriti (Applied Biosystems) con las condiciones termales siguientes: un ciclo de desnaturalización inicial a 95 ºC durante 5 min; 30 ciclos: desnaturalización a 90 ºC durante 30 s, primer alineamiento a 50 ºC durante 30 s, segundo alineamiento a 52 ºC durante 1 min y extensión a 72 ºC durante 1 min; y un ciclo de extensión final a 72 ºC durante 8 min. Condiciones similares a lodescrito por Oliveira et al. (2007) y Albufera et al. (2009).
El BOXAIR-PCR fingerprinting para enterobacterias se realizó con el cebador 5' CTACGGCAAGGCGACGCTGACG-3 y las concentraciones en un volumen final de 20 µl fueron: 2 µl del cebador BOXAIR a 20 pmol, buffer de PCR 1X (tris 10 mM, KCl 50 mM; pH 8.3), dNTPs 0.2 mM, MgCl2 2.5 mM, 1U de ADN polimerasa Taq (Fermentas) y 4 µl de ADN de plantilla a 5 ng/µl. Los ciclos termales para BOXAIR fueron un ciclo de desnaturalización inicial a 95 ºC durante 4 min, 30 ciclos: desnaturalización a 94 ºC durante 30 s, alineamiento a 50 ºC durante 60 s, extensión a 65 ºC durante 1 min y un ciclo de extensión final a 65 ºC durante 8 min. La amplificación se realizó en un termociclador Veriti Thermal Cycler (Life Technologies).
Los productos de PCR de REP Y BOXAIR fueron separados mediante electroforesis en gel de agarosa al 2% en buffer TBE 1X durante 50 min a 100 v y visualizados bajo luz UV (Oliveira et al., 2007; Albufera et al., 2009).
Análisis de patrones VNTRS para REPPCR y BOX A1R
El análisis de las huellas dactilares genéticas (ADN fingerprinting) de REP-PCR y BOX A1R fue realizado a partir de los patrones de bandeos transformados en matriz bidimensional de ausencia (0) o presencia (1) generados por el programa GelAnalyzer v. 2010, aplicando una matriz de (dis)similaridad utilizando el coeficiente de JACCARD y el desarrollo del dendograma utilizando el método clásico de distancia genética paired group UPGMA mediante el programa PAST 3.23 (Hammer et al., 2001).
Poder discriminatorio de los métodos de tipificación
El poder discriminatorio se midió mediante el índice de diversidad (1-D) de Simpson cuyo valor oscila entre 0 y 1, que indica la probabilidad promedio de que un sistema de tipificación asigne un tipo diferente a dos cepas no relacionadas muestreadas al azar de una población o representa la probabilidad de que dos individuos de la misma especie seleccionados aleatoriamente de una muestra presenten diversidad genética. Para la interpretación del índice, cuanto mayor es el valor, mayor es la diversidad genética de la muestra (Oliveira et al., 2007).
RESULTADOS
El 100% de los aislados (n=60) fueron similares a las características microbiológicas en agar sangre de ovino al 5% y al perfil bioquímico compatible a Gallibacterium sp. Además, todas las cepas se confirmaron mediante reacción de la cadena polimerasa (PCR), amplificando los genes ribosómicos 16S y 23S para Gallibacterium sp (790 bp) y para Gallibacterium anatis (1080 bp) o Gallibacterium genomospecies 1 y 2 (1030 bp) (Figura 1).
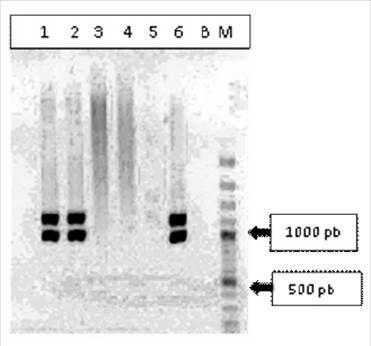
Figura 1 Detección de Gallibacterium spp por PCR del gen 16S rRNA. Carril 1-2: ADN obtenido de cepa de Gallibacterium spp; Carril 3-4: muestras negativas; Carril 5. Control negativo, ADN de Pasteurella multicida ATCC® 12945; Carril 6. Control positivo, ADN de G. anatis ATCC® 542; B: Blanco de PCR; M, Marcador de peso molecular GeneRuler 100 bp Plus DNA Ladder (Thermo Scientific)
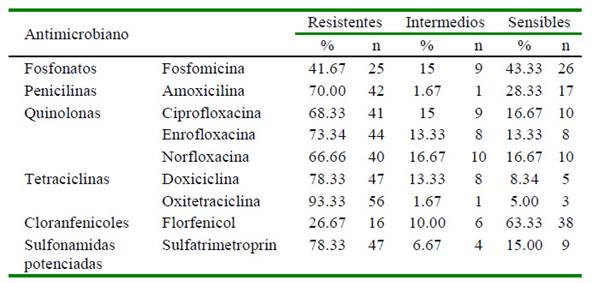
Los aislados mostraron una elevada multirresistencia a antibióticos. El 98.3% de los aislados fueron resistentes al menos a una o más familias de antibióticos y el 11.7% de los aislados fueron multidrogo resistentes (resistencia a las seis familias de antibióticos). Los aislados mostraron mayores perfiles de resistencia a las tetraciclinas: Oxitetraciclina 93.3% (56/60), Sulfonamidas potenciadas: Sulfatrimetropin 78.3% (47/60) y Quinolonas: Enrofloxacina 73.3% (44/60), mientras que la mayor sensibilidad fue para los Fosfonatos: Fosfomicina 43.3% (26/60) (Cuadro 1).
Cuadro 1 Perfiles de resistencia a nueve antibióticos en cepas aisladas de Gallibacterium anatis en aves comerciales del Perú con infecciones respiratorias
Fingerprinting ERIC-PCR
De las 60 cepas de Gallibacterium sp se obtuvieron 44 perfiles genéticos codificados de E1 a E44 (Cuadros 2 y 3). Los tamaños de productos amplificados presentaban rangos entre 300 y 3000 bp, formando un grupo general de 17 bandas: 300, 350, 400, 500, 600, 650, 700, 800, 900, 1000, 1200, 1400, 1500, 2000, 2250, 2500 y 3000 bp. El número de bandas por perfil variaba de 6 a 12, distribuidos en la totalidad de muestras, sin presentar patrones de bandeos conservados, lo que podría sugerir presencia de una gran diversidad clonal de Gallibacterium sp. El índice de diversidad Simpson (1-D) en este método de tipificación varió entre 0.76 y 0.97 aplicando el programa estadístico PAST 3.23, indicando que existe diversidad clonal (Figura 2).
Cuadro 2 Genotipificación y multidrogoresistencia de 60 cepas de Gallibacterium sp mediante REP, ERIC y BOXAIR, según el año de aislamiento, sistema de producción, órgano y localidad (Parte I)
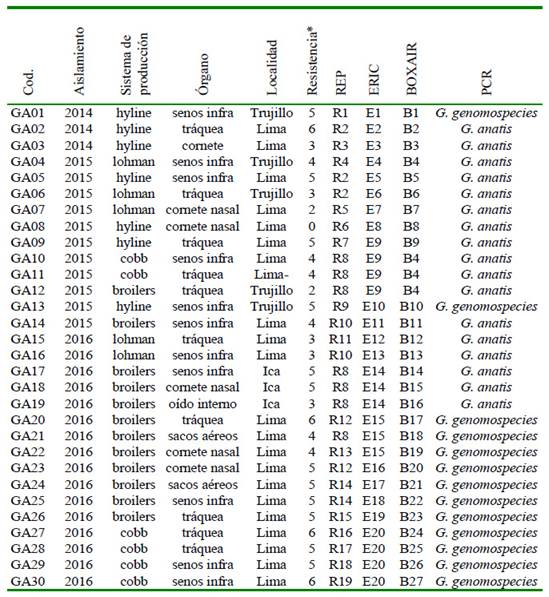
* Seis familias de antibióticos: (1) cloranfenicoles, (2) penicilinas, (3) fosfonatos, (4) quinolonas, (5) tetraciclinas, (6) sulfamidas potenciadas
Cuadro 3 Genotipificación y multidrogoresistencia de 60 cepas de Gallibacterium sp mediante REP, ERIC y BOXAIR, según el año de aislamiento, sistema de producción, órgano y localidad (Parte II)
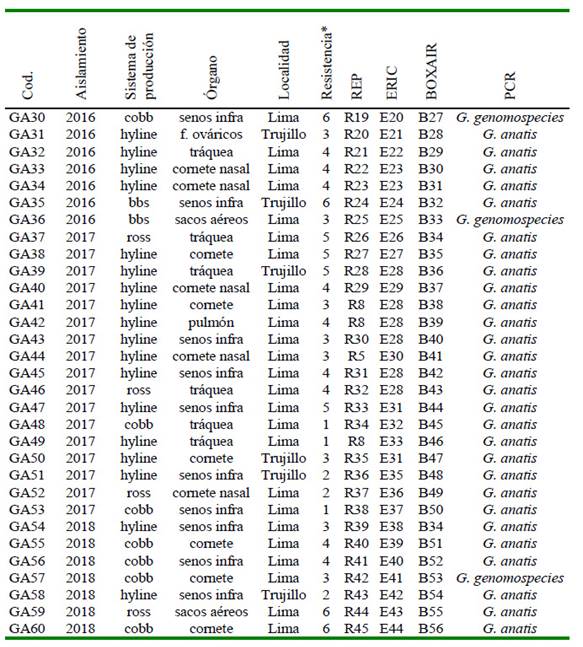
* Seis familias de antibióticos: (1) cloranfenicoles, (2) penicilinas, (3) fosfonatos, (4) quinolonas, (5) tetraciclinas, (6) sulfamidas potenciadas
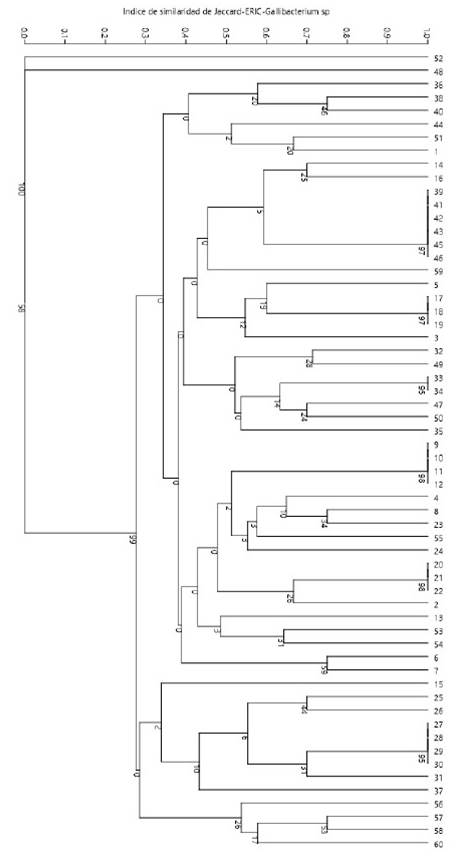
Figura 2 Enterobacterial Repetitive Intergenic Consensus-PCR (ERIC-PCR). Árbol filogenético de Gallibacterium sp mediante el algoritmo paired group de UPGMA obtenido a partir de una matriz bidimensional de ausencia o presencia obtenido a partir de GelAnalyzer v. 2010, aplicando el programa estadístico PAST 3.23 con un similarity index de Jaccard y un bootstraps de 1000 réplicas
Repetitive Extragenic Palindromic (REPPCR)
El ensayo de amplificación de los cebadores REP-1R y REP-2 mostró 45 patrones genéticos (clones) codificados como R1-R45 (Cuadros 2 y 3). Los tamaños de productos amplificados presentaban los rangos de 300 aa 3000 bp, formando un grupo general de 13 bandas: 300, 350, 400, 500, 700, 800, 900, 1000, 1250, 1600, 2000, 2600 y3000 bp (patrones). El número de bandas por perfil variaba de 2 a 7, distribuidos en la totalidad de muestras, sin presentar patrones de bandeos conservados, lo que podría sugerir presencia de una gran diversidad clonal de Gallibacterium sp. El índice de diversidad Simpson (1-D) en este método de tipificación oscila entre 0.61y 0.97 aplicando el programa estadístico PAST 3.23, indicando que existe diversidad clonal (Figura 3).
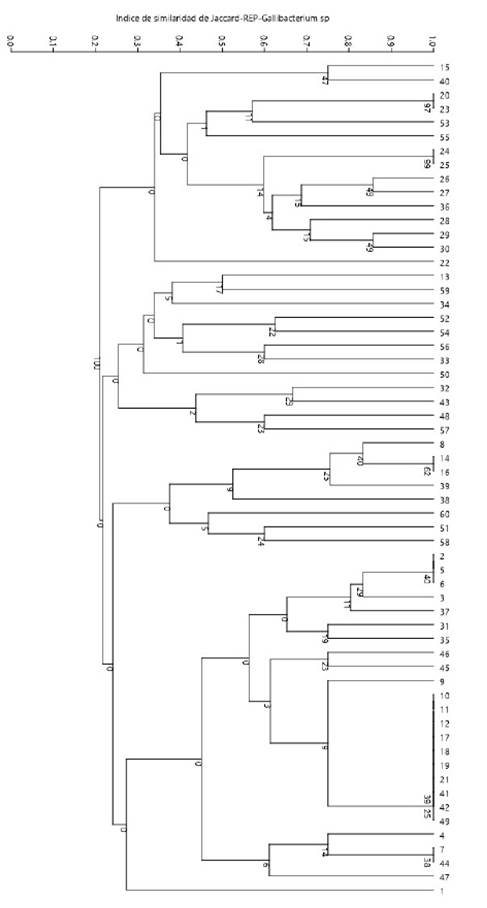
Figura 3 Repetitive extragenic palindromic (REP-PCR). Árbol filogenético mediante el algoritmo paired group de UPGMA obtenido a partir de una matriz bidimensional de ausencia o presencia por GelAnalyzer v. 2010. DNA fingerprinting (huella genética de ADN) de cepas de Gallibacterium sp aplicando el programa estadístico PAST 3.23 con un similarity index de Jaccard y un bootstraps de 1000
BOXAIR-PCR Fingerprinting Enterobacterias
En el ensayo de amplificación del cebador BOXAIR se obtuvieron 56 patrones genéticos (clones) codificados como B1-B56 (Cuadros 2 y 3). Los tamaños de productos amplificados presentaban los rangos de 200 a 3000 bp formando un grupo general de 12 bandas: 200, 300, 400, 600, 700, 800, 1000, 1250, 1400, 1700, 2200 y 3000 bp (patrones). El número de bandas por perfil variaba de 3 a 11 distribuidos en la totalidad de muestras, sin presentar patrones de bandeos conservados, lo que podría sugerir presencia de una gran diversidad clonal de Gallibacterium sp. El índice de diversidad Simpson (1-D) en este método de tipificación varió entre 0.5 y 0.97 aplicando el programa estadístico PAST 3.23, indicando que existe moderada diversidad clonal (Figura 4).
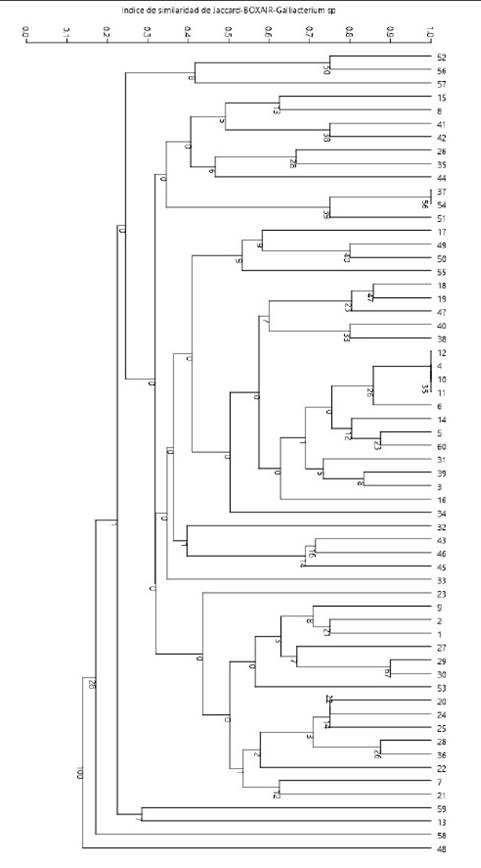
Figura 4 BOXAIR-PCR fingerprinting enterobacterias. Árbol filogenético mediante el algoritmo clásico paired group de UPGMA obtenido a partir de una matriz bidimensional de ausencia o presencia por GelAnalyzer v. 2010. DNA fingerprinting (huella dactilar genética de ADN) de Gallibacterium sp aplicando el programa estadístico PAST 3.23 con un similarity index de Jaccard y un bootstraps de 1000 réplicas
DISCUSIÓN
La avicultura en el Perú está siendo afectada por diversos patógenos bacterianos con nuevas características genéticas, afectando considerablemente la producción de huevos y carne, causando gran mortandad de aves si se asocian con otros microorganismos (Bojesen et al., 2011a). Bacterias altamente variables como Gallibacterium sp son un ejemplo de las sufren frecuentes cambios genéticos (Mendoza et al., 2014).
Diversas investigaciones muestras a Gallibacterium sp como un patógeno multidrogo resistente a diversos antimicrobianos. Los estudios de Bojesen et al., 2011b indicaron resistencia de G. anatis a más de tres antibióticos en el 65% de las cepas de campo; por otro lado, Mendoza et al. (2014) encontraron que 96 cepas de G. anatis mostraron multirresistencia, tres fueron resistentes a todos los antibióticos estudiados y, el 98% presentó resistencia a la sulfamida, resultado similar a los hallazgos de Bojesen et al. (2011a). En el presente estudio, 59/60 cepas mostraron resistencia a por lo menos 1 o más familias de antibióticos, 7/60 fueron multidrogo resistentes a las seis familias del ensayo. Los aislados con mayores perfiles de resistencia fueron las tetraciclinas (Oxitetraciclina 93.33%, 56/60) y las sulfonamidas potenciadas (Sulfatrimetropin 78.33%, 47/60), similar al trabajo de Bojensen et al. (2011b). La disminución a la sensibilidad antimicrobiana puede deberse al uso inadecuado de estos, lo cual constituye un riesgo para el control de la bacteria, dando indicios de presencia activa de variabilidad genética continua en Gallibacterium sp (Martina et al., 2016)
Estudios de variabilidad genética de G. anatis han sido descritos en varios países de Europa así como en Perú (Bojesen et al., 2003a,b,c; Christensen et al., 2003, 2004; Bojesen y Shivaprasad, 2007a; Bojesen et al., 2007b; Christensen y Bisgaard, 2010; Mendoza et al., 2014), donde encuentran 24 biovares: la subespecie haemolytica comprende los biovares 1-4, 6, 7, 10-24; la subespecie anatis el biovar Pa; genomospecies 1 el biovar 5 y 8; y la genomoespecie 2 el biovar 9. De estos, Pa es el más frecuente, seguido de los biovares hemolíticos 1, 3, 4, 12 y 18 (Mendoza et al., 2014). Estos estudios manifiestan la gran actividad genética presente en Gallibacterium sp, exigiendo el desarrollo y uso de metodologías alternativas a las convencionales que permitan identificar en forma oportuna el agente infeccioso mutante para el control y la producción de autovacunas efectivas.
En el Perú no existe información sobre la variabilidad genotípica en cepas de Gallibacterium sp basadas en amplificación patrones VNTRS mediante REP, ERIC Y BOXAIR, con excepción del estudio de Mendoza et al. (2014), utilizando ERIC-PCR en los aislamientos de G. anatis. El método demostró ser altamente discriminatorio y útil para la evaluación epidemiológica, logrando identificar 24 genotipos en las 96 cepas. La aplicación de las tres técnicas de tipificación demostraron ser extremadamente confiables, reproducibles, rápidas y altamente discriminatorias en otros países como Brasil y EEUU, logrando tipificar aislados de Salmonella enteritidis, especialmente con los basados en la VNTRS (Versalovic et al., 1994; Oliveira et al., 2007). En forma similar, en el presente estudio la PCR fingerprinting utilizando los tres cebadores mostró distintos perfiles o patrones de bandeos en la mayoría de los aislamientos.
La REP-PCR presentó 45 perfiles (R1R45) determinando 45 genotipos, además de 4 clados. Además, se determinaron seis grupos con perfiles genéticos idénticos circulantes en Lima, Libertad e Ica. La ERIC-PCR presentó 44 perfiles (E1-E44) determinando 44 genotipos, 5 clados, y 6 grupos con perfiles genéticos idénticos dispersos en Lima, Libertad e Ica. La BOXAIR presentó 56 perfiles (B1-B56) determinando 56 genotipos, 8 clados, y 2 grupos con perfiles genéticos idénticos dispersos en Lima y Libertad. Las tres técnicas de genotipificación podrían dar pistas sobre una alta variabilidad existente en Gallibacterium spp, lo que sugiere su origen común, pero hay que tener en cuenta la presencia de dos especies dentro del estudio: G. anatis y G. genomospecies. Los resultados demuestran que existe una diversidad genética en el linaje clonal en la mayoría de los aislados de las dos especies, además de actividad genética y variabilidad intra e interespecífica en las cepas de Gallibacterium sp. Este hallazgo implica que se podría influir o incrementar la capacidad patógena por cada genotipo nuevo (Bojesen y Shivaprasad, 2007a; Neubauer et al., 2009).
Los perfiles de DNA detectados por las técnicas de REP y ERIC demuestran moderado nivel discriminatorio y los perfiles de DNA de BOXAIR demostraron ser altamente discriminatorios al determinar 56/60 genotipos. Por otro lado, no se pudo rastrear si la diversidad clonal presente estaba correlacionada según el órgano de aislamiento, año o lugar de aislamiento; sin embargo, si hubo presencia de correlación en la mayoría de los genotipos, con el aumento de la resistencia antimicrobiana, en por lo menos de una familia de antibióticos. La aplicación de los tres métodos de tipificación para las 60 muestras de Gallibacterium sp en los aislados de aves mostraron perfiles únicos, lo que sugiere que la fuente bacteriana originaria de la diversidad clonal es la misma especie y también la interacción o asociación con otras bacterias estrechamente relacionadas (Paudel et al., 2017; Wang et al., 2018).
CONCLUSIONES
La rep-PCR (REP, ERIC y BOXAIR) realizada como se describe en el presente estudio fue útil para la tipificación de Gallibacterium sp, dado que fue capaz de discriminar aislamientos potencialmente diferentes de las ciudades de Lima, Libertad y Ica. Sin embargo, el uso combinado de métodos genotípicos y fenotípicos permitió una discriminación más precisa entre aislamientos.












 uBio
uBio 

