INTRODUCCIÓN
La producción ovina en Colombia tradicionalmente se ha desarrollado en condiciones extensivas que corresponden principalmente a sistemas de producción campesina y economías de subsistencia (Espinal et al., 2006). El censo del Instituto Colombiano Agropecuario (ICA, 2018) presenta un inventario nacional estimado de 1 578 684 cabezas, distribuidas principalmente en los departamentos de La Guajira (42.4%), Magdalena (10.9%), Cesar (7.9%), Córdoba (7.1%) y Boyacá (6.7%), los cuales representan el 75% de la producción ovina en Colombia. Los rebaños están formados en su mayoría por ovinos criollos (80-85%), además de 10-15% de mestizos y cerca de 5% de ejemplares de razas puras introducidas al país (Grajales y Tovio, 2010). Es así, que la base genética ovina está constituida principalmente por animales criollos de lana, de ascendencia europea, y animales criollos de pelo, de ascendencia africana.
El tipo criollo es el producto del cruzamiento de varias razas que fueron introducidas al país durante la época de la conquista. Entre estas razas destacan la Churra, la Manchega y la Lacha, las cuales poblaron la zona andina montañosa. Tras siglos de cruzamientos, el ovino criollo adquirió características de rusticidad, prolificidad y resistencia a parásitos externos e internos, debido a su adaptación a las condiciones adversas del trópico colombiano (Martínez et al., 2009). El manejo genético que se le ha dado a las poblaciones ovinas criollas ha llevado al incremento de su homocigocidad y, por tanto, a la pérdida de su productividad (Ocampo, 2014).
Fenotípicamente se pueden identificar tres tipos: el Sudán o amarillo que se caracteriza por su constitución maciza, de buen tamaño, rústico en extremo y buen productor de carne, de pelaje amarrillo al bayo, cabeza ancha y larga, sin cuernos con pelo corto fino y adherente; el etíope o rojo , con pelaje de ese color pudiendo llegar al negro, cabeza ancha, redondeada y sin cuernos; y el abisinio, de conformación cárnica, de gran tamaño y buena conformación muscular, de cabeza y orejas de fuerte pigmentación (Vivas, 2013).
El uso adecuado de la biodiversidad de los animales domésticos en los diferentes sistemas de producción es parte de las estrategias de vida de las familias en el medio rural (CONABIO, 2011). El manejo genético de las razas autóctonas puede, asimismo, facilitar la seguridad alimentaria en un mundo cada vez más demandante (FAO, 2015). Los estudios en genética molecular utilizando marcadores moleculares vienen permitiendo el estudio de los orígenes de las especies y determinar la distribución geográfica de su diversidad. Los polimorfismos de ADN determinan los marcadores más convenientes para realizar estudios moleculares sobre la diversidad genética (Vivas, 2013). Así, en el sector ovino se han desarrollado diversos estudios, donde los microsatélites han probado ser una herramienta exitosa para evidenciar una posible diferenciación genética entre poblaciones (Quiroz et al., 2007). Sin embargo, gracias a la rápida evolución de la genómica en animales de granja han surgido nuevas metodologías que pueden generar una descripción global de los sistemas biológicos con mayor precisión y eficiencia (Mainero, 2015).
Figura 1 Ubicación geográfica de las poblaciones ovinas. Triángulos rojos: Valles Interandinos del Río Magdalena (departamentos Tolima y Caldas); círculos azules: Piedemonte Llanero (departamentos Meta y Caquetá)
Los SNPs (polimorfismos de nucleótido simple) son marcadores moleculares que identifican el cambio de un nucleótido por otro sobre la secuencia de ADN de un organismo. Los SNPs pueden identificar miles de cambios en las secuencias de ADN de varios animales, lo que los hace altamente eficientes entre los marcadores moleculares, habiéndose utilizado en diversos programas de conservación y de mejora genética (Barrero, 2005). El presente estudio tuvo como objetivo caracterizar genéticamente las poblaciones de Ovino Criollo de Pelo Colombiano mediante el uso del microarreglo de ADN OvineSNP50 BeadChip de Illumina®.
MATERIALES Y MÉTODOS
Material Biológico
Se recolectaron muestras de tejido biológico de 160 individuos de la raza Ovino Criollo de Pelo Colombiano (OCPC) provenientes de las zonas Piedemonte Llanero (PDML), ubicados en los departamentos de Meta y Caquetá (n=121); y de la zona de los Valles Interandinos del Rio Magdalena (VIRM), ubicados en los departamentos de Tolima y Caldas (n=39), Colombia. Los ovinos fueron escogidos al azar para disminuir el sesgo de consanguinidad (Figura 1).
ADN y Genotipificación por SNPs
Los animales fueron enviados a una planta de beneficio local para la venta de la carcasa. Luego del sacrificio se tomaron muestras del músculo Longissimus dorsi para la extracción de ADN. Para esto, se utilizaron 0.02 g de músculo utilizando el protocolo fenol cloroformo isoamílico (Acevedo et al., 2008). El ADN fue cuantificado en un espectrofotómetro Thermo Fisher Scientific (Thermo Fisher Scientific, USA). Se comprobó el producto extraído mediante electroforesis en gel de agarosa al 1%. Para la genotipificación se utilizó el chip OvineSNP50 BeadChip de Illumina® (Illumina, San Diego, USA) siguiendo el protocolo Infinium® Assay Super II Illumina® para su uso en la plataforma HiScan®SQ System (Illumina, San Diego, USA) y escaneo de los BeadChips.
Base de datos y Control de Calidad de los SNPs
Para los análisis estadísticos se generó una base de datos, de la cual se excluyeron los individuos que estaban altamente emparentados (relación >0.25), y aquellos individuos que presentaban un porcentaje mayor al 10% de genotipos perdidos (-mind 0.1). Posteriormente, mediante el software PLINK v. 1.9 (Purcell et al., 2007) se excluyeron aquellos SNPs que tuviesen genotipificados menos del 90% de los individuos (-geno 0.1), que tuviesen el MAF (minor allele frequency) por debajo del 5% (-maf 0.05), que tuviesen la probabilidad menor p<10-6, para la desviación del equilibrio Hardy-Weinberg (-hwe 1e-6) y aquellos que estuviesen en desequilibrio de ligamiento (-indep-pairwise 50 5 0.5). Finalmente, solo los SNPs que estuviesen localizados en cromosomas autosómicos fueron usados para los análisis.
Análisis Estadísticos
Para la detección de las desviaciones del equilibrio Hardy-Weinberg se utilizó el paquete «genepop» del software R (R Development Core Team, 2014) (Rousset, 2008), con una prueba global de 10 000 permutaciones, además de calcular los F-estadísticos mediante los estimadores de Weir y Cockerham (1984). La determinación de las heterocigosidades observadas y esperadas, y el cálculo de los estadísticos F (Fst, Fis y Fit) se realizaron con el paquete «adegenet» (Jombart y Ahmed, 2011) del mismo software. Este paquete fue usado, además, para realizar el análisis de componentes principales (PCA) y un análisis AMOVA (Analysis of Molecular Variance) para determinar la variación dentro de la población y entre subpoblaciones.
RESULTADOS Y DISCUSIÓN
Se excluyeron 49 animales de los 160 muestreados inicialmente por consanguinidad y por al porcentaje de genotipos perdidos por animal (30.6% de individuos excluidos). De los 11 individuos que quedaron, 82 eran del PDML y 29 del VIRM. Posteriormente, se excluyeron 8864 SNPs de los 54 413 SNPs que tiene el chip (16.3%).
La heterocigosidad esperada (He) del OCPC, como única población, fue de 0.374, y para las subpoblaciones PDML y VIRM fue de He=0.357 y He=0.396, respectivamente. La He del OPCC fue más alta que las reportadas para las razas Namaqua Afrikaner (He=0.220) y Merino (He=0.321) por Kijas et al. (2009), posiblemente debido como resultado de sus cruces con varias razas desde la época de la conquista. Por otro lado, debe considerarse que en dicho estudio se utilizó un bajo número de SNPs, con lo que se pudo generar un sesgo en el porcentaje de polimorfismos evidenciados. Por otro lado, Deniskova et al. (2018), utilizando el mismo número de SNPs que en el presente trabajo en 25 razas ovinas en Rusia, encontraron la mayor He para la raza Baikal Fine-fleeced (0.395) y la menor para la raza Kuchugur (0.353), observándose que la población del OCPC se encuentra dentro del rango reportado para las razas rusas, denotando que la raza colombiana presenta una diversidad genética notable. De manera similar, Mastrangelo et al. (2014, 2017) reportan diversidades genéticas (He) para las razas sicilianas que van desde 0.379 a 0.390.
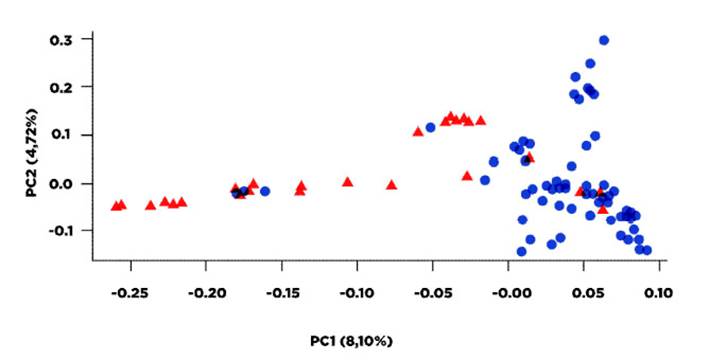
Figura 2 Representación espacial en el análisis factorial de correspondencias en dos dimensiones de los individuos pertenecientes a dos poblaciones ovinas. Triángulos rojos: Valles interandinos del Río Magdalena (departamentos Tolima y Caldas); círculos azules: Piedemonte Llanero (departamentos Meta y Caquetá), Colombia
Los valores para el Fis fueron 0.089 y 0.075 para la población de PDML y VIRM, respectivamente. Estos valores de coeficiente de endogamia (Fis) positivos muestran un exceso de homocigotos para ambas subpoblaciones, con lo que se supone presentan un cierto grado de consanguinidad. El coeficiente de endogamia para el total de la población (Fit) fue de 0.124; es decir, mayor al de las subpoblaciones PDML y VIRM. La mayor pérdida de heterocigotos a nivel de población con respecto a las subpoblaciones puede deberse al efecto Wahlund, efecto que podría ser la causa de la desviación del equilibrio Hardy-Weinberg que se presenta en el OCPC.
Los resultados obtenidos para Fis, Fit y la desviación del equilibrio Hardy-Weinberg evidencian la existencia de un tipo de subestructura poblacional. Esto se apoya al observar los resultados del AMOVA, donde el valor del Fst entre las dos subpoblaciones es de 0.042, valor relativamente bajo, pero significativamente diferente de 0 (p<0.001), que demuestra un nivel de diferenciación entre las dos poblaciones. La subestructura poblacional se evidencia aún más en el análisis de componentes principales (PCA, Figura 2), donde los dos primeros ejes explican la varianza del 12.8% de la varianza total de la población; valor relativamente bajo, pero que muestra una tendencia de aislamiento entre los individuos de la población del PDML con los individuos del VIRM.
La subestructura poblacional que se evidencia en el estudio responde al efecto del aislamiento geográfico que existe entre las dos zonas geográficas, el cual genera un cierto aislamiento genético debido a la Cordillera Oriental que las atraviesa. Otra explicación es la generalización del nombre «criollo», otorgado a todo animal que no encaja dentro del prototipo de una raza «pura». Estos fenotipos y genotipos han estado sometidos a un proceso de selección, tanto natural como artificial, generando una alta diversidad de animales. La FAO, en el Plan de Acción Mundial sobre los Recursos Zoogenéticos y la Declaración de Interlaken declara: «ante la amenaza que supone para la ganadería el cambio climático, el potencial que ofrecen las razas autóctonas podría ser una solución de adaptación para el sector ganadero» (FAO, 2007).
En conclusión, la población denominada OCPC posiblemente corresponda a dos grupos raciales en las zonas geográficas estudiadas. De otro lado, por población y por subpoblaciones, la diversidad genética del OCPC comparada con otras razas a nivel mundial es óptima. Los SNPs demuestran tener un alto potencial para ser usados en trabajos referentes a la conservación genética de los animales domésticos, aunque su costo es aún muy elevado para los países en desarrollo (según el Banco Mundial), respecto a otro tipo de marcador molecular, como lo son los microsatélites.












 uBio
uBio 

