INTRODUCCIÓN
La ocurrencia de abortos o absorciones embrionarias es una de las principales causas de la baja eficiencia reproductiva en el ganado bovino (Disking y Morris, 2008; Disking et al., 2012). Estos pueden deberse a la depresión endogámica, observada por el incremento de la consanguinidad en los animales, principalmente en el ganado bovino, o a efectos genéticos (Mylrea, 1963; Wijeratne y Stewart, 1971), determinado por la presencia de haplotipos asociados a la reproducción en estado homocigota del embrión (VanRaden et al., 2012).
Diversas investigaciones han identificado la presencia de haplotipos relacionados con la reducción de las tasas reproductivas. Al inicio se reportaron tres haplotipos (VanRaden et al., 2011) y hacia 2017 se sumaron dos más para el ganado Holstein (Cooper et al., 2013), los cuales se asocian a efectos negativos en el desarrollo embrionario y abortos (Norman et al., 2012).
Los efectos negativos de la presencia de estos haplotipos en la reproducción estiman un impacto sobre la tasa de concepción de -0.35% para el tipo HH1, y algo similar (-0.36%) para el haplotipo HH2 y HH3, y cuyas frecuencias de los heterocigotos fueron reportadas de 4.5, 4.6 y 4.7% respectivamente (VanRaden et al., 2011). Al evaluar la tasa de no retorno a 60 días (NR60) se estimó un impacto fue de -1.1, -1.7 y -3.1% para los haplotipos HH1, HH2 y HH3, respectivamente. Por otro lado, Fritz et al. (2013) reportaron frecuencias de 3.6 y 3.9% para los haplotipos HH4 y HH5, respectivamente, asociado a una pérdida en nacimientos en vacas en -1.74% para el HH4 y de -2.17% para el HH5.
Asimismo, los haplotipos han sido relacionados a grupos genéticos de toros que fueron utilizados en poblaciones de vacunos Holstein, tanto para obtener machos reproductores como vacas productoras, lo cual generó un impacto en la incidencia y presencia de los haplotipos reproductivos en la ganadería lechera local. Reproductores tales como Pawnee Farm Arlinda CHIEF y Walkway Chief MARK, en quienes se reporta la presencia del haplotipo HH1 (Larkin et al., 2012, VanRaden et al., 2012), han sido utilizados para producir progenie orientada a seleccionar machos para su empleo en centrales de inseminación artificial, generando una gran influencia genética en la población de vacas resultantes.
El objetivo de la presente investigación fue determinar, en un rebaño lechero en la costa del Perú, la presencia de haplotipos reproductivos asociados con una baja eficiencia reproductiva, así como determinar la correlación entre la tasa de concepción genómica en vacas y el número de servicios por preñez.
MATERIALES Y MÉTODOS
Los datos del estudio fueron obtenidos de la base de datos de una ganadería bajo un sistema intensivo dedicada a la crianza de vacunos lecheros en la costa central del Perú. Los animales de fundación correspondieron a genética norteamericana. De los 8833 registros de producción/ reproducción con que cuenta la empresa, se consideraron 6350 registros que corresponden a los años 20002016 (primer semestre), de 2628 vacas en total, con una media de los últimos cinco años de 509 vacas por año. El establo se encuentra certificado libre de TBC y brucelosis por el Servicio Nacional de Sanidad Agraria (SENASA).
El sistema de manejo de la ganadería se basa en un sistema de alimentación TMR (Ración Totalmente Mezclada), determinándose seis grupos de alimentación: vacas recién paridas, de alta, media y baja producción, vacas en seca y preparto. La detección de celo se realizó mediante el uso de podómetros Westfalia GEA™, que es un sistema de medición de actividad y monitoreado por el software de gestión de rebaños Dairy Plan C21™. Para la confirmación de preñez a los 35 días se utilizó un ecógrafo portátil (Caresono SD9000).
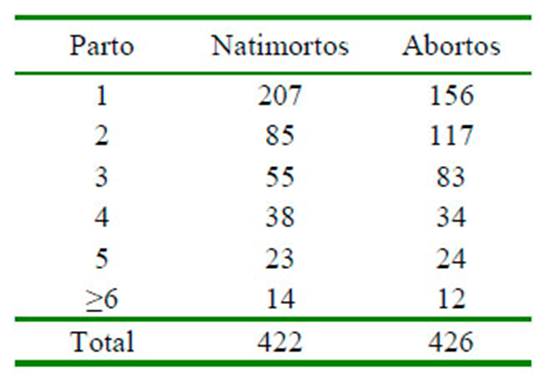
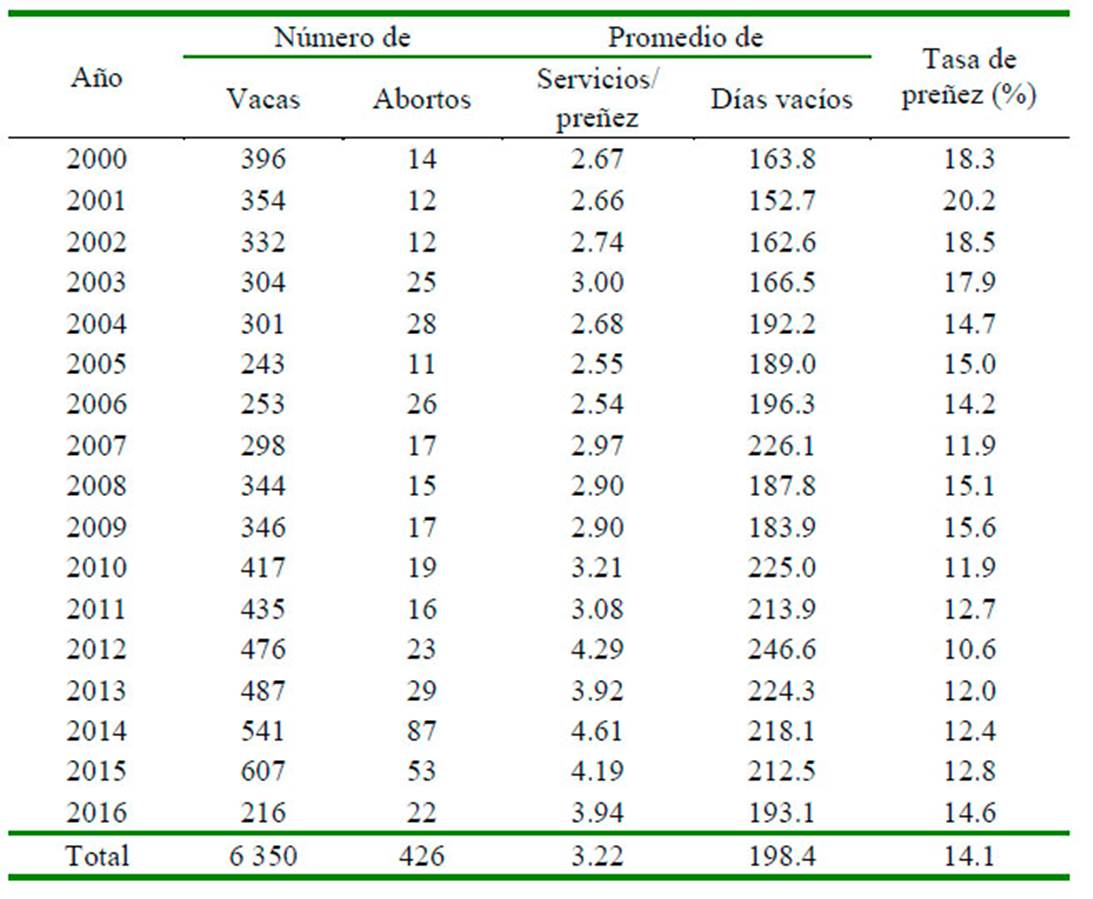
Los abortos registrados por año, el perfil reproductivo del rebaño medido como el promedio de servicios por preñez, el promedio de días vacíos, y la tasa de preñez fue calculado siguiendo la metodología descrita por VanRaden et al. (2004) y se presenta en el Cuadro 1. En el periodo 2000-2016 re registró 422 casos de natimortos y 426 abortos (Cuadro 2).
Se construyó la genealogía con base a la información de los registros genealógicos, tanto de la línea paterna como materna, para ser analizados con los programas informáticos Endog V4.8 (Gutiérrez y Goyache, 2005) y Pedigraph V2.4 (Garbe y Da, 2008), identificando 17 478 animales, utilizando la base de datos de la Asociación Holstein de los Estados Unidos (Holstein Association USA, 2017) y los certificados genealógicos del establo lechero en estudio.
El tamaño de la muestra a genotipar se determinó utilizando la metodología descrita por Krejcie y Morgan (1970), considerando una precisión de 0.05, un tamaño de rebaño de 500 vacas, y la probabilidad de ocurrencia promedio de los haplotipos, obteniendo un tamaño de muestra de 59 animales. Se seleccionaron 60 animales para el genotipado utilizando una rutina para generar números aleatorios en una hoja electrónica usando la función aleatorio(), independiente si las vacas presentaban historial de abortos o un número determinado de partos.
Se tomaron muestras de pelo de la cola de los animales seleccionados, considerando un mínimo de 40 pelos con bulbo piloso intacto para la extracción de ADN. Las muestras se colocaron en las tarjetas colectoras otorgadas por el laboratorio y fueron enviadas al laboratorio de GeneSeek (Neogen Corporation, USA), siguiendo las regulaciones del Departamento de Agricultura de Estados Unidos de Norteamérica Servicio de Inspección Sanitaria de Animales y Plantas (USDA APHIS) para muestras biológicas. El genotipado se realizó con el GeneSeek Genomic Profiler for Dairy-LD (GGP 50k, Igenity Prime). El análisis reportó resultados de 45 características que el USDA considera en sus evaluaciones genéticas y genómicas para ganado lechero Holstein, en la que incluye los haplotipos reproductivos y los valores de la tasa de concepción de la vaca genómico (CCR: Cow Conception Rate).
Cuadro 1 Distribución del número de abortos, número de servicios por preñez, días vacíos y tasa de preñez por año (periodo 2000-2016)
Cuadro 3 Frecuencias de haplotipos reproductivos en bovinos lecheros de una ganadería de la costa central del Perú

El CCR es definido como la capacidad de quedar preñada una vaca en cada servicio, donde una puntuación de +1 indica que la vaca tiene un 1% más de probabilidades de quedar preñada que las vacas con una evaluación de 0. La estimación de dicho valor genómico es determinada por la frecuencia alélica de la población base menos el genotipo y el efecto poligénico, con un 10% de varianza aditiva y se ajusta el modelo: DP = media + S genotipos(efectos) + poli + error (CDCB, 2017).
RESULTADOS Y DISCUSIÓN
De las 60 muestras enviadas a GeneSeek, solo se genotiparon 58, excluyéndose dos muestras por posible contaminación del ADN. Con los resultados del genotipado se determinaron las frecuencias de los portadores para los haplotipos asociados a la reproducción (HH) y el valor genómico de la Tasa de concepción de las vacas (CCR), que es un indicador de la eficiencia reproductiva del punto de vista genómico.
Haplotipos HH
En los 58 genotipados se encontraron tres de los cinco haplotipos reportados en la raza Holstein que se encuentran asociados a la baja eficiencia reproductiva (VanRaden et al., 2011; Cole et al., 2014). Estos fueron los haplotipos HH1, HH3 y HH5, cuyas frecuencias de portadoras fueron similares a las reportados en otras poblaciones de ganado Holstein (VanRaden et al., 2011, 2012; Fritz et al., 2013). Las frecuencias se presentan en el Cuadro 3.
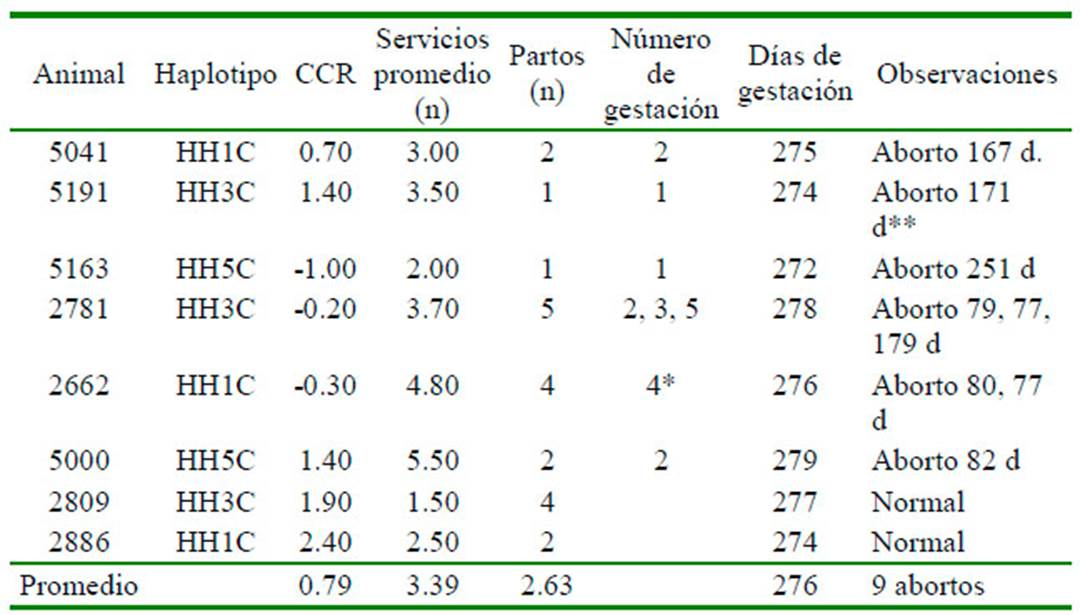
Al analizar el comportamiento reproductivo de las vacas portadoras del haplotipo a través de los partos que han tenido, se encontró que el 75% de las vacas portadoras (6 de 8 vacas) presentaron abortos durante su vida productiva, independientemente del número de partos, como es el caso de la vaca 2781 que en tres de los cinco partos registrados presentó abortos. Se entiende que la presentación de aborto debe darse como causa del haplotipo en estado de homocigosis del embrión, que en el caso de la vaca 5191 pudo deberse ya que el embrión tenía una probabilidad de 25% de ser homocigota al ser el padre portador para HH3, mientras que en los otros casos los reproductores utilizados en la reproducción no fueron portadores para los haplotipos. La presentación de los sucesos de aborto se resume en el Cuadro 4.
El 54% de las vacas no portadoras (27/50) para los haplotipos reproductivos presentaron abortos a lo largo de su vida reproductiva, y el 46% restante no presentó abortos. En el análisis de los datos se observó que los toros utilizados en la inseminación artificial en el grupo de vacas que presentaron abortos fueron heterocigotos para los haplotipos reproductivos (Cuadro 5).
En el grupo de vacas portadoras, la media de gestación al aborto fue de 129.2 días, y para el grupo de vacas no portadoras que presentaron un evento de aborto fue de 134.7 días, valores muy similares, lo cual no permite establecer un efecto en la pérdida de la gestación atribuible a la presencia de los haplotipos reproductivos.
Tasa de Concepción de las vacas
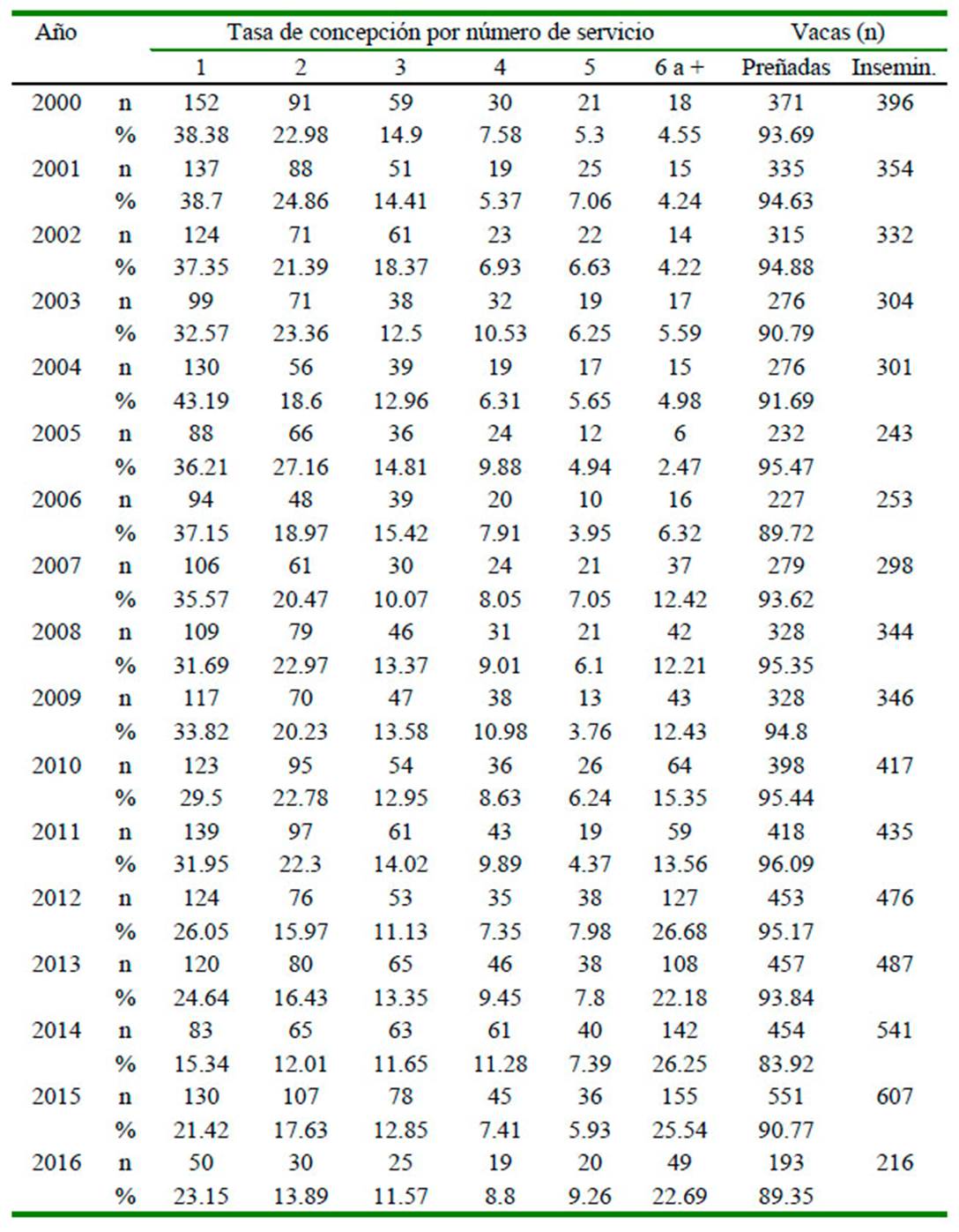
La tasa de concepción en el periodo de estudio fue menor en los últimos años que en los primeros años (Cuadro 6 ), pudiendo deberse a una posible endogamia del rebaño (Parland et al., 2007; González-Recio et al.,2007).
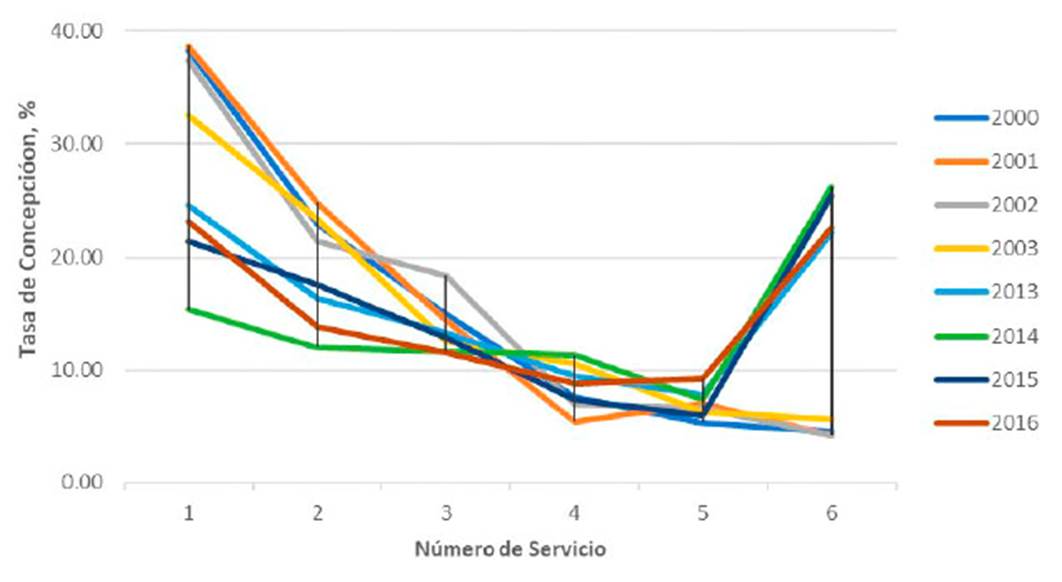
La Figura 1 muestra la reducción de la tasa de concepción por número de servicio entre los cuatro primeros años (2000-2003) y los cuatro últimos años (2013-2016), observándose una reducción significativa de la tasa de concepción (Cuadro 1).
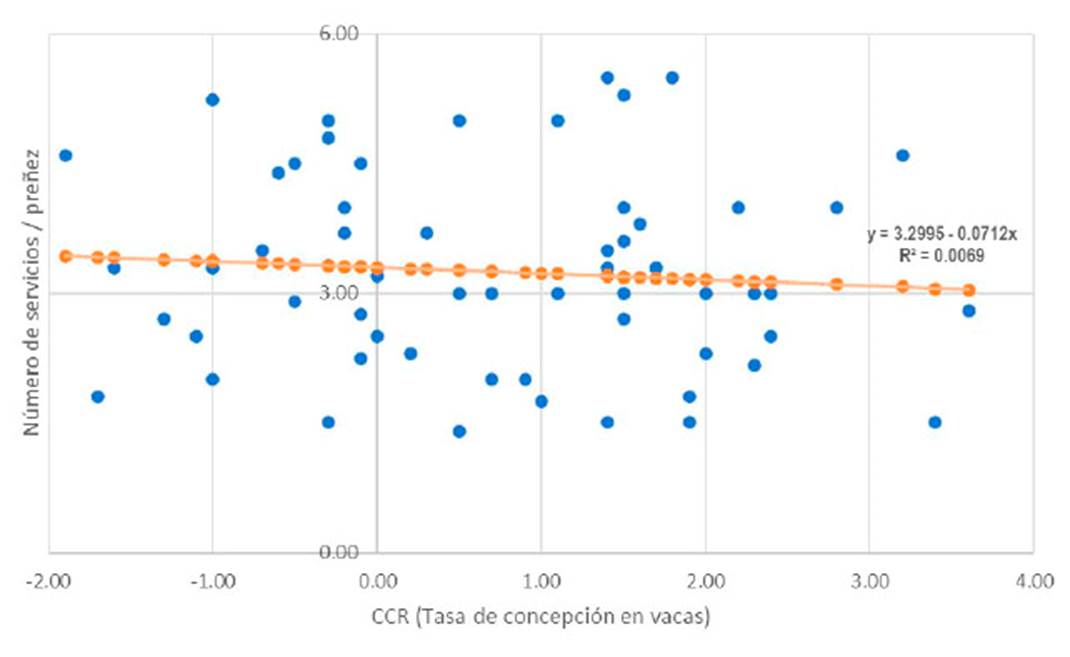
Se encontró una baja correlación (8.30%) entre los valores de la tasa de concepción determinados genómicamente (CCR) con el número de servicios efectivos para preñez (p>0.05), y la tendencia de la variación, calculado mediante el coeficiente de regresión, fue de -0.07115 servicios por valor de +1 de CCR, no siendo estadísticamente significativo, lo que permite indicar que la eficiencia de la reproducción, medido como CCR no es un buen indicador de la performance reproductiva de las vacas, ya que existen otros factores que pueden estar influenciando la tasa de preñez (Mylrea, 1963; Wijeratne y Stewart, 1971) (Ver Figura 2).
Grupos Genéticos
Al evaluar la información de la genealogía de las vacas se pudo establecer cinco grupos genéticos de padres (Cuadro 7), considerando como padre fundador aquel animal donde se reporta por primera vez el haplotipo o es ancestro común con los reproductores utilizados en la reproducción y, que, en muchos casos, se utilizaron reproductores portadores de los haplotipos tipo HH.
Cuadro 5 Comportamiento reproductivo de vacas no portadoras para los haplotipos reproductivos y que presentaron abortos
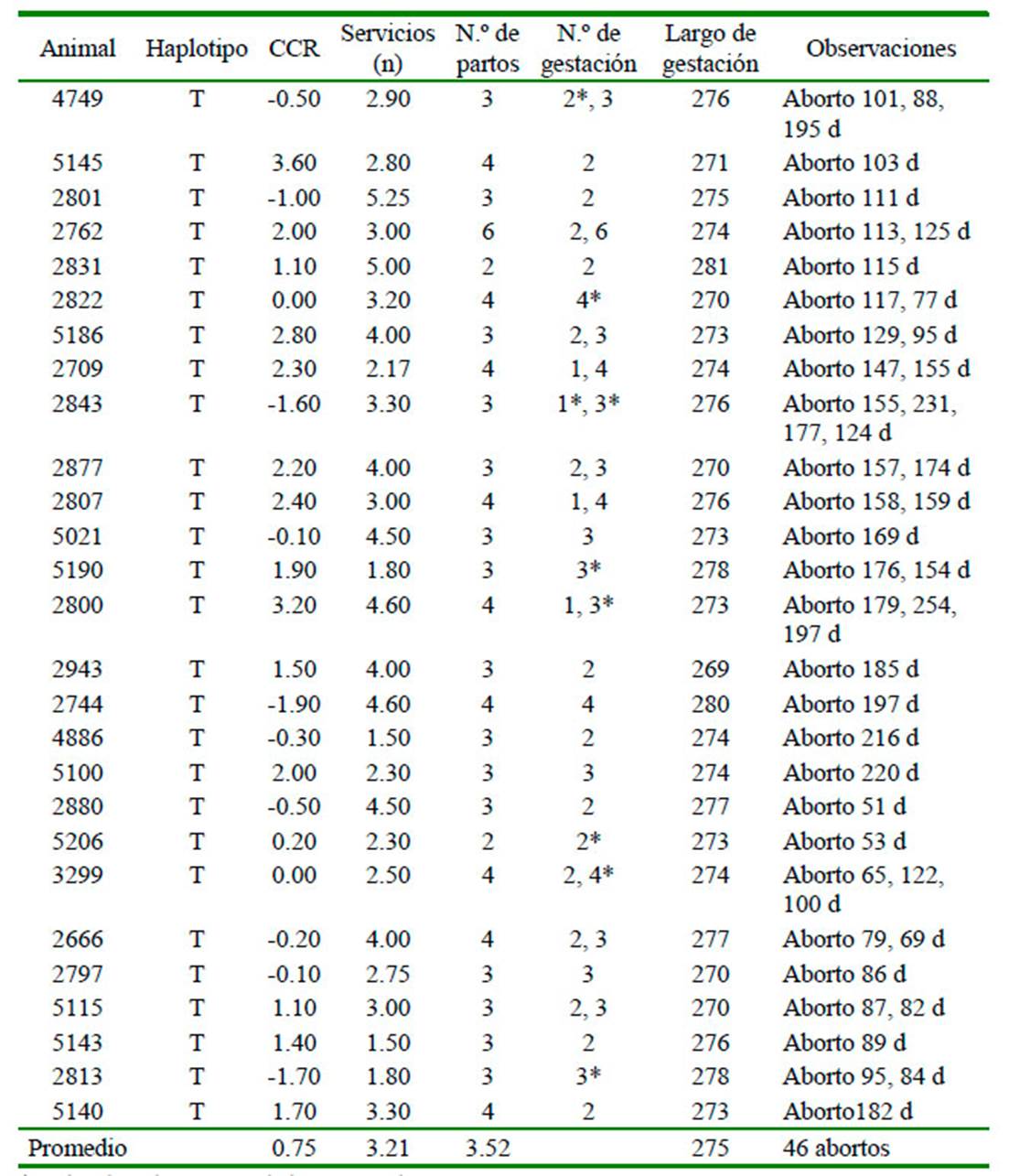
* Hubo dos abortos en dicha gestación
T: No es portador para los haplotipos reproductivos
Cuadro 6 Tasa de concepción del rebaño a través del número de servicios por año, periodo 2000 - 2016.
Cuadro 7 Grupos genéticos identificados en una población de vacas Holstein de una ganadería de la costa central del Perú
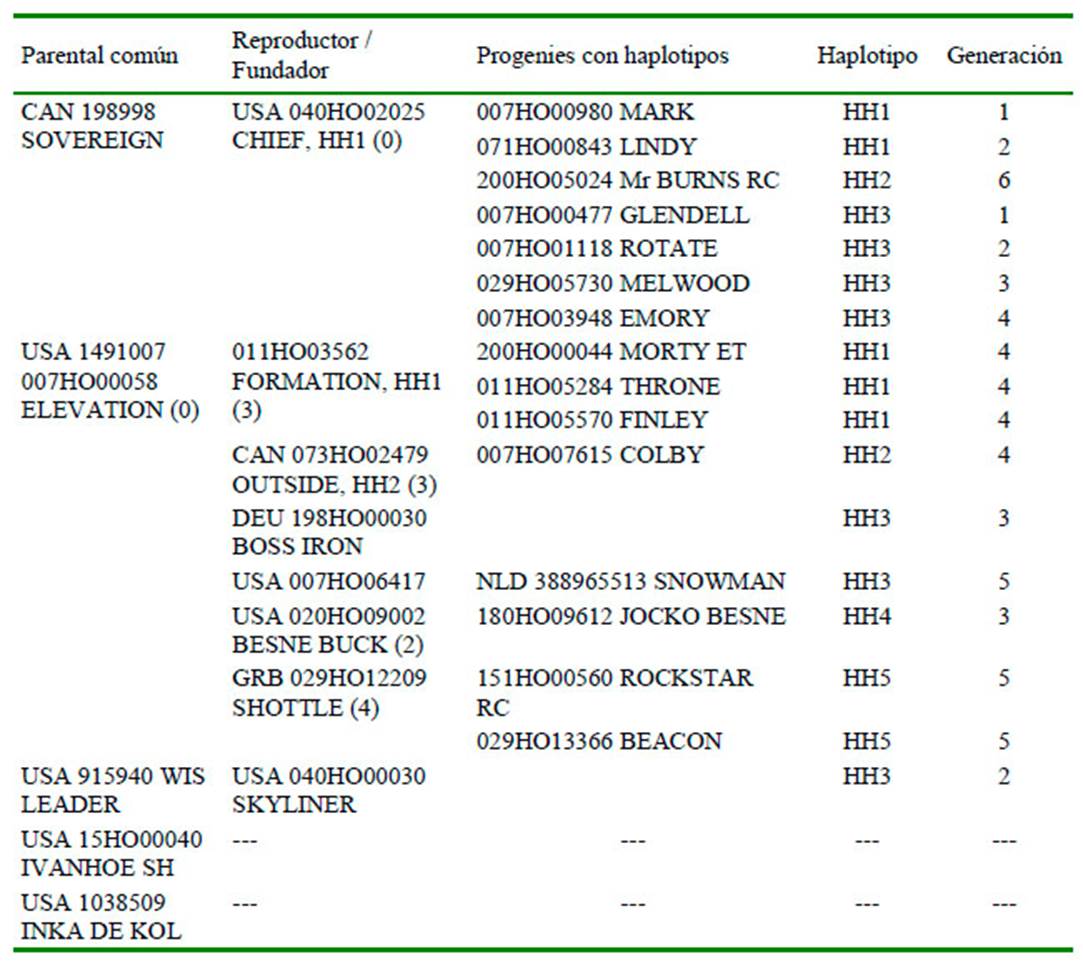
Tres de los cinco grupos genéticos han presentado, a través de su genealogía, los haplotipos asociados a la reproducción. El primer grupo genético corresponde al toro CHIEF, donde se reporta por primera vez el haplotipo HH1 (VanRaden et al., 2011), el segundo corresponde al toro MARK ANTONY que se reporta el haplotipo HH2, el tercero al toro GLENDELL y SKYLER, donde se reporta el haplotipo HH3 y el cuarto grupo genético al toro SHOTTLE donde se reporta el haplotipo HH5.A través de la progenie de cada grupo genético se introdujo al rebaño los haplotipos reportados en el análisis genómico, determinado mediante el programa de análisis Pedigraph (Garbe y Da, 2008).
CONCLUSIONES
La frecuencia de portadores para los haplotipos del tipo HH1 y HH3 fue de 5.2% y para el haplotipo del tipo HH5 de 3.4%.
El comportamiento reproductivo de las vacas portadoras no fue afectado por la condición de la presencia del haplotipo reproductivo (HH1, HH3 y HH5).
Los valores genómicos para tasa de concepción en vacas (CCR) no resultó ser un buen indicador del comportamiento reproductivo medido como número de servicios por preñez, siendo no significativa la correlación y la regresión entre las dos características.
Se determinaron cinco grupos genéticos de padres en el rebaño en estudio y cuatro de ellos presentaron progenie que han sido padres de las vacas y que son portadores para los haplotipos reproductivos












 uBio
uBio