INTRODUCCIÓN
El ganado bovino criado en tierras altas presenta respuestas fisiológicas que determinan su grado de aclimatación a dicho entorno; destacándose la hipertensión arterial pulmonar como el carácter más asociado a dicha aclimatación (Holt y Callan, 2007; Zeng, 2016). Esta característica está determinada por el incremento del tejido muscular de las arteriolas pulmonares, reduciendo su diámetro interno, a diferencia de otros bóvidos como los yaks (Durmowicz et al., 1993). La vasoconstricción de las arteriolas pulmonares ocasiona un incremento de la resistencia al flujo sanguíneo en la que también participa el grado de viscosidad de la sangre como resultado del elevado número de glóbulos rojos, teniendo un efecto en la hipertensión arterial (Vogel et al., 2003). Dicha condición es usual en bovinos criados en ambientes como los Andes, donde los bovinos presentan recuentos altos de glóbulos rojos (RGR), hematocrito (HCT) y hemoglobina (Hb) (Claxton y Ortiz, 1996; Ocampo, 2004). Sin embargo, poblaciones de bovinos criados en las tierras altas de Etiopía presentan valores hematológicos y de presión arterial pulmonar (PAP) bajos, a pesar de estar expuestos al estímulo hipóxico crónico, de allí que se les considera como animales adaptados a ese ambiente (Wuletaw et al., 2011).
Para identificar las características genéticas que permiten la aclimatación y adaptación a la altura de especies animales, entre ellas el bovino, se han estudiado diversos genes, entre ellos el gen denominado proteína 1 para dominio endotelial PAS (EPAS1) (Hara et al., 1999; Simonson et al., 2010, Newman et al., 2011; Wang et al., 2014). Este gen codifica el factor inducible por hipoxia tipo 2 alfa (HIF2α), subunidad alfa sensible al oxígeno, de un heterodímero resultante de la unión con la subunidad común beta o translocador nuclear del receptor de aril hidrocarburos (ARNT) codificado por el gen ARNT. El heterodímero es altamente expresado en células endoteliales vasculares y tiene participación en la sobrerregulación de diversos genes como respuesta al estímulo hipóxico; entre ellos, el factor de crecimiento endotelial vascular (VEGFA) y la eritropoyetina (EPO) (Hara et al., 1999).
En el gen EPAS1 se han encontrado mutaciones o polimorfismos de nucleótido simple (PNSs) en las posiciones de c.G1816G>A y c.1828G>A del exón 12, ambos formando un mismo haplotipo y generando variación en la secuencia de los aminoácidos p.A606T y p.G610S pertenecientes al dominio de degradación dependiente de oxígeno ODDD (Newman et al., 2015; Heaton et al., 2016) y que están presentes en el 75% de bovinos Angus con PAP mayor a 50 mmHg (Newman et al., 2015). Además, se han encontrado otras mutaciones no sinónimas en los exones 7, 9, 12 y 13 que generan variaciones en las posiciones p.E270Q, p.P362L, p.A671G y p.L701F pertenecientes a los dominios PAS-B, e ID de la proteína (Heaton et al., 2016). En equinos se encontró una mutación en la posición p.R144C que es conservada en diferentes especies animales, la cual amplifica la respuesta transcripcional mediada por EPAS1como respuesta a la hipoxia sobre genes de endotelina 1 (EDN1), lactato deshidrogenasa A (LDHA), supresor tumoral von HippelLindau (VHL) y EPO (Liu et al., 2019).
En cuanto al gen EPO, se ha encontrado una mutación con deleción en la posición c.32delG del exón 2 que ocasiona una modificación del marco abierto de lectura (ORF) produciendo ARNm de cadena más corta, el cual genera eritropoyetina de cadena corta en mayor concentración en ciertos individuos que padecen de eritrocitosis (Zmajkovic et al., 2018). Por otro lado, en poblaciones de patos adaptados a condiciones de vida a gran altitud se encontraron PNSs en genes como el óxido nítrico sintetasa NOS, EPAS1, y factor inducible por hipoxia egl-9 homologo 1 (EGLN) destacándose la presencia de mutaciones no sinónimas en los exones 6 y 12 del gen EPAS1 y en el EGLN se encontró un PNS ubicado en el exón 2 que no generó una variación en el aminoácido (Graham y McCracken, 2018). Por su parte, el gen del factor inducible por hipoxia HIF1 expresado en condiciones de hipoxia actúa como regulador maestro de la homeostasis de oxígeno bajo condiciones de hipoxia (Semenza, 2005), en un proceso de cascada el cual media en la expresión de algunos de los genes antes descritos como el EPO y VEGFA (Gilbert-Kawai et al., 2014) y de genes implicados en el transporte y metabolismo de la glucosa como el transportador de solutos familia 2 miembro 1 (SLC2A1) o GLUT-1 en diferentes tejidos (Hayashi et al., 2004; Shao et al., 2014).
En lo que respecta al análisis del ADN de varias especies, se cuenta con información genómica generada por diversos grupos de investigadores que guardan archivos en repositorios como el perteneciente al NCBI (Stothard et al., 2015; Das et al., 2015). La información es libre y además se dispone de herramientas bioinformáticas que permiten evaluar regiones del genoma a fin de identificar PNSs que puedan servir como marcadores asociados a determinadas características de interés. En ese sentido, el presente estudio tuvo como objetivo identificar marcadores genéticos de tipo PNS en regiones exónicas de 10 genes inducibles por hipoxia usando los genomas de un grupo de 41 bovinos de varias razas bovinas cárnicas y lecheras. Asimismo, verificar la existencia de algunos de estos PNSs en regiones exónicas de cuatro genes previamente evaluados y su posible asociación basada en la variación de sus frecuencias alélicas respecto a grupos de bovinos Brown Swiss con mal de altura y de bovinos criollos y Brown Swiss criados en los Andes que expresaron valores extremos de Hb, toda vez que estas poblaciones son consideradas como rústicas y adaptadas a la altura.
MATERIALES Y MÉTODOS
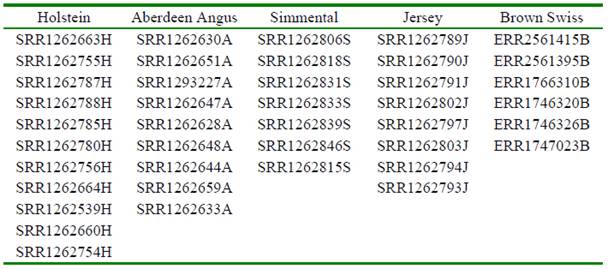
En una primera etapa se identificaron los PNSs en regiones exónicas de los genes EPAS1, HIF1a, EDN1, ARNT, HIF3a, VEGFA, EPO, NOS2, EGLN y SLC2A1, a fin de ubicar mutaciones no sinónimas en las secuencias de ADN. Para ello se empleó la información genómica de 41 bovinos Brown Swiss (n=6), Holstein (11), Jersey (8), Aberdeen Angus (9) y Simmental (7), existente en la base de datos Sequence Read Archive (SRA) del National Center for Biotechnology Information (NCBI, EEUU) (Cuadro 1). La información de los genomas fue descargada en el servidor de la Universidad Nacional Agraria La Molina (UNALM) para su análisis:
La identificación de los PNSs se efectuó mediante el alineamiento de las secuencias obtenidas y el genoma de referencia ARS-UCD1.2 disponible en el NCBI (GCF_002263795.1), utilizando las herramientas bioinformáticas Alineador Burrows-Wheeler (BWA), Picard tools, SamTools y BcfTools;
En la indexación con el genoma de referencia se emplearon las herramientas BWA y SamTools (Li et al., 2009; Li y Durbin, 2010);
En la creación de la secuencia «diccionario», el ordenamiento, la marcación de duplicados, la indexación y la unión de archivos se empleó la herramienta Picard tools (Picard Toolkit, 2019);
El alineamiento de lecturas se hizo con la herramienta BWA;
La identificación y llamado de variantes se hizo con la herramienta BcfTools (BCF Tools, 2019).
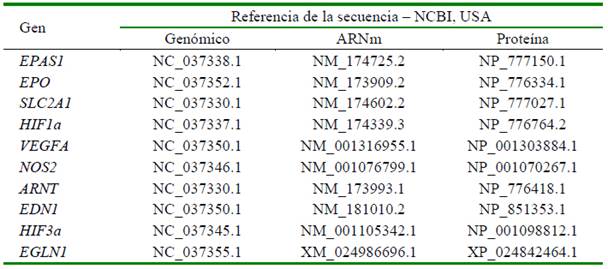
Se obtuvo archivo de extensión .vcf con peso de 19.8 GB, del cual se extrajeron los PNSs ubicados en las coordenadas correspondientes a los genes antes referidos, generándose archivos FASTA con las variantes encontradas. Estos archivos fueron alineados con sus correspondientes archivos de ARNm y de proteínas codificadas existentes en la base de datos de NCBI (Códigos de referencias en el Cuadro 2), usando las herramientas Mega Blast (https://blast.-ncbi.nlm.nih.gov/Blast.cgi) y Splign (https://www.ncbi.nlm.nih.gov/sutils/splign/splign.cgi). De esta manera se identificaron los PNSs que generaban variación en la codificación de los aminoácidos de la proteína resultante. Para este grupo de PNSs se calcularon las frecuencias del alelo menor (MAF en inglés), el equilibrio de Hardy-Weinberg, y el distanciamiento genético usando la prueba G (Sokal y Rohlf, 1995), existentes entre los dos grupos conformados por bovinos productores de carne y leche empleando el programa GENEPOP v. 4.7. (https://genepop.-curtin.edu.au/). Por último, se efectuó un estudio de desequilibrio de ligamiento (LD, por la sigla en inglés) entre PNSs y la identificación de posibles haplotipos usando el algoritmo denominado columna sólida de LD del programa Haploview v.4.2 (Barret et al., 2005)
En la segunda parte del estudio se eligieron cuatro regiones exónicas de los genes EPAS1, EPO, VEGFA y NOS2 a partir de los resultados del análisis bioinformático previo. Para ello, se emplearon 60 muestras de ADN provenientes de bovinos criollos y Brown Swiss criados en zonas altoandinas de las regiones Cusco, Pasco y Puno, Perú, en altitudes comprendidas entre 3213 y 4309 msnm. Estas muestras fueron seleccionadas dentro de un grupo de 323 bovinos de los que se analizaron sus características hematológicas en un estudio comparativo entre grupos raciales criados en condiciones altoandinas. Las muestras de ADN de los 60 individuos fueron distribuidas en cuatro grupos diferenciados por su concentración de Hb (alta: 15.25±0.81 g/dl; baja: 9.59±1.40 g/dl), dividiéndose en Brown Swiss con Hb alta (BSHbA) (n=13), Brown Swiss con Hb baja (BSHbB) (n=11), criollos con Hb alta (CRHbA) (n=13) y criollos con Hb baja (CRHbB) (n=12); además de un quinto grupo formado por 11 bovinos Brown Swiss con mal de altura (BSMA) que tuvieron una concentración de Hb de 15.77±2.82 g/dl.
Exones seleccionados de los genes descritos anteriormente fueron amplificados mediante la reacción en cadena de polimerasa (PCR) usando los cebadores que se detallan en el Cuadro 3. Las muestras fueron preparadas de manera que se obtuvieran volúmenes de 50 µl con una concentración mínima de ADN de 60 ng/µl. Los amplicones fueron corridos en gel de agarosa al 1.5% para observar la calidad de las bandas y tamaño del amplicón generado. Luego, las muestras fueron remitidas para su secuenciación mediante metodología Sanger. Los resultados obtenidos (archivos .bsf1) fueron analizados con el programa BioEdit v. 7.2.5 (Hall, 1999) y la herramienta Clustal Omega, mediante el alineamiento de las secuencias de los exones amplificados para cada gen dentro de cada grupo de animales. Los resultados de este análisis fueron empleados para determinar las frecuencias alélicas de los PNSs y diferencias poblacionales entre grupos raciales y entre grupos según la concentración de Hb. También se calcularon los correspondientes LDs y se identificaron los haplotipos tal y como fue descrito anteriormente.
RESULTADOS
Análisis Bioinformático de 41 Genomas Bovinos
Se encontraron 79 PNSs en las regiones exónicas de los 10 genes evaluados. De estos, 17 PNSs no son sinónimos, ocasionando una variación en la secuencia de aminoácidos (Cuadro 4).
Cuadro 4 Polimorfismos de nucleótido simple (PNSs) identificados en 41 genomas bovinos que generan mutaciones no sinónimas en 10 genes inducibles por hipoxia
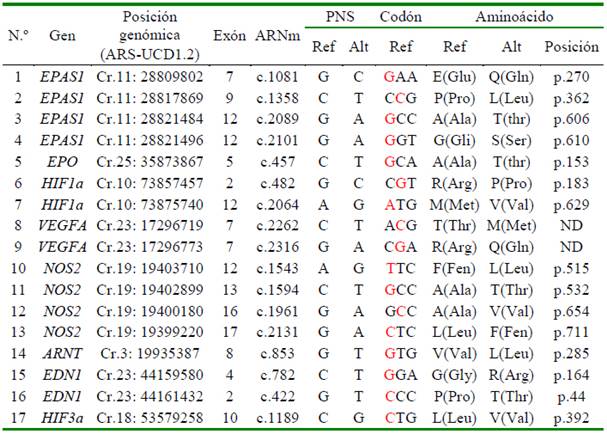
En color rojo ubicación de PNS dentro de codón
ARNm: ARN mensajero; Ref: Referencia; Alt: Alterno; ND: no definido
Al comparar las frecuencias alélicas entre los grupos de bovinos cárnicos y lecheros se observaron diferencias en tres PNSs del gen EPAS1 ubicados en c.1081G>C del exón 7 (p<0.05) y c.2089G>A (p<0.05), c.2101G>A (p<0.01), ambos en el exón 12. El tercer PNS ubicado en la posición c.1358C>T del exón 9 no tuvo una frecuencia diferente entre bovinos cárnicos y lecheros (p>0.05). Asimismo, el resto de PNSs no tuvo una frecuencia alélica diferente entre los grupos cárnicos y lecheros (Cuadro 5).
Cuadro 5 Frecuencia del alelo menor (MAF) y equilibrio de Hardy-Weinberg (HW) de PNSs en genomas de bovinos
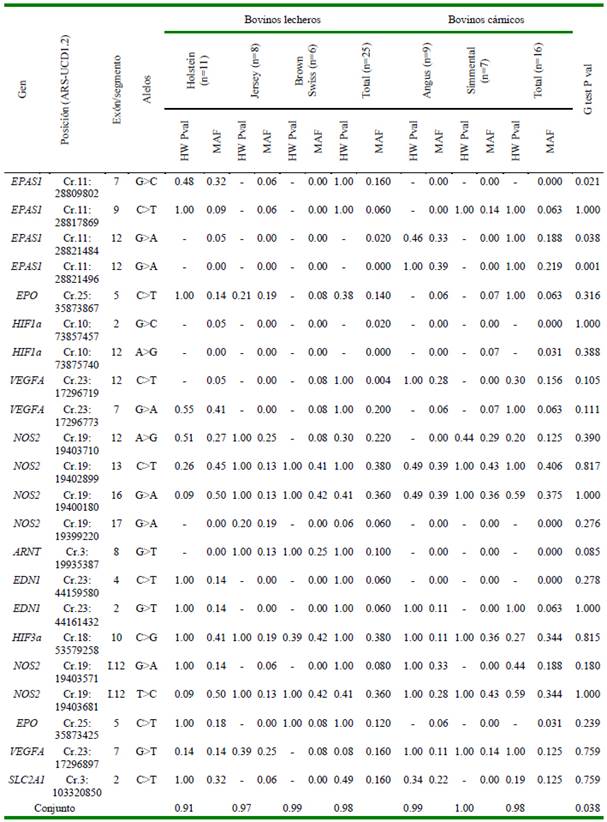
I.12: Intrón 12
Luego de efectuar el análisis general de desequilibrio de ligamiento (LD) con las 41 muestras, se observó que los dos PNSs del gen EPAS1 ubicados en el exón 12 presentaron una puntuación de logaritmo de probabilidades (LOD en inglés) de 5.33 y r2 de 0.70. Cabe señalar que cuando se compararon los grupos de bovinos cárnicos y lecheros de forma independiente, no se observó dicho ligamiento en estos últimos, mientras que en el grupo de bovinos cárnicos el ligamiento tuvo una puntuación LOD de 3.95 y r2 de 0.82.
Por otro lado, con el análisis general de las 41 muestras, se observó que dos PNSs del gen NOS2 ubicados en las posiciones c.1961G>A (exon16) y c.1594C>T (exón 13) tuvieron una puntuación LOD de 7.41 y un r2 de 0.61, pero cuando se analizaron únicamente a bovinos cárnicos se observó un ligamiento entre los PNSs c.1594C>T (exón 13) y Cr.19: 19403681T>C (intrón 12), los cuales tuvieron una puntuación LOD de 2.14 y r2 de 0.52; mientras que en bovinos lecheros, se observó el ligamiento entre los PNSs c1594C>T (exón 13) y c.1961G>A (exón 16) con una puntuación LOD de 5.9 y r2 de 0.75, y estos a su vez estuvieron ligados con un tercer PNS ubicado en la posición Cr.19: 19403681T>C (intrón 12), con puntuaciones LOD de 8.57 y 6.68, y r2 de 0.92 y 0.83, respectivamente.
Considerando las 41 muestras, se observaron otros PNSs que al parecer están ligados o son coheredados. Estos fueron el c.2316G>A ubicado en el exón 7 del gen VEGFA con el c.782C>T (exón 4) del gen EDN1 y entre los PNSs c.1081G>C del gen EPAS1 con c.2316G>A del gen VEGFA. (LOD: 2.06). Sin embargo, en el caso específico de bovinos lecheros se observó una puntuación LOD de 2.04 entre c.2316G>A del gen VEGFA y el c.233C>T del gen SLC2A1.
En lo que se refieren a los haplotipos del gen EPAS1, en el grupo de bovinos cárnicos se observó un bloque de cinco haplotipos formados por el PNS del exón 9 y los dos reportados en el exón 12. Estos fueron CGG (71.9%), CAA (18.8%), TGG (6.3%) y CGA (3.1%). En el caso de los bovinos lecheros se obtuvo un grupo de cinco haplotipos formados por los PNSs de los exones 7, 9, y uno del exón 12 (c.2089G>A); de ellos, los haplotipos GCG (79.2%), CCG (12.8%), GTG, (4.8%) tuvieron una mayor frecuencia.
En el gen NOS2 en bovinos lecheros se observó un bloque conformado por siete haplotipos con los PNSs c.2131G>A, c.1961G>A, c.1594C>T, Cr.19: 194035- 71G>A y Cr.19: 19403681T>C. Los haplotipos más frecuentes fueron GGCGT (53.9%), GATGC (28.0%), AGCGT (6.0%) y GATAC (5.9%). Para el caso de los bovinos cárnicos se observó un bloque de siete haplotipos formados por los PNSs c.1594C>T, Cr.19: 19403571G>A, Cr.19: 19403681T>C y c.1543A>G, siendo los más frecuentes CGTA (44.3%), TGCA (21.2%), CGTG (11.8%).
PNSs en ADN de Bovinos Criollos y Brown Swiss
Al secuenciar las 60 muestras de bovinos locales, se identificaron 10 PNSs en los amplicones obtenidos de los cuatro genes considerados. Estos guardan correspondencia con las posiciones identificadas en los 41 genomas presentados en los Cuadros 4 y 5.
Al comparar las frecuencias alélicas de estos PNSs entre bovinos criollos y Brown Swiss, solo fueron diferentes para dos PNSs ubicados en las posiciones Cr.19: 19403681T>C (intrón 12) y c.1536C>T (exón 12) del gen NOS2. Este último PNS no genera una variación en la secuencia de aminoácidos p.E512E del producto. También se pudo observar que la frecuencia del PNS c.1081G>C del gen EPAS1 fue de 0.10 en los bovinos criollos y en los bovinos CRHbB llegó a 0.17 (Cuadro 7). Además, cuando se agruparon las muestras en los dos grupos generales diferenciados por la concentración de Hb no se pudo apreciar diferencias en las frecuencias alélicas que pudieran estar asociadas con dichos grupos (p>0.05) (Cuadro 6). Tampoco se apreciaron diferencias en las frecuencias de alelos entre los bovinos con mal de altura y los otros cuatro grupos de bovinos criollos y Brown Swiss (Cuadro 7).
Cuadro 6 Frecuencia del alelo menor (MAF)y equilibrio de Hardy-Weinberg (HW) de polimorfismos de nucleótido simple (PNSs) no sinónimos observados en bovinos criados en los Andes peruanos
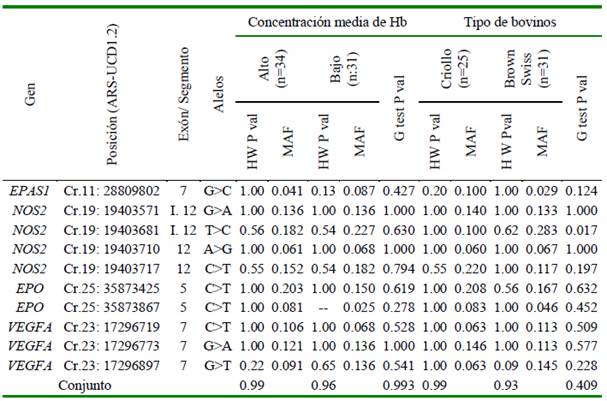
I.12: Intrón 12
Hemoglobina alta (15.25±0.81 g/dl); baja: (9.59±1.40 g/dl)
Cuadro 7 Frecuencia del alelo menor (MAF) y equilibrio de Hardy-Weinberg (HW) de polimorfismos de nucleótido simple (PNSs) observados en bovinos por raza y nivel de hemoglobina dentro de raza
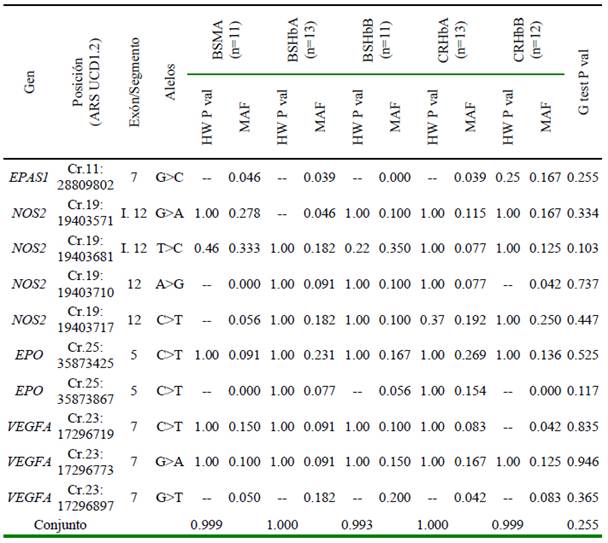
I.12: Intrón 12
Hemoglobina (Hb) alta (15.25±0.81 g/dl); baja: (9.59±1.40 g/dl)
BSMA: Brown Swiss con mal de altura, BSHbA: Brown Swiss con Hb alta, BSHbB: Brown Swiss con Hb baja, CRHbA: Criollo con Hb alta, CRHbB: Criollo con Hb baja
En lo referido al análisis de desequilibrio de ligamiento de los PNSs, en el grupo Brown Swiss se observó la existencia de ligamiento entre los dos PNSs del gen NOS2 ubicados en las posiciones c.1543A>G y c.1536C>T, ambas dentro del exón 12, con una puntuación LOD de 3.1 y un r2 0.54. En el grupo BSHbB, el ligamiento tuvo una puntuación LOD de 2.2 y un r2 de 1.0. Además, en este grupo racial se identificó un bloque formado por cuatro haplotipos constituidos por los PNSs Cr.19: 19403681T>C, c.1543A>G y c.1536C>T del gen NOS2: TAC, CAC, TGT y TAT existentes en el 61, 28, 6 y 5% del grupo; mientras que en los bovinos criollos se observó un bloque conformado por los dos últimos PNSs del gen NOS2 antes referidos, encontrándose tres haplotipos: AC, AT, GT en el 78,16 y 6% de los animales de este grupo.
Otro análisis para identificar haplotipos considerando esta vez a los 10 PNSs de los cuatro genes estudiados, permitió identificar un haplotipo común a los cinco grupos de estudio. Este fue CGGGTACGCC constituido por los PNSs VEGFA c.2262C>T, VEGFA c.2316G>A, VEGFA Cr.23: 17296897G>T, NOS2 Cr.19: 19403571G>A, NOS2 Cr.19: 19403681T>C, NOS2 c.1543A>G, NOS2 c.1536C>T, EPAS1 c.1081G>C, EPO Cr.25: 35873425C>T y EPO c.457C>T. Sin embargo, la frecuencia de este varió entre grupos, observándose en bovinos BSMA y BSHbA una frecuencia de 0.51 y 0.55, mientras que en bovinos BSHbB, CRHbA y CRHbB fue de 0.35, 0.35 y 0.33, respectivamente. También se identificaron dos haplotipos propios de bovinos criollos, el primero CGGG-TATGCC con frecuencias de 0.15 en CRHbA y 0.13 en CRHbB y el segundo CGGG-TACGTC con frecuencias de 0.13 en CRHbA y 0.08 en CRHbB. No se identificó un haplotipo que fuera único para el grupo de bovinos BSMA que tuviera una frecuencia alta como para ser considerado que este asociado con dicho grupo de animales.
DISCUSIÓN
La elección de exones para amplificar en los grupos locales de bovinos criollos y Brown Swiss se basó en la identificación de PNSs no sinónimos existentes en los 41 genomas bovinos de la base datos SRA del NCBI. Para el caso del gen EPAS1 los resultados fueron concordantes en lo que se refiere a PNSs del exón 12 con los resultados de Newman et al. (2015) y Heaton et al. (2016); además, se evidenció la ausencia de PNSs en las posiciones c.2089G>A y c.2101G>A en bovinos lecheros, por lo que no se incluyó dicho exón en el análisis de las muestras de criollos y Brown Swiss locales; sin embargo, esto no implica que no existan dichas variantes en bovinos criollos, quedando pendiente dicha evaluación.
La variante del gen EPAS1 ubicada en la posición c.1081G>C del exón 7 que genera un cambio en p.E270Q, está presente en los genomas de Holstein y Jersey y aunque no fue identificada en Brown Swiss (Cuadro 5), se decidió analizarla. Ello permitió identificar el PNS en bovinos Brown Swiss y los criollos, aunque en baja frecuencia, no pudiéndose encontrar diferencias entre estos grupos raciales ni en cuánto a la concentración de Hb, posiblemente debido al escaso número de muestras por grupo.
Un PNS que tuvo frecuencia alélica diferente entre los criollos y los Brown Swiss fue el Cr.19: 19403681T>C del gen NOS2 (p<0.05). Este PNS se ubica en una región intrónica, pero de acuerdo con los resultados de este estudio, está ligado con PNSs identificados en regiones exónicas del mismo gen. Como se sabe, el gen NOS2 está más vinculado con la repuesta inflamatoria (Accinelli y Lopez, 2018) y su trascripción está regulada por el incremento de las concentraciones del factor de necrosis tumoral alfa (TNFα) e interleucina beta (IL-β) en células endoteliales vasculares (Zulueta et al., 2002). Además, el NO tiene efecto sobre el desarrollo de tejido muscular y endotelial vascular, participando en la regulación del tono vascular, entre otras funciones (Shao et al., 2015).
Debido a la importancia de estas funciones y considerando los procesos inflamatorios relacionados con el síndrome de mal de altura, se esperaba encontrar alguna asociación entre los PNSs del gen NOS2 con el grupo de bovinos BSMA que los diferenciara de los cuatro grupos de bovinos criollos y Brown Swiss diferenciados por su concentración de Hb. Sin embargo, al no detectarse este tipo de asociación, se tendría que analizar las posibles diferencias en la expresión génica de genes inducibles por hipoxia entre los grupos considerados. Esta es una alternativa de trabajo con relación a estudios de adaptación a condiciones de altura (Verma et al., 2018), que podría permitir entender que los potenciadores o inhibidores del gen NOS2 que controlan su expresión, así como la de muchos otros genes que participan en este proceso.
Por otro lado, la evaluación de haplotipos considerando a los 10 PNSs permitió identificar un haplotipo propio de los bovinos BSMA que pudiera tener una alta frecuencia en dicho grupo y aunque se pudo identificar dos haplotipos propios para bovinos criollos, la suma de ambas frecuencias observadas en CRHbA y CRHbB, estuvo en un rango entre 21 y 28%, por lo que no son referenciales para esta población.
Con base a estos resultados, sería conveniente desarrollar un estudio de GWAS con mayor número de muestras, como los realizados por Wei et al. (2016) en ovinos o Liu et al. (2019) en equinos, e incluir información fenotípica referida a características fisiológicas que se presentan en el síndrome de mal de altura o que sean propias de animales adaptados a entornos de altitud, tal como se hacen en estudios para características de interés productivo o de adaptación ambiental (Martinez et al., 2014; De Leon et al., 2019), a fin de identificar PNSs de efecto mayor o que puedan estar implicados en los procesos metabólicos intermedios del síndrome de mal de altura y en la adaptación de poblaciones bovinas locales a la altura.
CONCLUSIONES
Se identificaron 17 polimorfismos de nucleótido simple (PNSs) en regiones exónicas que generan mutaciones no sinónimas en los 10 genes inducibles por hipoxia evaluados.
No se identificaron PNSs en las cuatro regiones exónicas de los genes estudiados que estuvieran asociados con la variación extrema de hemoglobina ni con el mal de altura en bovinos criados en la zona altoandina del Perú.
Considerando los 10 PNSs estudiados en bovinos criados en la altura, se pudo identificar dos haplotipos propios de bovinos criollos, aunque presentes en baja frecuencia.
No se pudo identificar un haplotipo propio de bovinos Brown Swiss con mal de altura.












 uBio
uBio