INTRODUCCION
El cuy (Cavia porcellus), como cualquier especie productiva, es susceptible a sufrir enfermedades que traen consigo pérdidas económicas, influenciadas directamente por las deficientes condiciones de crianza y alimentación y niveles de bioseguridad en los lugares de crianza (Matsuura et al., 2010; Morales S, 2012). Dentro de las enfermedades, una de las más graves es la salmonelosis, causada en el 95% de casos por el serotipo Typhimurium (Chauca, 1997). Esta origina morbilidad entre 0.87 y 52.7% y mortalidad hasta del 95%, con manifestaciones clínicas variadas (Morales et al., 2007).
Se ha demostrado la existencia de S. Typhimurium en aislados de cuyes en aparente buen estado de salud utilizados como reproductores (Chero et al., 2017), lo que indicaría que estos animales actuarían como portadores y diseminadores de la enfermedad. Con respecto al proceso patogénico en la salmonelosis, existe un grupo de genes que actúan de manera coordinada, codificando factores solubles que modifican la fisiología celular del hospedero o protegen a la bacteria de la acción antimicrobiana para asistir al patógeno a expresar su virulencia en la célula hospedero y finalmente manifestar la signología (Murugkar et al., 2003; van Asten y Van Dijk, 2005; Martínez, 2007).
Estos factores de virulencia, evaluados previamente en el modelo murino, se encuentran en diversos serovares de Salmonella, incluido Salmonella Typhimurium, los cuales son codificados por genes como spvB, invA, tolC, sopB, spiA, entre otros; que cumplen funciones biológicas asociadas al reconocimiento del hospedero, adhesión e invasión, supervivencia y muerte de macrófagos, etc., con las que se posibilita el desarrollo y mantenimiento de la enfermedad (Skyberg et al., 2006). Por ello, es importante conocer los factores de virulencia que existen en S. Typhimurium aislados de cuyes con salmonelosis y de cuyes aparentemente sanos. Así, el presente estudio tuvo como objetivo evaluar los de genes de virulencia spvB, spiA, cdtB, sipB, tolC, sitC, lpfC, sifA, sopB y pefA presentes en S. Typhimurium de cuyes enfermos y cuyes aparentemente sanos.
MATERIALES Y MÉTODOS
Material Experimental
Se analizaron 100 aislados de Salmonella Typhimurium de cuyes, conformados por 10 aislados de hisopados vaginales/rectales de cuyes hembra aparentemente sanas y 90 aislados recuperados de muestras de órganos de cuyes diagnosticados con salmonelosis, provenientes de granjas de producción de Huaral, Manchay y Huancayo (Perú). Dicho material fue colectado en estudios previos, enmarcados en el proyecto «Desarrollo de una vacuna para el control y prevención de la salmonelosis en la producción de cuyes» Contrato N.° 362- PNICP-PIAP-2014 (Innóvate Perú), e identificados a través de PCR convencional, detectando la presencia de los genes invA (presente en S. enterica subsp enterica) y flicC (presente en el serovar Typhimurium).
El procesamiento de las muestras se realizó en la Unidad de Biología y Genética Molecular de la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos, Lima, Perú.
Reactivación de Aislados
Los 100 aislados de S. Typhimurium fueron reactivados siguiendo el protocolo establecido por Sánchez y Corrales (2005): Cada aislado fue sembrado por agotamiento en una placa de agar Xilosa Lisina Desoxicolato (XLD, Britania) e incubados a 42 °C por 24 h en condiciones aeróbicas. Luego se verificó el crecimiento de colonias características color rojo, lactosa negativas, diámetro de 1.5 mm y ligeramente convexas, las cuales pueden presentar un precipitado color negro al centro debido a la producción de sulfuro de hidrógeno (Flores, 2003). Una vez confirmado el crecimiento de salmonellas puras en placa, se tomó una colonia por cada aislado y se inoculó en 2 ml de caldo Luria Bertani (LB, Merck), manteniendo en incubación con agitación a 37 °C por 3 h.
Extracción de ADN
Luego del proceso de incubación y verificando la turbidez de la solución debido al crecimiento bacteriano, se extrajo el ADN de cada muestra mediante el método de ebullición, de acuerdo con el protocolo de Karimnasab et al. (2013). Brevemente, los cultivos fueron centrifugados a 15 000 g por 5 min, se descartó el sobrenadante y se homogenizó con 50 µl de agua ultrapura estéril libre de DNasas y RNasas (Invitrogen) a fin de resuspender el pellet formado. Posteriormente, las muestras se sometieron a ebullición (100 °C) por 5 min, seguidos de una centrifugación a 15 000 g durante 5 min. Finalmente, se recuperaron los sobrenadantes y se colocaron en viales de 600 µl. Finalmente se cuantificaron las concentraciones de ADN obtenidas con el espectrofotómetro (260 nm), obteniéndose valores de concentración entre 35-75 ng/µl de ADN.
PCR Múltiple
Para evaluar los 10 genes de virulencia de S. Typhimurium presentes en los aislados se utilizó la técnica de PCR Múltiple, haciendo uso de los cebadores descritos por Skyberg et al. (2006) (Cuadro 1). Se realizaron dos PCR múltiples, la primera reacción para amplificar los genes spvB, spiA, sipB, tolC y cdtB, y la segunda, para amplificar los genes sitC, lpfC, sifA, sopB y pefA. La mezcla para las PCR Múltiples consistió en ambos casos de Buffer 10X PCR (100 mM KCl, 100 mM Tris-HCl, 20 mM de MgCl2), 0.25 mM de desoxinucleótido trifosfato, mix de cebadores (cada cebador a 0.5 mM), 25 mM de MgCl2, 1 U de Taq Polimerasa; y 2.5 µl de ADN, en un volumen total de 20 µl para cada muestra.
En todos los ensayos se procesó como control positivo la cepa Salmonella Typhimurium ATCC 14028 bajo las mismas condiciones que los aislados.
La reacción de amplificación de los genes se llevó a cabo bajo las siguientes condiciones: desnaturalización inicial 95 °C por 5 min, 30 ciclos de desnaturalización a 94 °C por 30 s, hibridación a 66.5 °C por 30 s, extensión a 72 °C por 1 min, y una extensión final a 72 °C por 10 min, en un termociclador MyCycler (Bio-Rad). Los productos de PCR fueron analizados mediante electroforesis en un gel de agarosa al 2%, con TBE 0.5X como buffer de corrida a 100 V y 100 mA por 1 h. Para visualizar las bandas de ADN se utilizó bromuro de etidio como revelador, y el tamaño de los amplificados fueron determinados con un marcador de peso molecular de 100 pb (DNA Ladder, Gene Ruller, Fermentas).
Análisis de Datos
Los resultados se expresaron en porcentajes organizados en tablas de doble entrada. Así mismo, se usó la prueba exacta de Fisher para determinar la asociación entre la presencia o ausencia de cada gen y el aparentemente estado sanitario de los cuyes, con un nivel de confianza de 95%.
RESULTADOS Y DISCUSIÓN
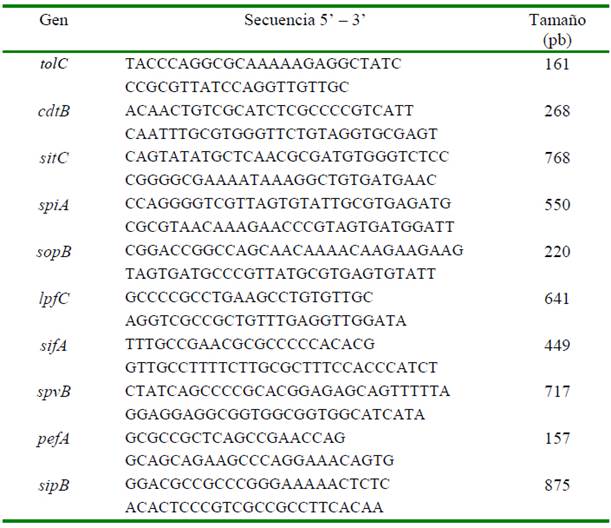
Se evaluó la presencia de 10 genes que codifican factores de virulencia en Salmonella Typhimurium aislados de cuyes aparentemente sanos (n=10 aislados) y enfermos (n=90 aislados). Estos genes pudieron ser detectados mediante el uso de la técnica de PCR Múltiple: la primera reacción permitió evaluar los genes tolC (161 pb), cdtB (268 pb), sipA (550 pb), spvB (717 pb) y sipB (875 pb) (Figura 1a) y la segunda reacción amplificó fragmentos de los siguientes genes: pefA (157 pb), sopB (220 pb), sifA (449 pb), lpfC (641 pb) y sitC (768 pb) (Figura 1b).
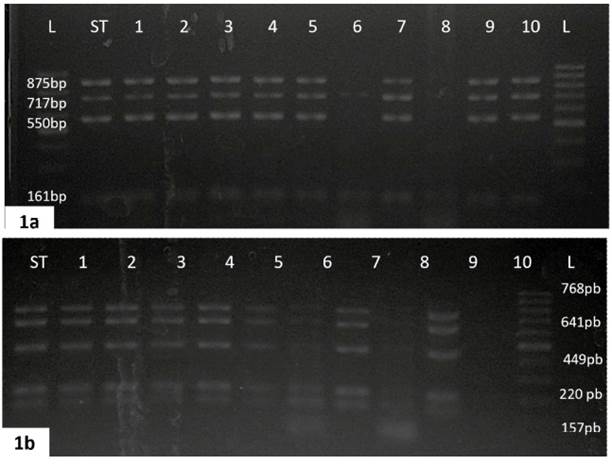
Figura 1 Detección de factores de virulencia de Salmonella Typhimurium aislados de cuyes sanos y enfermos mediante PCR múltiple. 1a) Los productos amplificados obtenidos son SipB (875 pb), SpvB (717 pb), SipA (550 pb) y tolC (161pb). L= Marcador de peso molecular 100 pb. ST= ATCC 14028. 1 - 10= orden de cepas analizadas. El gen cdtB (268 bp) no fue amplificado en ningún caso. 1b) Productos amplificados son SitC (768 pb), lpfc (641 pb), SifA (449 pb), SopB (220 pb) y pefA (157 pb)
Los 10 genes de virulencia evaluados en aislados de S. Typhimurium de cuyes aparentemente sanos y enfermos presentaron altos porcentajes de detección, los cuales se encuentran entre el 66 al 100% y entre 65.6 al 88.9%, respectivamente (Cuadro 2); sin diferencia estadística entre estos grupos de porcentajes de detección de genes de virulencia.
Cuadro 2 Presencia de genes de virulencia (%) y Test exacto de Fisher en aislados S. Typhimurium de cuyes
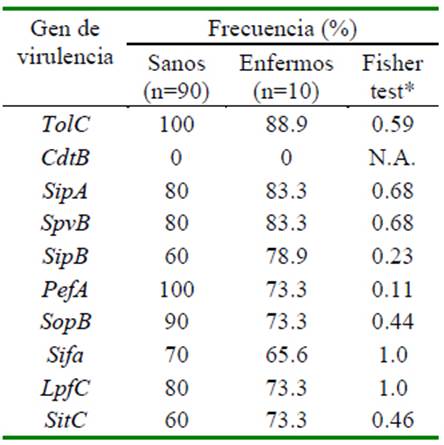
* Nivel de confianza: 95%
N.A.= No Aplicable
Los genes sipB, sitC y sopB ubicados en la Isla de Patogenicidad de Salmonella 1 o SPI-1 (Perret y Zhou, 2013; Kim y Lee, 2017); spiA y sifA ubicados en SPI-2 (Figueroa y Verdugo, 2005; Kim y Lee, 2017) y el gen lpfC, el cual es un operon fimbrial (Sabbagh et al., 2010) se detectaron en un porcentaje variable entre 65.6 y 83.3%; presentándose en mayor frecuencia los genes spiA (83.3%) y sipB (78.9%) dentro de los aislados de cuyes enfermos. En el caso de los aislados de S. Typhimurium de cuyes aparentemente sanos se obtuvieron frecuencias entre 60 al 90%, siendo el gen predominante sopB, el cual se presenta en el 90% de los aislados. Por otro lado, los genes codificados en plásmidos de virulencia, pefA y spvB (Pilla y Tang, 2018) se detectaron en el 73.3 y 83.3% en los aislados de cuyes enfermos, respectivamente, mientras que en los aislados de cuyes aparentemente sanos, pefA y spvB se presentaron en el 100 y 80% de los casos; y, finalmente, el gen tolC, de origen cromosómico, se presentó en mayor frecuencia en todos los aislados, apareciendo en el 88.9% de aislados de cuyes enfermos y en el 100% de aislados de cuyes aparentemente sanos. El gen cdtB, codificado en la Isla de Patogenicidad 11 (SPI-11) en Salmonella Typhi (Sabbagh et al., 2010) no fue identificado en los aislados de este estudio.
Dicha similitud entre cepas de animales enfermos y sanos podría relacionarse con la ubicación de los genes mencionado previamente. A nivel de las diversas Islas de Patogenicidad de Salmonella (SPI), la adquisición de una SPI puede convertir un microorganismo comensal en patógeno; sin embargo, este proceso no es un garante de la transformación del microorganismo, pues la virulencia dependerá tanto de la bacteria receptora y del hospedero (Marcus et al., 2000; Martínez, 2007). En relación a este último, es conocido que cambios a nivel de la microbiota autóctona intestinal, debido a la administración de antimicrobianos, ocasionan que la carga bacteriana potencialmente patógena aumente considerablemente (Casadevall, 2017); de forma similar, los genes localizados en plásmidos de virulencia (Martínez, 2007), se propagan por transferencia horizontal (Pilla y Tang, 2018); sin embargo, se conoce que no todos los miembros de un mismo serovar contienen el plásmido de virulencia, sino que su presentación suele ser variable en serovares con amplio rango de hospederos, como es el caso de Salmonella Typhimurium (Silva et al., 2017).
El relación al gen tolC, cuyo origen cromosómico confiere importancia durante la colonización e infección del hospedero, secreción y exportación de moléculas y toxinas, capacidad innata de multidrogo resistencia, con lo que es esperable su alta frecuencia de detección por tratarse de un gen altamente conservado en todos los serovares de Salmonella (Horiyama et al., 2010; Ricci y Piddock, 2010), donde el knockout del mismo anula su capacidad de supervivencia en la célula hospedera, a pesar que la bacteria aún puede replicarse (Buckley et al., 2006).
No se encontraron diferencias estadísticas en los porcentajes de detección de cada gen de virulencia entre los cuyes aparentemente sanos y enfermos (Cuadro 2); es decir, no hubo asociación aparente entre la presentación individual de los genes de S. Typhimurium con el estado sanitario del cuy, concluyendo que no había relación entre genes de virulencia y estados de salud y enfermedad. Con ello, estos resultados son semejantes a los obtenidos en otros trabajos, tal es el caso de Skyberg et al. (2006) quienes evaluaron el mismo panel de genes de virulencia en S. Typhimurium procedentes de aves de producción sanas y enfermas, sin hallar diferencia entre cepas; o como lo reportado por Tamang et al. (2014), quien es detectaron que más del 96% de aislados analizados de cerdos enfermos y sanos poseían diversos genes de virulencia, incluídos spiA, sipB, tolC, lpfC, sifA, sitC y sopB; sin embargo, la prevalencia de dichos genes en cerdos enfermos se asociaron además a fenotipos multiresisitentes a antimicrobianos, lo cual los convierte en aislados potencialmente más peligrosos que constituyen un riesgo a la salud pública.
Por último, es importante mencionar que Salvatierra et al. (2018) establecieron un solo grupo genético de S. Typhimurium, el cual sigue una dispersión clonal entre los centros de producción intensiva de cuyes en Lima, similar a lo obtenido en este estudio. Por ello, la presencia de un patrón de alta homogeneidad genética de factores de virulencia de Salmonella Typhimurium aislados de cuyes tanto enfermos como aparentemente sanos debería ser tomado en consideración, pues representaría un potencial riesgo para la producción, donde dichos animales podrían actuar como portadores asintomáticos y diseminadores de la enfermedad (Schultz et al., 2018).
CONCLUSIONES
Los aislados de Salmonella Typhimurium presentaron 9 de los 10 factores de virulencia evaluados, con frecuencias variables y mayores al 60%, tanto en cuyes enfermos como aparentemente sanos.
No se encontró diferencia significativa entre los factores de virulencia presentes en aislados de Salmonella Typhimurium provenientes de cuyes enfermos y aparentemente sanos.
No se encontró asociación entre la presentación individual de los factores de virulencia de S. Typhimurium evaluados y el estado sanitario del cuy.












 uBio
uBio