INTRODUCCION
Argopecten purpuratus (Lamarck, 1819) es un molusco bivalvo que se distribuye entre Paita, Perú (5ºS, 81ºO) a Valparaíso, Chile (33ºS, 71ºO) (Avendaño y Cantillánez, 2008); especie hermafrodita funcional cuyas gónadas, tanto femenina como masculina, maduran simultáneamente. La primera madurez gonadal se estima a los 65 mm de longitud de valva, entre 10 a 12 meses de edad (Pazos et al., 1993). Desovan entre 1 a 10 millones de óvulos (Bermúdez et al., 2004; FAO, 2006).
Su ciclo de vida se inicia 24 h pos-fecundación empezando con una etapa larval plantónica, la que puede durar entre 18 a 22 días; una larva trocófora de forma ovalada seguida de una larva umbonada y una última fase larva pediveliger (FAO, 2006). Cuando la larva mide entre 200 a 330 µm se inicia la fijación, asentándose a un sustrato adecua- do, comenzando su existencia bentónica, sien- do el inicio de esta etapa un momento crítico de no encontrar un sustrato o estimulo adecuado en donde fijarse (Bonar et al., 1990; Tapia et al., 1993).
Las señales químicas son fundamenta- les para desencadenar la respuesta del asentamiento larval, inductores artificiales se aso- cian a sustancias neurotransmisoras y sus precursores, sustancias químicas tales como potasio, L-3-3,4-dihidroxifenilalanina, acido r- aminobutírico, colina y sus derivados son inductores naturales que se han caracteriza- do a partir de la respuesta del asentamiento, como consecuencia de la presencia de los sustratos naturales, tales como: individuos coespecíficos, biofilms microbianos y especies presa (Rodríguez et al., 1992). Se define como biofilm o biopelícula a una comunidad de microorganismos que crecen embebidos en una matriz de exopolisacáridos y adheridos a una superficie inerte o un tejido vivo (Donlan, 2002). Los microorganismos colonizadores son muy variados (microalgas, hongos, protozoos y bacterias), siendo las bacterias las dominantes (Sutherland, 2001), y en menor número se encuentran otras macromoléculas como proteínas, ADN y productos diversos procedentes de la lisis bacteriana (Branda et al., 2005).
Los factores bióticos como los biofilms monoespecíficos o multiespecíficos pueden inhibir o incentivar la fijación de larvas de moluscos sobre un substrato (Yang et al., 2013). La respuesta de asentamiento a estos biofilms se genera, al parecer, por la presencia de polisacáridos o glicoproteínas extracelulares unidos a la pared bacteriana (Hadfield, 1986), o bien por compuestos solubles liberados por los biofilms (Bonar et al., 1990). Inductores relacionados a biofilms microbianos son importantes en el asentamiento larval de muchos invertebrados mari- nos bentónicos, habiéndose observado inducción tanto por biofilms de diatomeas y de cianobacterias (Morse A y Morse D, 1984).
Varios investigadores afirman que las larvas de moluscos se asientan en respuesta a un tipo específico de biofilm formado sobre un sustrato (Fitt et al., 1990; Tamburri et al., 1992; Wainman et al., 1996); en cambio otras pueden exhibir una variedad de respuestas frente a la presencia o ausencia del biofilm (Maki et al., 1992). Por su parte, Wolfaart et al. (1994) refieren que los mayores porcentajes de asentamientos inducidos por un biofilm se producen después de su estabilización entre los 14 a 21 días de iniciada su formación. Los microorganismos a menudo construyen y viven dentro de comunidades multicelulares asociadas a superficies conocidas como biopelículas o biofilms. La estructura precisa, química y fisiología de la todas las biopelículas varían con la naturaleza de los microorganismos residentes y medio ambiente local (Branda et al., 2005), y secuestran nutrientes originando estrategias de su- pervivencia bajo condiciones oligotróficas (Decho, 2000).
Riquelme et al. (1995), utilizando gónadas de reproductores de A. purpuratus, aislaron la bacteria Alteromonas haloplan-ktis con capacidad de producir una sustancia antimicrobiana capaz de inhibir tanto bacterias gram positivas como negativas, especial- mente a bacterias patógenas para estas lar- vas, tales como Vibrios anguillarum, V. alginolyticus y Aeromonas hydrophila, las cuales son resistentes a diversos antibióticos. Por su parte, Araya et al. (1999), encontraron en larvas de A. purpuratus 11 cepas con actividad inhibitoria contra el crecimiento de V. anguillarum. Asimismo, Godoy et al. (2011) caracterizaron 46 cepas aisladas e identificadas mediante secuenciación del gen 16S rARN, donde 25 (54%) pertenecieron a la clase Gammaproteobacteria, 11 (24%) Bacilli, 5 (11%) Actinobacteria, 4 (9%) Alfaproteobacteria y 1 (2%) Flavobacteria. Los géneros más abundantes en las muestras de agua fueron Alteromonas, Brevibacterium, Bacillus, Microbacterium y Marinobacter, mientras que, en las de lar- vas de A. purpuratus fueron Pseudo- alteromonas, Bacillus, Marinobacterium, Alteromonas y Vibrio.
Ganesan et al. (2010) aislaron tres cepas bacterianas, Pseudoalteromonas sp, Macrococcus sp y Bacillus sp de Ulva lactuca, de mejillón (Perna canaliculus) y de alícuotas de agua de mar. A partir de ellas se construyó biofilms monoespecíficos y se ensayó la fijación de larvas de Perna canaliculus en estadio pediveliger, encontrando que el asentamiento en los biofilms de Macrococcus sp y Bacillus sp fueron significativamente mayores frente a un control sin biofilm.
El objetivo del presente estudio fue aislar, seleccionar y caracterizar genéticamente bacterianas nativas de Argopecten purpuratus que, a través de un biofilm, estimulen su asentamiento larval.
MATERIALES Y METODOS
El estudio estuvo conformado por cuatro tratamientos, tres con bacterias y uno sin bacterias (control) con tres repeticiones por tratamiento (Cuadro 1).
Muestreo Bacteriológico
Se suspendieron colectores comercia- les de mallas de Netlon® en la Bahía de Sechura, norte del Perú, durante 15 días para tomar tres muestras de 10 cm2 de la malla y una muestra de Ulva lactuca. Las muestras se colocaron en cajas petri esterilizadas y depositadas en una caja térmica a 15 °C para su transporte al laboratorio de bacteriología de la empresa Aquapecten Norte S.A.C., ubicada en Sechura, Piura.
Aislamiento y Cultivo de Cepas Bacterianas
Se utilizó la metodología propuesta por Avendaño-Herrera et al. (2002), para lo cual se colocaron las muestras en tubos falcón con 50 ml de solución salina estéril al 0.85%, agitándose hasta que se desprendió el biofilm, momento en que se retiró la malla y la U. lactuca. Los tubos falcón con la suspensión bacteriana fueron almacenados a 4 °C.
Se hicieron diluciones seriadas de la suspensión bacteriana hasta 10-4. De cada muestra se tomó una alícuota de 100 µl y se sembró por duplicado en placas petri con Agar Tripticasa Soya (TSA) y se incubó durante 24 h a 30 ºC. Se contó el número de unidades formadoras de colonias por mililitro (UFC ml1) y se seleccionaron las colonias más representativas por cada placa. El criterio para la selección de cepas bacterianas fue la dominancia de los morfotipos crecidos en las placas; por pruebas enzimáticas (oxidasa, catalasa, proteasa) y por la taxonomía molecular. Las cepas aisladas fueron suspendidas en caldo (TSB) hasta las pruebas respectivas.
Selección de Cepas
Dominancia de los morfotipos
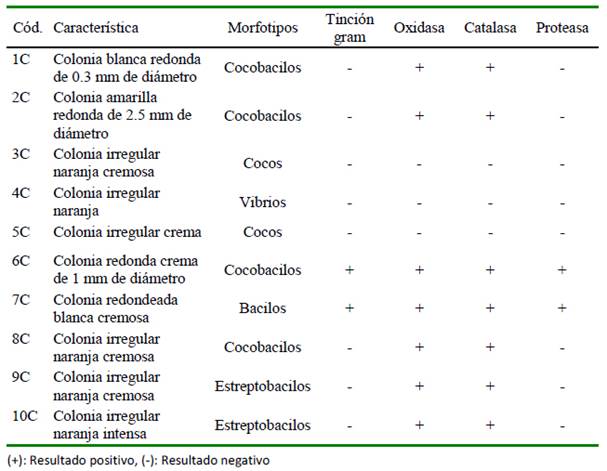
Cada muestra de las cepas aisladas se extendió en una lámina porta objeto y se dejó secar a temperatura ambiente, y luego se fijó con una gota de metanol; se flameó tres veces y se le colocó una gota de violeta de genciana dejando reaccionar por 1 min, luego se enjuagó con abundante agua y se le agregó 2 gotas de lugol dejando reaccionar por 1 min. Se adicionó dos gotas de acetona y se dejó reaccionar por 5 s para luego en- juagar con abundante agua. Se le agregó dos gotas de safranina dejando reaccionar por 1 min. Esta tinción mostró un color rosado rojizo para las bacterias gram negativas. La muestra se lavó con agua y se observó en un microscópico invertido a 100X con aceite de inmersión. Los criterios de selección fueron bacterias gram positivas y que presenten formas de bacilos (cocobacilos, bacilo, diplobacilo y estreptobacilo).
Pruebas enzimáticas
- Oxidasa. Se tomó una muestra de cada cepa aislada y se suspendió a un disco de papel filtro, previamente impregnado en una solución de diclorhidrato de tetrametil-p-fenilendiamina al 1% (reactivo de Kovacs). Se consideraron positivas las que cambiaron a azul púrpura luego de 15 s de colocado el reactivo.
- Catalasa. A cada muestra de las cepas aisladas se les añadió 2 a 3 gotas de una solución de H2O2 al 3%. Se consideraron positivas las reacciones que produjeron efervescencia.
- Proteasa. Las cepas aisladas fueron cultivadas en medio agar sólido enriquecido con leche desnatada bacteriológica (skim milk) al 1% como fuente proteica, e incubadas durante 72 h a 27 ºC. La formación de un halo claro alrededor del crecimiento bacteriano se consideró (+) para producción de proteasa.
Identificación taxonomía molecular
La identificación mediante taxonomía molecular de las cepas bacterianas fue a par- tir del análisis de las secuencias parciales del gen 16S rARN usando la técnica de PCR para el ADN extraído. Las secuencias obtenidas fueron alineadas con las secuencias de la base de datos GenBank (https://www.- ncbi.nlm.nih.gov/genbank/) usando para ello el software online Nucleotide Blast (http:// blast. ncbi.nlm.nih.gov/Blast.cgi) (Zhang et al., 2000), comparándose el porcentaje de similitud existente.
Amplificación del gen 16S rRNA
Se utilizó el kit gSYNC™ DNA Extraction Kit (https://www.geneaid.com/data/files/1605679909234517178.pdf) para amplificar el gen 16S rRNA. Para cada muestra, se adicionó en un microtubo (Eppendorf, Alemania) de 0.5 ml: 31.7 µl de agua ultrapura, 10.0 µl de solución buffer 5X, 4.0 µl de MgCl2, 1.0 µl del mix de dNTP, 1.0 µl del iniciador 27F (5'-AGAGTTTGATCCT GGCTCAG-3') (Lane, 1991 citado por Xie et al., 2007), 1,0 µl del iniciador 1492 R (5'- CGGCTACCTTG TTACGACTT-3'), (Lane, 1991 citado por Xie et al., 2007), 0.3 µl de taq polimerasa, 1.0 µl del ADN bacteriano extraído. La amplificación se realizó en un termociclador Techne FTC3102D, cuya pro- gramación fue: i) Pre-desnaturalización a 95 ºC por 5 min; ii) 40 ciclos de las siguientes etapas: 94 °C por 30 s (desnaturalización), 50 ºC por 45 s (hibridación) y 72 °C por 1 min (polimerización); y iii) polimerización final a 72 ºC por 7 min.
La verificación de la amplificación se hizo al migrar los amplicones en geles de agarosa al 2% conteniendo también 5 µl de bromuro de etidio y como tampón de migración 120 ml de TAE 1X y 2 µl de tampón de depósito y 10 µl de cada amplicón procedentes de la PCR. La migración se realizó a 68 V durante 30 min. Conjuntamente se hizo migrar un marcador de peso molecular de 1000 pb. Los geles fueron visualizados utilizando un transiluminador UVP White/UV para verificar la presencia de ADN.
- Secuenciación del ADN
Se retiró 20 µl de los productos obtenidos de la amplificación por PCR y de los cebadores correspondientes, y se colocaron en tubos eppendorf de 0.5 ml. Las muestras fueron empacadas en hielo seco y enviados a la empresa Macrogen (https://dna.macro- gen.com/eng/member/login.jsp). Los resulta- dos de la secuenciación obtenida las cepas aisladas pasaron a la etapa de la selección.
Impregnación de las Cepas Bacterianas
Se usaron cuatro tanques de 1000 L de capacidad, previamente desinfectados con cloro (20%) y llenados con 600 L de agua de mar filtrada a 0.5 µm y esterilizada por UV. Se adicionó mediante el Medio Guillar f/2 los macronutrientes (1.0 ml L-1) y micronutrientes (1.0 ml L-1), así como las vitaminas biotina y tiamina (0.5 ml L-1), metasilicato (0.5 ml L-1) y las cepas bacterianas nativas, teniendo en cuenta los tratamientos T1 (tanque con adición de cepa 1: Oceanobacillus), T 2 (tan- que con adición de cepa 2: Bacillus), T3 (tan- que con adición de cepa 1 y 2: Oceano- bacillus y Bacillus) y T0 (tanque sin adición de cepas). La concentración de las cepas adicionadas fue de 1 x 104 células ml-1. En cada tanque se sumergieron en forma vertical 240 colectores de malla Netlon® durante 6 días. Las mallas fueron previamente des- infectadas con cloro (20%).
Preparación de los Tanques
Se usaron 12 tanques de 10 000 L en los que se distribuyeron los cuatro tratamientos con sus tres repeticiones. Los tanques fueron desinfectados con cloro (20%) y se llenaron con agua de mar filtrada a 1.0 µm y esterilizada por UV. Además, se les instaló una piedra difusora para dar aireación continua. Seguidamente se suspendieron 80 colectores impregnados con las cepas bacterianas aisladas, según tratamiento. A todos los tanques se les incorporó microalgas (Isochrysis galbana, Chaetoceros calcin-trans, Chaetoceros gracilis, Pavlova lutherii y Tetraselmis suecica) a concentración de 30 000 células ml-1.
Selección de las Larvas Competentes
Las larvas de 22 días pos-eclosión fueron seleccionadas utilizando un tamiz de 200 µm con el que se obtuvo larvas de 225 a 280 µm de longitud anteroposterior. Las larvas seleccionadas fueron depositadas en recipientes con 3 L de agua de mar microfiltrada (0.22 µm) y esterilizada mediante UV. Se colocó 1 ml de la muestra en placas petri para su observación al estereoscopio, seleccionándose aquellas que mostraron características de larvas competentes, según lo descrito por Tapia et al. (1993); y las características morfolgicas descritas por Tamburri et al. (1992). Se consideró el desplazamiento de las larvas, la presencia de mancha ocular, el tamaño de larva (250 µm), la presencia y desarrollo del pie. Las larvas seleccionadas fue- ron depositadas en los tanques experimenta- les, a una concentración de 1 larva/ml.
Asentamiento Larval
Después de 72 h se sacudieron los colectores en baldes de 10 L. Se tomó una muestra de 100 ml en una placa petri para su conteo con la ayuda de un estereoscopio. Se determinó el número de larvas asentadas en cada uno de los colectores. Los resultados se extrapolaron al total de larvas asentadas por colector. Se consideró como larva asentada aquella que permaneció adherida al sustrato desde su traslado del tanque experimental a una placa petri para su conteo.
RESULTADOS
Aislamiento y caracterización de las cepas bacterianas nativas
Se logró aislar 5 cepas (1C al 5C) de las muestras de Ulva lactuca y 5 cepas de la malla Netlon® (6C al 10C), con una con- centración de 1.23x104 y 2.24x104 UFC ml-1, respetivamente. Se caracterizaron cinco morfotipos (cocobacilos, cocos, vibrios, baci- los, estreptobacilos) y frente a la tinción se observó una predominancia de bacterias gram negativas, excepto las cepas 6C y 7C. En la prueba de oxidasa y catalasa las cepas 3C, 4C y 5C tuvieron una reacción negativa, mientras que las demás cepas reaccionaron positivamente. En el caso de la prueba de proteasa solo las cepas 6C y 7C presentaron reacción positiva (Cuadro 2).
Identificación molecular y selección de las cepas bacterianas nativas
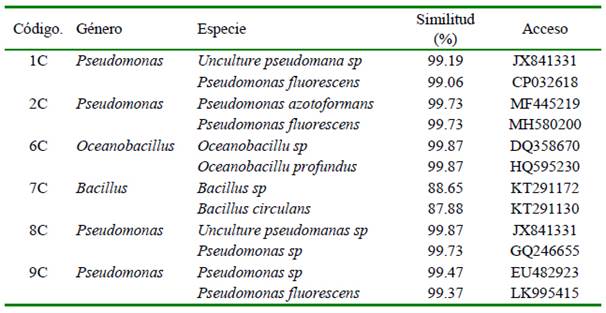
De las 10 cepas bacterianas nativas aisladas, a través del programa BLAST, solo se pudo amplificar a siete de ellas, identificándose a nivel de género a tres de ellas: Pseudomonas, Oceanobacillus y Bacillus. Para el género Pseudomonas fueron las cepas 1C, 2C, 8C, 9C y 10C, siendo las especies P. fluorescens, P. azotoformans, P. tolaasii, Pseudomonas sp y una especie no cultivable (Pseudomonas sp) (Cuadro 3). La cepa 6C fue identificada como parte del gé- nero Oceanobacillus con las especies Oceanobacillus sp y Oceanobacillus profundus; la cepa 7C como parte del género Bacillus, siendo las especies Bacillus sp y Bacillus circulans. Estas especies fueron seleccionadas para la fijación de larvas de A. purpuratus.
Asentamiento larval de A. purpuratus
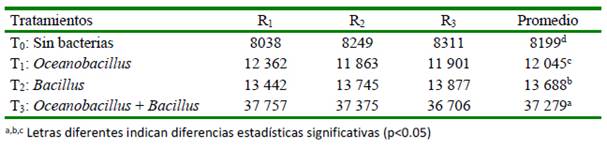
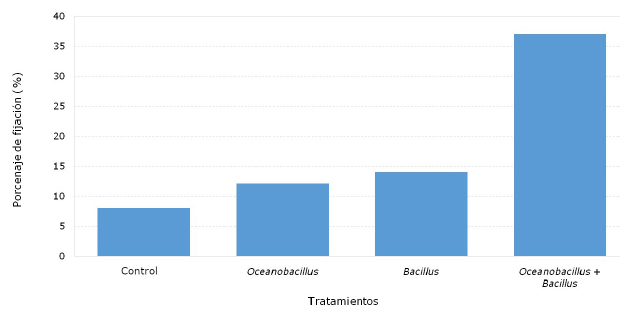
El Cuadro 4 muestra el promedio del asentamiento larval de A. purpuratus para cada tratamiento. Los promedios son significativamente diferentes entre sí (p<0.05). La Figura 1 muestra el asentamiento larval de A. purpuratus, observándose que la combinación de ambas cepas presentó un mayor porcentaje de fijación (37%) frente al tratamiento control (8%).
DISCUSION
Las cepas bacterianas nativas aisladas del cultivo de A. purpuratus fueron mayor- mente Pseudomonas, seguido de Oceanobacillus y Bacillus, lo que se contrasta con los resultados reportados por Diringer et al. (2015) al caracterizar por metagenómica la microbiota asociada a A. purpuratus de la Bahía de Sechura determinando bacterias del género Pseudomonas en las vísceras y bacterias del género Bacillus en la hemolinfa; así mismo Godoy et al. (2011) encontraron bacterias del género Bacillus.
Diversos estudios han demostrado que las larvas de moluscos se asientan en res- puesta a un tipo específico de biofilm; sin embargo, las bacterias pueden inhibir o incentivar la fijación de larvas de moluscos sobre un substrato (Yang et al., 2013). En este sentido se evaluó el efecto del biofilm formado por las cepas Oceanobacillus y Bacillus por separado y en combinación sobre la fijación de larvas de A. purpuratus, donde los resultados demuestran que los biofilms monoespecíficos formados por las cepas bacterianas ensayadas estimularon la fijación de larvas de A. purpuratus, a diferencia del tratamiento control sin las cepas bacterianas; sin embargo, en larvas de Balanus amphitrites pueden no tener efecto (Maki et al., 1992). En el presente estudio se demostró que los biofilms formados a partir de cepas bacterianas de Oceanobacillus y Bacillus tienen una mayor fijación (12 y 14%, respectivamente) frente al tratamiento control (8%), pero al combinar las dos cepas mejoró la capacidad de fijación en 37%. En el trabajo de Ganesan et al. (2010), los biofilm formados por Pseudoalteromonas sp no indujeron la fijación, mientras que los forma- dos por Macrococcus sp y Bacillus sp lo hicieron (50 y 60%, respectivamente). Estos resultados confirman que los biofilms formados por cepas del género Bacillus ayudan en la fijación de larvas de A. purpuratus mejorando el asentamiento de semillas de este molusco.
CONCLUSIONES
Se aislaron 10 cepas bacterianas nativas de Ulva lactuca y de la malla Netlon® colectadas en la Bahía de Sechura, Piura y se pudo caracterizar a siete géneros, entre los que predomina- ron Pseudomonas, Oceanobacillus y Bacillus.
Los biofilms formados a partir de cepas de Oceanobacillus y Bacillus por separado incrementaron el asentamiento larval de A. purpuratus en 12 y 14%, respetivamente, mientras que al estar asociadas el incremento fue de 37%.












 uBio
uBio