INTRODUCCIÓN
La ganadería ovina es una de las actividades más importante para la población rural en especial en las zonas altoandinas del Perú (INEI, 2013). En la década de 1950 la División Ganadera de la empresa Cerro de Pasco Corporation inició la formación de una nueva raza ovina rústica y adaptada a las condiciones ambientales de la sierra central peruana. La base para la formación del ovino Junín comprendió borregas criollas que fueron cruzadas inicialmente con carneros Corriedale de origen chileno (Snyder, 1959; Narvaez et al., 2005). Las borregas, producto de estos cruces, formaron cuatro grupos para ser empadradas, de forma selectiva y cerrada, con reproductores de dos razas recíprocas norteamericanas (Columbia y Panama) y el tipo Warhill (Wyoming, USA) y Corriedale de distintos orígenes (norteamericano y Nueva Zelanda) (Snyder, 1959). Los animales de estos cruces fueron posteriormente seleccionados y apareados siguiendo el método Neale de la Universidad de Nuevo México y consistente en cruzar animales de la misma calidad fenotípica (Snyder, 1959; Narvaez et al., 2005; Rojas, 2014). La aplicación de este método y el manejo del rebaño genéticamente cerrado utilizando selección masal con apareamientos selectivos y/o correctivos llegó a incluir 14 líneas hasta fusionarlas en una sola línea, llegando en menos de 10 años a formar el Tipo Junín (Snyder, 1959; Gamarra y Villarroel, 1978).
Las características de este animal fueron identificadas posteriormente por la Dra. H.N. Turner del Commonwealth Scientific and Industrial Research Organization (CSIRO) de Australia en 1973 que finalmente derivó en el reconocimiento de Raza Junín (Rojas, 2014). El ganado Junín es un ovino de doble propósito (carne y lana), posee robustez, conformación equilibrada y adaptación al pastoreo en zonas altoandinas a campo abierto, donde es criada y se encuentra aclimatada para la región de los Andes entre 3500 a 4500 msnm (Rojas, 2014).
El proceso de selección fue iniciado por la entonces División Ganadera de la Cerro de Pasco Corporation durante las décadas del 50 y 60 y que se han mantenido hasta la fecha por la Sociedad Agrícola de Interés Social (SAIS) Tupac Amaru Ltda. N.°1, quienes son depositarios de más 100 mil ejempla- res de esta raza. Con esta población, la SAIS Tupac Amaru inició en 2010 el registro genealógico oficial de estos animales para la formación del Libro de Pedigrí de la raza Junín (Rojas, 2014). No obstante, a pesar de la importancia económica para el país y como re- curso genético a conservar no existen estudios respecto de la variabilidad genética ni estructura poblacional del ovino Junín. El conocimiento de la diversidad genética de las poblaciones domésticas es importante para la implementación de planes de mejoramiento exitosos y para la conservación del recurso genético (FAO, 2015; Fabbri et al., 2019).
El uso de secuenciamiento de alto rendimiento para la caracterización genética de animales domésticos se ha vuelto común en la última década gracias al abaratamiento de la tecnología (Eusebi et al., 2020). El uso de microarreglos de alta densidad de marcadores polimórficos de nucleótido simple (SNP) permite estudiar miles de marcadores de este tipo en todo el genoma, lo cual permite caracterizar la diversidad genética, evolución y adaptación de los animales domésticos con una mayor precisión que con otros marcado- res como el uso de microsatélites (Lenstra et al., 2012).
En el presente trabajo se evaluaron 20 animales del plantel de ovinos de raza Junín, usando microarreglos de alta densidad de más de 600 mil marcadores de tipo SNP para estudiar la diversidad y la estructura poblacional de ovinos de esta raza. Asimismo, se realiza- ron comparaciones de la variabilidad genética encontrada con lo reportado para otras razas de ovinos de distintos orígenes.
MATERIALES Y MÉTODOS
Lugar de estudio y muestras
Se colectaron muestras de sangre de 20 ovinos de raza Junín de la SAIS Tupac Amaru Ltda. N.° 1 ubicada en el distrito de Canchayllo, provincia de Jauja, región Junín, Perú, en septiembre de 2019. Los animales (10 hembras y 10 machos) pertenecían a la clase superior (Plantel) de la Unidad de Producción Pachacayo. La sangre fue colectada por punción de la vena yugular o cefálica en tubos con EDTA y almacenado a 4 °C hasta el análisis genético. Adicionalmente se muestrearon 10 ovinos de raza Corriedale de la provincia de Junín a fin de comparar la diversidad genética de ambas poblaciones.
Secuenciamiento de microarreglos de SNPs
Las 30 muestras fueron genotipificadas usando el microarreglo de SNPs Ovine600Kv1 (Illumina). Este microarreglo contiene 606 006 marcadores SNPs distribuidos a lo largo de todos los cromosomas de la oveja, incluidos los cromosomas sexuales y mitocondrial.
Base de datos SNP de ovinos
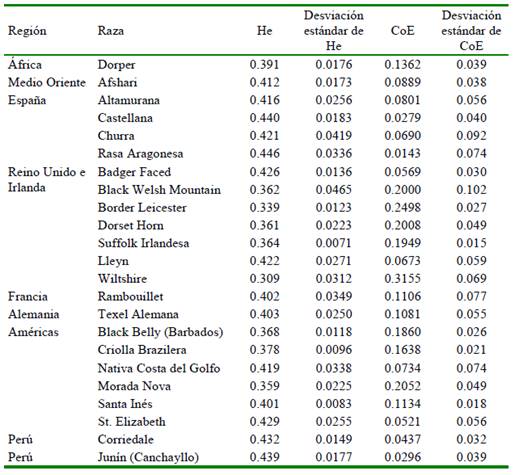
A fin de poner los datos genómicos del ganado Junín en un contexto amplio en comparación a otras razas de ovejas, se descargaron los datos genómicos públicos de Kijas et al., 2012 y Beynon et al., 2015 que contienen información de SNP de 21 razas de ovejas: África (Dorper), Medio Oriente (Afshari), España (4 razas: Altamurana, Castellana, Churra y Aragonesa), Reino Unido e Irlanda (7 razas: Badger Faced, Black Welsh Mountain, Border Leicester, Dorset Horn, Suffolk Irlandesa, Lleyn y Wiltshire), Francia (Ramboullet), Alemania (Texel Alemana), las Américas (6 razas: Black Belly de Barba- dos, Criolla Brazilera, Nativa del Golfo, Morada Nova, Santa Inés y St. Elizabeth). Se incluyó la información generada en este estudio para la raza Junín y de la raza Corriedale (ambos de Perú) y se conformó una base de datos final de 23 razas con 862 individuos. Dado que los datos públicos de las 21 razas fueron genotipadas usando el microarreglo de marcadores tipo SNP Ovine50K que contiene 50 mil marcadores SNP, se filtraron los marcadores comunes a ambos microarreglos (Ovine50K y Ovine600Kv1) y se obtuvieron 42 000 marcadores compartidos que fueron tomados en cuenta en adelante.
Se utilizó el software Plink v. 1. 9 (Purcell et al., 2007) para obtener marcadores con bajo desequilibrio de ligamiento. Los datos se filtraron asumiendo ventanas de 50 SNPs donde se calculó el desequilibrio de ligamiento entre todos los pares de SNPs y se descartó un SNP al azar cuando la correlación entre los alelos de los dos SNPs era mayor al 1%. Las ventanas para este cálculo se deslizaron a lo largo de cada cromosoma dando un salto cada 10 SNPs. Se removieron aquellos SNPs cuando el alelo de menor frecuencia tenía una frecuencia de 1% o menos, lo que dejó un total de 8176 SNPs genotipificados en 862 individuos de las 23 razas.
Determinación de valores genéticos poblacionales y análisis de estructura poblacional mediante análisis de escala multidimensional (MDS)
Se calculó la heterocigosidad esperada para cada cromosoma de la raza Junín usando el programa Plink v. 1.9. Del mismo modo, se evalúo la presencia de equilibrio de Hardy- Weinberg mediante un test exacto de Fisher entre el número de alelos observados y esperados para cada locus. Para visualizar la estructura poblacional de las razas analizadas se llevó a cabo un análisis de escala multidimensional (MDS) usando PLINK v1.9 y el paquete BITE (Milanesi et al., 2017) en el ambiente R.
RESULTADOS
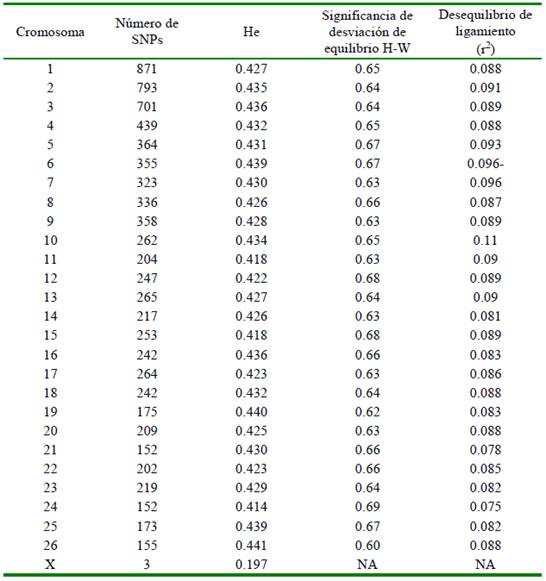
La raza Junín presenta una heterocigosidad esperada de 0.429 (± 0.007) estimada a partir de 8173 SNPs de los cromosomas autosómicos del 1 al 26. El cromosoma X estaba representado por solo 3 SNPs y por lo tanto no fue incorporado en estos análisis. En promedio el número de marcadores por cromosoma fue de 314 (± 190) variando desde un máximo de 871 (cromosoma 1) a un mínimo de 152 (cromosoma 24) (Cuadro 1). Ninguno de los autosomas presentó desviación del equilibrio Hardy-Weinberg (valores de la probabilidad de significación de p>0.05). El desequilibrio de ligamiento por cromosoma fue bajo en promedio (8 ± 0.6%).
La variabilidad genética de las 23 razas de ovejas fue bastante similar, aunque algunas razas como la Wiltshire (Reino Unido) presentaron niveles relativamente bajos de heterocigosidad esperada. La raza Junín presenta una mayor heterocigosidad esperada que 10 de las otras razas incluyendo todas las británicas, pero menor que la heterocigosidad esperada de las razas españolas, francesa y alemana. Aun así, la raza Junín presenta un menor coeficiente de endogamia que todas las otras razas analizadas (Cuadro 2).
Cuadro 1 Valores de heterocigosidad esperada (He), desviación del equilibrio de Hardy Weinberg (H-W) y desequilibrio de ligamiento para los cromosomas del ovino Junín
El análisis de estructura poblacional utilizando el análisis de escala multidimensional (MDS) encontró que la mayor divergencia entre las razas fue de 5.5%, lo que es consistente con la literatura de ovinos utilizando microsatélites, SNPs y genomas completos. Los animales de la raza Junín se agruparon unos con otros reflejando la uniformidad de la raza. Además, estos individuos se super- pusieron parcialmente con los individuos de la raza Corriedale muestreados en Junín (Perú) y con animales de la raza Nativa Costa del Golfo (Figura 1). Los individuos de la raza Junín también quedaron más cerca de individuos Criollos Brasileros que de las razas españolas, resaltando que la raza Junín tiene un componente criollo en sus antecedentes genéticos.
DISCUSIÓN
El ovino Junín es una raza formada en el Perú mediante selección genética masal utilizando a ovejas criollas y cruzadas progresivamente con reproductores machos de hasta cinco razas procedentes del oeste norteamericano. La selección genética selectiva y cerrada se inició hace 50 años y a pesar de su importancia en la ganadería de la sierra central peruana poco se conoce sobre su genética y diversidad. En este trabajo se usaron microarreglos de SNP de alta resolución para estudiar la diversidad genética de la raza. Se encontró que los valores de variabilidad en el ganado Junín son muy similares a otras razas (Kijas et al., 2012; Beynon et al., 2015). La heterocigosidad esperada calcula- da para la población del ovino Junín fue de 0.429 y con un bajo coeficiente de endogamia sugiriendo una buena variabilidad genética para esta población. La alta diversidad genética sugiere el buen manejo reproductivo de los rebaños, seguramente como resultado del mejor control genealógico emprendido en la última década.
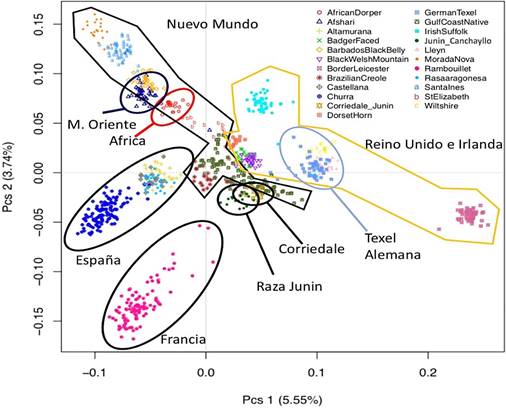
Figura 1 Gráfico de escala multidimensional a partir de marcadores SNP para 862 ovinos de 23 razas incluyendo la raza Junín. Los 20 individuos de raza Junín presentan una estructura genética particular que lo diferencia de otras razas
En el análisis de escalas multidimensionales para las 23 razas en estudio se observó un agrupamiento de la variabilidad de la población de ovino Junín, lo que sugiere una identidad de la raza. Este estudio presenta por primera vez evidencia molecular de la individualidad de la raza Junín frente a otras razas. Esta individualidad tal vez refleje la participación de tres razas norteamericanas muy pocas veces utilizadas en mejora- miento genético en otras regiones del país. Se observa una cercanía de la población del ovino Junín con la raza Corriedale, raza predominante en el Perú y base inicial de las borregas (criollas vs Corriedale) y con las poblaciones de ovino criollo de Brasil, de ascendencia de ovejas españolas. Estos resultados son esperados dado que el ovino criollo peruano y el ovino Corriedale fueron usados para la formación de la raza Junín, de allí que comparten par- te de esa diversidad genética (Rojas, 2014).
El ovino Junín es un importante recurso genético de los Andes peruanos, pues es la tercera población existente en el Perú adaptada para zonas altoandinas, con buen rendimiento de carne y lana. La información brin- dada en este trabajo puede servir como base para implementar medidas de conservación necesarias para asegurar un desarrollo ganadero del ovino Junín compatible con el uso sostenible de la diversidad genética (FAO, 2007). La falta de viabilidad económica de los sistemas de producción ganadera y a la destrucción de los hábitats de las razas ganaderas nativas, como acontece en la ganadería andina actual, puede llevar a la pérdida de la diversidad genética y acelerar la erosión genética como ocurre con casi 30% de razas de ganado de todo el mundo, en especial en países en vías de desarrollo (FAO, 2015; Zhang et al., 2018). Por ello es importante conocer la diversidad genética de las razas ganaderas adaptadas a la geografía peruana como el ovino Junín y las poblaciones criollas de ovinos para conservarlas y asegurar el recurso genético para el futuro.












 uBio
uBio