INTRODUCCIÓN
Los animales de laboratorio utilizados en investigación biomédica o para la producción de reactivos biológicos requieren estar libres de patógenos debido a que pueden alterar sus características fisiológicas (Baker, 1998; Nicklas, 2002). Escherichia coli no se considera como una bacteria que ocasione enfermedades comunes en estos animales (Baker, 1998), existiendo escasa información sobre la presencia de E. coli patógena en la microbiota de los animales de bioterio.
E. coli es una bacteria con diversas variantes que normalmente se encuentran colonizando el intestino del humano y de los animales (Majowicz et al., 2014), formando parte de la biota, ya que han logrado una simbiosis en los procesos metabólicos e incluso algunas cepas sintetizan cofactores y protegen al hospedero de la invasión por microorganismos patógenos (Jang, 2017). No obstante, hay algunas cepas de E. coli que, debido a mecanismos de intercambio de material genético, han adquirido la capacidad de ocasionar infecciones entéricas. Estas se clasifican en seis patotipos diarrogénicos: enterotoxigénica (ETEC), enterohemorrágica (EHEC), enteroinvasiva (EIEC), enteropa-tógena (EPEC), enteroagregativa (EAEC) y de adherencia difusa (DAEC) (Croxen et al., 2013). Además, se encuentran diversos patotipos asociados con infecciones extraintestinales en las que se incluyen cepas causantes de infección de vía urinarias conoci- das como uropatógena (UPEC), otras más responsables de meningitis (MNEC) en recién nacidos y clonas involucradas en cuadros de septicemia (SEPEC) y patógenas de aves (APEC) que también pueden causar infecciones urinarias (Kaper et al., 2004).
Ensayos de enzimas multilocus primero y posteriormente de secuencias multilocus integraron a E. coli en diferentes filogrupos. Clermont et al. (2013) utilizando un ensayo de PCR múltiplex en el que se identifican algunos genes específicos lograron establecer un procedimiento práctico para definir los filogrupos de la bacteria (A, B1, B2, C, D, E y F). Con dicho procedimiento se corroboró que los filogrupos B2, D, E y F corresponden a cepas patógenas, mientras que los grupos filogenéticos A, B1 y C integran a las cepas comensales que constituyen la microbiota del intestino (Clermont et al., 2013; Leimbach et al., 2013).
La barrera entre simbiosis y virulencia es el resultado de un complejo balance entre el estado del hospedero y la presencia y ex- presión de factores de virulencia en la bacteria (Pérez-Brocal et al., 2013). Esto adquiere importancia en E. coli debido a que puede presentar diferentes patotipos, filogrupos y serotipos que se relacionan con la etiopatogenia de varias enfermedades en hu- manos, algunas de las cuales pudieran ser de origen zoonótico. Ante ello, se requiere evaluar los animales que se utilizan en investigación con el interés de verificar en forma periódica el posible ingreso de nuevos microorganismos que puedan introducir cambios o variables en los reactivos de investigación. Es por lo que el objetivo del presente trabajo fue conocer si en la microbiota intestinal de ratas Wistar de una colonia de bioterio se encontraban cepas de E. coli patógenas.
MATERIALES Y MÉTODOS
Las muestras se obtuvieron de una población de trece ratas Wistar (Rattus norvegicus) pertenecientes a un bioterio nivel 1 ubicado en la zona centro de México. el cual durante ese periodo no contaba con ISO o certificación en calidad ante alguna institución normativa en esta materia; sin embargo, el bioterio se regía en altos estándares de calidad impuestos por los manuales y reglamentos internos, así como las Buenas Prácticas de Laboratorio (BPL) dado que se trata de un bioterio de barrera. Los individuos se muestrearon siguiendo la Norma Oficial Mexicana NOM-062-ZOO-1999 y el reglamento de bioseguridad del bioterio. Los animales se mantuvieron alojados en cajas (modelo GR900, Techniplast) con ventilación individual y filtros HEPA (High Efficiency Particulate Air). La temperatura del lugar de alojamiento era controlada dentro de un rango de 20 a 23 °C y humedad relativa entre 50 y 60%. El material de cama y el agua se esterilizaba mediante autoclave y el alimento PicoLab Roden Diet 20 5053 (LabDiet) es- taba irradiado. El agua y alimento se ofrecía ad libitum.
En el lapso en el que fue realizado el estudio, las ratas se encontraban aisladas en un cuarto específico para la estabulación de esta especie y cepa. En otras salas igualmente aisladas se encontraba una colonia de ratones C57BL/6. Los equipos, materiales, insumos y alimentos de cada grupo eran específicos para la especie. El fin de estos animales y cepas era la investigación biomédica y producción.
Se tomaron muestras de heces de la zona del ano utilizando hisopos estériles impregnados con solución salina fisiológica (SSF), considerándose suficiente la cantidad colectada de heces por este medio. Los hisopos se depositaron en medio de transporte de agar Stuart y se refrigeraron hasta su siembra en un periodo menor a 24 h. Las muestras se sembraron en agar MacConkey y se incubaron durante 18 h a 37 °C. Posterior al crecimiento se seleccionaron al azar 5 colonias por muestra (3 lactosas positivas y 2 lactosas negativas). Se obtuvieron 39 colonias lactosas positivas y 26 lactosas negativas, dando un total de 65 aislamientos. Las cepas aisladas se identificaron por medio de pruebas bioquímicas para fermentación de glucosa- lactosa, indol, VP, urea, malonato, fenil alanina, motilidad, y producción de ácido sulfhídrico.
A las cepas identificadas como E. coli se les extrajo DNA por ebullición, tomándose el sobrenadante. El DNA fue cuantificado en un espectrofotómetro NanoDrop 2000 (Thermo Fisher Scientific, USA). Se utilizó la técnica de la PCR para identificar si pre- sentaban genes asociados a los patotipos ETEC, EPEC, EIEC, y EHEC patógenos para humanos. Las condiciones para la PCR se realizaron según lo reportado por López- Saucedo et al. (2003) con modificaciones propias. Se desarrolló la amplificación de los genes de manera individual, utilizando 2 µl de PCR Master Mix 2X (Thermo, Acientífica USA), 0.5 µM de cada iniciador, 0.5 µg de ADN molde, ajustando a un volumen de 20 µl con agua libre de nucleasas. Las condiciones de amplificación fueron: desnaturalización inicial a 95 ºC (1 min, 1 ciclo), 30 ciclos a 95 ºC, alineamiento 55 °C y solo para st fue de 48 °C y una extensión a 72 ºC (30 s por cada temperatura), una extensión final a 72 ºC por 5 min. Como controles positivos para el patotipo ETEC se utilizó la cepa H10407 (lt, st), para EHEC EDL 933 (eaeA, stx1, stx2), EIEC O136: NM (ial) y para EPEC la cepa E2348 / 69 (bfpA, eaeA), y un control negativo de solo agua.
Para identificar los filogrupos de las cepas de E. coli se aplicó la técnica de PCR multiplex (Clermont et al., 2000). Las PCR se llevaron a cabo en el termociclador MiniAmp (Applied Biosystems, USA). Los productos de ambos PCR se corrieron en geles de agarosa en TBE al 1% y se tiñeron con Gel Red TM Nucleic acid Gel Stain, 10000X en agua, y se visualizaron en un transiluminador de rayos UV (UVP, USA). El tamaño de las amplificaciones se determinó por comparación con el marcador de talla molecular escalera de 100 pb (Thermo Scientific, USA).
Para los ensayos de formación de biopelículas se utilizó el protocolo descrito por de Oliveira-García et al. (2003) con modificaciones propias utilizando las cepas de E. coli, O42 (OND:H10) enteroagregativa y la cepa HB101 (E. coli K12) como controles positivo y negativo respectivamente. Las células tratadas se tiñeron con cristal violeta al 1% y por medio de un espectro se determinó la formación de biopelículas a 630 nm de densidad óptica (DO), comparando con los con- troles negativos.
La susceptibilidad bacteriana a los antimicrobianos se evaluó realizando una sus- pensión bacteriana de E. coli en SSF con una densidad igual al tubo N.° 0.5 del Nefelómetro de McFarland, se sembró en medio Müller- Hinton, se colocaron sensidiscos con 15 antibióticos (Oxoid, UK) (Cuadro 1) y se in- cubó durante 18 a 24 h a 37 °C en aerobiosis. La lectura se realizó midiendo el diámetro del halo de inhibición y para la interpretación se utilizaron los criterios del manual del Clinical and Laboratory Standards Institute (CLS, 2018).
RESULTADOS
De las 13 ratas se aislaron 65 cepas que se identificaron y conservaron; sin embargo, al sembrarlas nuevamente para su caracterización solo se recuperaron 40 cepas que fue- ron incluidas en el estudio de caracterización. Utilizando un esquema de pruebas bioquímicas más completo se pudo definir que 26/40 cepas (65%) correspondieron a E. coli, 9 (22.5%) a Proteus mirabilis, 4 (10%) a Citrobacter freundii y una (2.5%) a Proteus vulgaris. Las cepas de E. coli que fueron caracterizadas provinieron de ocho ratas (61.5%).
El ensayo de PCR para definir la presencia de genes de virulencia en las cepas de E. coli recuperadas mostró que ninguna de ellas amplificó los marcadores genéticos que las asociara con algún patotipo de cepas causantes de diarrea (EPEC, EAEC, EIEC, ETEC y EHEC). Sin embargo, el ensayo para conocer el filogrupo mostró la amplificación de chuA y yjaA, genes que permiten incluir- las en el grupo B2 de cepas responsables de infecciones extraintestinal (ExPEC). Por otro lado, en todos los ensayos de caracterización los controles positivos amplificaron para los productos esperados.
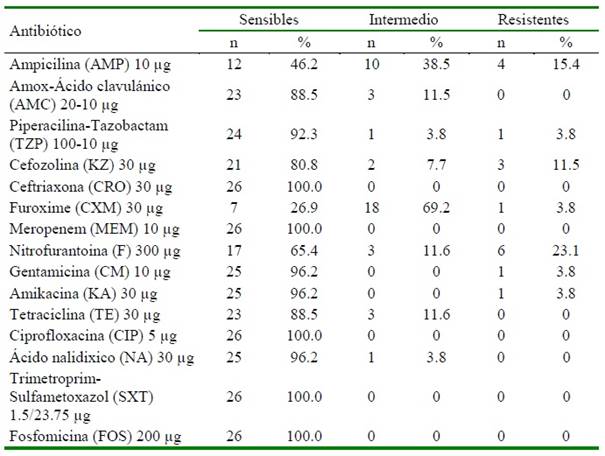
Cuadro 1 Susceptibilidad antimicobiana de cepas de E. coli aisladas de 13 ratas Wistar (Rattus norvegicus) pertenecientes a un bioterio nivel 1 de la zona centro de México
Las cepas de E. coli caracterizadas como pertenecientes al filogrupo B2 no formaron biopelículas en los dos tiempos de incubación (24 y 48h), presentando una DO similar a la del control negativo (24h 0.01, y 48h 0.0032). Por otro lado, la lectura a las 24h y 48h de incubación para el control positivo de la cepa O42 presentó una DO de 0.034 y de 0.083, respectivamente.
El ensayo de susceptibilidad a los antimicrobianos de las 26 cepas identificadas como E. coli mostró que 6/26 (23%) fueron resistentes a alguno de los antibióticos analizados (Cuadro 1). Los antibióticos a los que presentaron mayor resistencia fueron la nitrofurantoina (23.1%, [6 cepas]), ampicilina
(15.4%, [4 cepas]) y cefozolina (11.5%, [3 cepas]). Solo una cepa presentó multiresistencia a cinco antibióticos, tres a tres antibióticos, una a dos y una a solo un antibiótico.
DISCUSIÓN
Para conocer la calidad de los animales de laboratorio y definir si pudieran ser de utilidad para algunos ensayos es importante caracterizar su microbiota intestinal. E. coli es un integrante de dicha microbiota; sin embargo, la adquisición de genes relacionados con virulencia, incluidos los asociados a resistencia a los antimicrobianos, en su proceso evolutivo ha dado lugar a la emergencia de clonas patógenas responsables de diferentes cuadros clínicos, de allí que su presencia en animales de laboratorio podría convertirse en un riesgo para la salud pública (Puvaca, 2021). En el presente estudio, si bien ninguna de las cepas estudiadas presentaba genes de virulencia relacionados con alguno de los patotipos asociados con enfermedad en humanos o animales, fue interesante observar que amplificaron chuA y yjaA, genes asocia- dos con el filogrupo B2 relacionado con cepas causantes de infecciones extraintestinales (Khairy et al., 2019). Al respecto este filogrupo se ha aislado de caballos en Japón donde las cepas mostraron patrones de multiresistencia (Sato et al., 2020), similar a lo observado en este estudio.
La multiresistencia a antibióticos es un problema de actualidad que cada vez adquiere mayor relevancia (OMS, 2019). Es por ello que se requieren estudios que muestren el comportamiento de bacterias de animales y humanos e implementar nuevas alternativas para el manejo de las enfermedades infecciosas y el control más estricto del empleo de antimicrobianos, tanto en humanos como en animales (Hernando-Amado et al., 2019). No obstante que en el presente estudio el porcentaje de cepas de E. coli resistentes a antibióticos fue de 23.1% (6 cepas) y solo una mostró multiresistencia (3.8%), estos resultados deben de ser considerados de importancia ya que los animales del estudio forman parte de un ecosistema donde interactúan con seres humanos y pueden ser la fuente de transmisión de estos patógenos con resistencia a los antimicrobianos (Hernando-Ama- do et al., 2019).
Los resultados obtenidos plantean preguntas a responder sobre el origen de la resistencia de las bacterias aisladas en anima- les de un bioterio que cuenta con protocolos de bioseguridad y cuya población son parte de una colonia cerrada. Al respecto, Bengtsson-Palme et al. (2018) refieren a cepas resistentes influenciadas por las condiciones del ambiente y las características específicas del microorganismo que presentan la capacidad de adquirir genes con mayor facilidad, lo que favorecería la facilidad de adaptación de la bacteria en reservorios con los que el humano tiene gran interacción. Es por ello la relevancia de definir si las bacterias de humanos se han adaptado a vivir en los animales y estos ser la fuente de transmisión y, por ende, de la etiología de diferentes padecimientos cada vez más difíciles de controlar.












 uBio
uBio 

