INTRODUCCIÓN
La salmonelosis constituye una de las ETA (Enfermedades transmitidas por alimentos) más comunes en el mundo, la cual afecta tanto a los animales como a seres humanos. Se reconoce que las aves de corral y el ganado vacuno y porcino constituyen sus principales reservorios, así como los productos cárnicos que derivan de ellos, los cuales en conjunto representan un riesgo para la salud pública (Gutiérrez et al., 2008).
La salmonelosis es una zoonosis causada por bacterias del género Salmonella, la cual comúnmente está asociada a cuadros diarreicos en humanos. Los principales agentes se encuentran dentro de las especies Salmonella enterica y Salmonella bongori; las cuales, a su vez se subdividen en más de 2500 serovariedades, donde predominan los serovares de la subespecie enterica en los aislados de muestras clínicas (Grimont y Weill, 2007; Caffer et al., 2008). Las serovariedades no tíficas de Salmonella spp cuentan con una amplia distribución a nivel mundial y representan un importante riesgo zoonótico (Doyle y Buchanan, 2013).
El papel de Salmonella en la cadena alimentaria mundial, sus repercusiones actuales y proyectadas frente a la salud pública son motivo de preocupación sanitaria. Es por ello que el empleo de métodos sensibles, específicos, rápidos y replicables para la detección de dichos agentes en muestras clínicas y en alimentos es fundamental para su identificación, monitoreo, prevención y control. Dentro de dichos métodos, las técnicas moleculares como el PCR representan una herramienta diagnóstica de alta sensibilidad para la detección de patógenos (Abd El Tawwab et al., 2013).
Durante el proceso de adaptación, la Salmonella ha generado una variedad de mecanismos para colonizar, invadir, replicar y sobrevivir dentro del huésped, debido en parte al desarrollo de un sistema de secreción de proteínas asociadas con la invasión bacteriana, el cual depende de un componente esencial codificado por el gen invA, presente en todas las cepas virulentas de Salmonella. Es por ello que se estableció la presencia del gen invA como una diana genética de Salmonella en métodos de PCR (Espinal et al., 2006; Martínez, 2007).
La serotipificación es parte de la caracterización epidemiológica de los aislados, ya que faculta la determinación de la prevalencia de una serovariedad en distintas zonas geográficas, y es pieza importante en los estudios de brotes al poder determinar la fuente de infección y vías de transmisión (Herikstad et al., 2002). La serotipificación permite determinar la composición antigénica del organismo, para luego agruparlos en serovariedades, mediante el empleo del esquema de Kauffmann-White (Caffer et al., 2008). El objetivo del presente trabajo fue, por lo tanto, la caracterización de 46 aislados compatibles bioquímicamente con Salmonella spp (mediante bioquímicas convencionales) Para ello se realizó la caracterización genética determinando la presencia del gen invA, así como la caracterización serológica, objetivo de mayor significancia por su importancia epidemiológica.
MATERIALES Y MÉTODOS
Muestras
Se analizaron 46 aislados de Salmonella spp de origen aviar, los cuales fueron aislados y almacenados en el Laboratorio de Bacteriología de la Universidad Nacional Mayor de San Marcos, entre los años 2012 y 2017. El cepario constituye un registro de todas las muestras positivas por evaluación fenotípica y bioquímica a Salmonella spp, sin discriminar por patogenicidad. Las cepas se encuentran preservadas en caldo cerebro corazón (BHI) con glicerol al 20% en tubos de 2 ml a una temperatura de -20 ºC. Este registro está conformado por muestras provenientes de huevos comerciales (21/46), folículos ováricos (6/46), carcasa de pollos de engorde (3/46), y de vísceras como bazo (9/46) e hígado (7/ 46). El servicio de bacteriología de alimentos es parte del servicio de diagnóstico que ofrece el laboratorio, de allí la recepción de mues- tras de esta naturaleza.
Como cepa de referencia positiva se empleó a la Salmonella Typhimurium ATCC 14028 y como cepa de control negativo a Escherichia coli ATCC 8739.
Reactivación de Cepas
Los 46 aislados de Salmonella spp y las dos cepas de referencia fueron reactivados empleando un protocolo modificado de Sánchez y Corrales (2005). Los viales fueron descongelados para proceder con la recuperación de los microorganismos. Se inoculó 10 µl de las cepas en el medio TSB (tripticasa de soya) y mantenidos a 37 °C durante 18 horas. Luego fueron sembrados por agotamiento en medios selectivos: Agar MacConkey (MC) y en Agar Xilosa, Lisina, Desoxicolato (XLD) e incubados a 37 °C durante 24 horas.
La siembra en agar MacConkey permite detectar con rapidez bacterias gran negativas no fermentadoras de lactosa como Salmonella spp (las colonias sospechosas son incoloras). La siembra en agar XLD se hizo para poder observar el crecimiento característico de Salmonella: colonias circulares transparentes con tonos rojizos y centro negro debido a la liberación de sulfuro de hidrógeno (H2S).
Las colonias de Salmonella se inocularon finalmente en tubos de microcentrífuga de 2 ml conteniendo 1500 µl de TSB e incubados a 37 °C durante 3 h.
Análisis de Muestras
Para la extracción del ADN genómico se utilizó el kit comercial Thermo Scientific GeneJET Genomic DNA Purification (ThermoFisher Scientific), siguiendo las especificaciones recomendadas por el fabricante. El material genético extraído se colocó en tubos de microcentrífuga de 1.5 ml y almacenados a -20 ºC.
Para la caracterización genética se realizó el PCR con el ADN extraído. Se emplearon cebadores de244 pb que codifican secuencias específicas del gen invA (Bhatta et al., 2007) (Cuadro 1). El volumen final de muestra fue ajustado a 20 µl, el cual contenía 10 µl de mezcla HotStarTaq® Master Mix 2X (Qiagen Group, Alemania), 1 µl de cebador invA1, 1 µl de cebador invA2, 6 µl de agua libre de nucleasas (H2O up) y 2 µl del ADN previamente extraído. Para la reacción de amplificación se empleó un termociclador Biometra TOne (Analytik Jena, Alemania) bajo las siguientes condiciones: incubación inicial a 94 °C durante 1 min, 30 ciclos con una desnaturalización de 30 s a 94 °C, hibridación de 30 s a 60 °C, extensión de 2 min a 72 °C y una extensión final durante 10 min a 72 °C.
Los productos de la PCR fueron analizados mediante electroforesis en gel de agarosa al 2% más buffer TBE 0.5X durante 75 min a 100V. Los productos fueron analizados en el transiluminador SafeView de luz azul. Para la determinación del tamaño se emplearon marcadores de peso molecular de 100pb Opti-DNA Marker (Applied Biological Materials). Como reactivo fluorescente para la visualización instantánea de las bandas de ADN se usó el colorante Safe-Green TM (Aplied Biological Meterials).
Posteriormente se procedió a establecer las serovariedades mediante el empleo de kits serológicos comerciales compuestos de antisueros somáticos y flagelares (Denka Seiken, Japón), siguiendo las recomendaciones indicadas por Caffer et al. (2008). La serotipificación se llevó a cabo en el Laboratorio de Enteropatógenos del Instituto Nacional de Salud (INS) del Perú.
RESULTADOS Y DISCUSIÓN
El análisis molecular de los aislados de Salmonella spp permitió identificar una secuencia del gen invA en las 46 muestras procesadas (Figura 1). Este hallazgo fue consistente a lo descrito por múltiples estudios en muestras de alimentos de procedencia animal (Das et al., 2012) incluyendo los de ori- gen aviar (Borges et al., 2013; Fardsanei et al., 2017) y de muestras clínicas colectadas de animales y humanos (De Oliveira et al., 2003; Luigi et al., 2015). De manera similar, diversos autores (Rahn et al., 1992; Malorny et al., 2003; Espinal et al., 2006; Turki et al., 2014) reportaron frecuencias relativamente altas del gen invA en aislados de Salmonella spp (97.3-99.6%). Esta ligera diferencia en las frecuencias en referencia al presente estudio pudo deberse a una pérdida o deleción de la isla de patogenicidad SPI1, lo cual podría afectar la detección del gen debido a una alteración en el sitio de inserción en el cromosoma y/o remoción de los genes contenidos en la isla, probablemente por la transferencia horizontal de dichos bloques de genes de virulencia (Espinal et al., 2006).
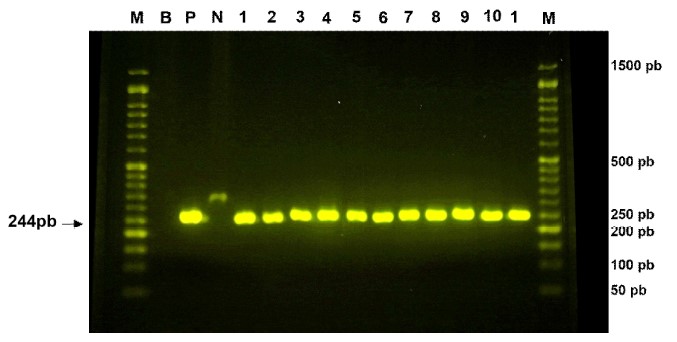
Figura 1 PCR para la detección del gen invA. Carriles: M: marcador de peso molecular de 100pb, B: Control blanco, P: Control positivo cepa Salmonella Typhimurium ATCC 14028, N: Control negativo cepa E. coli ATCC 8739, 1-11: Cepas en estudio presentan bandas de 244 pb
Es probable que la alta tasa (100%) de detección reportada en el presente estudio sea debido a que el gen invA representa a un gen de invasión altamente conservado entre los serotipos patógenos de Salmonella enterica (De Oliveira et al., 2003; Das et al., 2012).
A nivel serológico, en el presente estudio se identificaron los serotipos Infantis (16/46, 34.8%), Pullorum (16/46, 34.8%), Gallinarum (7/46, 15.2%), Enteriditis (4/46, 8.7%), y Typhimurium (3/46, 6.5%). De los cuales tres de ellos representan un riesgo en salud pública (23/46, 50%) (S. Infantis, S. Enteritidis y S. Typhimurium), debido a que son agentes con potencial zoonótico causales de salmonelosis no tifoidea en el hombre. Los serotipos restantes (23/46, 50%) S. Pullorum y S. Gallinarum son microorganismos responsables de ocasionar la pullorosis y tifosis aviar, respectivamente, en diversas especies de aves domésticas (Terzolo, 2009).
S. Enteritidis y S. Typhimurium son los serovares predominantes en infecciones por Salmonella no tifoidea en humanos; sin embargo, en los últimos años S. Infantis se ha convertido en un serotipo emergente entre los causantes de salmonelosis no tifoidea, hallazgo que podría estar asociado a la elevada frecuencia de este serovar en aves de corral. Además, su resistencia a múltiples fármacos parece ser propagada exitosamente entre productos de origen aviar y humanos a través de la cadena alimentaria (EFSA- ECDC, 2015, 2018; Pate et al., 2019). Se ha demostrado que las cepas emergentes de S. Infantis se caracterizan por mutaciones cromosómicas adaptativas y un megaplásmido que le confiere resistencia a múltiples antimicrobianos, metales pesados y desinfectantes. Debido a esto tiene una ventaja significativa, tanto en el medio ambiente como en el hospedero, haciendo que su difusión sea más eficiente y rápida (Aviv et al., 2014).
En Perú, se han reportado datos que demuestran que S. Infantis es el serotipo más frecuente en granjas avícolas (91.4%) (Valderrama et al., 2014), mientras que en 2011 se demostró que era el tercer serotipo más frecuente en aislados de muestras clínicas de humanos y de alimentos (Zamudio et al., 2011). Esto podría estar relacionado con los datos actuales que indican que el 65% (193/297) de muestras clínicas de humanos remitidas de diversas regiones del Perú hayan sido confirmadas como Salmonella Infantis (Quino et al., 2019). Estos hallazgos sugieren que es el serovar predominante desde el punto de vista epidemiológico en el país.
El presente trabajo no es un estudio probabilístico y se tuvo, además, la limitante del pequeño número de muestras evaluadas, y que no representan de ese modo a la región Lima. No obstante, el hallazgo de 50% de muestras positivas a serotipos de Salmonella con carácter zoonótico sigue siendo una advertencia.
CONCLUSIONES
Los serotipos hallados en el presente estudio fueron S. Infantis, S. Pullorum, S. Gallinarum, S. Enteritidis y S. Typhimurium en aislados de Salmonella de origen aviar del cepario de la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos.
Todos los aislados (n=46) identificados como Salmonella spp mediante bioquímica convencional codificaron el gen invA.












 uBio
uBio 



