INTRODUCCIÓN
Las enfermedades transmitidas por alimentos (ETA) son principalmente de origen animal, en donde las enterobacterias deben presentar un recuento bacteriano suficiente para producir la infección (OMS, 2021). Sin embargo, el impacto en la salud pública no solamente radica en el desarrollo de las enfermedades, sino también en la manifestación de resistencia a los antibióticos, entre ellos los β-lactámicos (Ochoa et al., 2009; Granda et al., 2019). Diversas investigaciones indican que la transmisión de bacterias resistentes a los antibióticos se puede realizar a través del consumo de alimentos como carne mal cocida, contacto con heces de animales o por contacto directo con personas portadoras de la bacteria (Marshall y Levy, 2011; Shrestha et al., 2017).
La resistencia antibiótica de las entero-bacterias, principalmente de Escherichia coli, a tetraciclinas o cotrimoxazol, ha sido reportada en humanos sanos y enfermos (Kola et al., 2012). Estos antibióticos junto con los β- lactámicos suelen utilizarse regularmente como promotores de crecimiento y para tratamientos en granjas avícolas (Kola et al., 2012). Es importante enfatizar que la gran capacidad de diseminación de genes de resistencia mediante plásmidos ha permitido resistencia cruzada frente a distintos antibióticos (Seral García et al., 2010). En países europeos se demostró que el 19% de Escherichia coli productora de BLEE aislada de humanos presentó genes BLEE, cuyos plásmidos fueron genéticamente indistinguibles de aislamientos de origen avícola (Leverstein-van Hall et al., 2011).
El consumo per capita de carne de pollo en el Perú en marzo de 2019 fue de 4.2 kg/hab/mes (MINAG, 2019). Esta demanda exige que los sistemas productivos avícolas sean más eficientes; sin embargo, la producción intensiva ha conllevado a utilizar antibióticos frente a distintos desafíos en aves, principalmente broilers y ponedoras, lo cual ha generado resistencia antibiótica (De Briyne et al., 2014). Esto indica, además, que la contaminación de enterobacterias productoras de BLEE también podría ser de origen animal (Ruiz-Roldán et al., 2018), pues las heces son el principal reservorio de transmisión de genes BLEE (Shrestha et al., 2017).
En ese contexto, el objetivo del estudio fue evidenciar la presencia de enterobacterias productoras de β-lactamasas de espectro extendido en carne de pollo expendidas en mercados de abasto del distrito de Santiago de Surco en Lima, Perú.
MATERIALES Y MÉTODOS
El estudio observacional descriptivo se desarrolló en mercados de abasto del distrito de Santiago de Surco de Lima Metropolitana entre marzo y abril de 2019. El procesamiento se llevó a cabo en el Laboratorio de Salud Pública y Salud Ambiental (procedimiento microbiológico y bioquímico) y en Laboratorio de Patología Aviar (extracción de ADN y diagnóstico molecular) de la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos (UNMSM), Lima, Perú.
Considerando los hallazgos detectados en investigaciones (Ruiz-Roldán et al., 2018; Cortez Sandoval y Shiva, 2019) de enterobacterias realizadas en distritos de Lima y que el distrito de Santiago de Surco tiene un control sanitario riguroso a nivel de mercados de abasto por la gestión municipal que actualmente prioriza la vigilancia sanitaria y la salud pública (A. Mansilla, Lima, comunicación personal), se consideró relevante detectar la presencia de enterobacterias con genes BLEE en mercados de abasto que tengan un control sanitario riguroso como lo presenta este distrito. El tamaño de muestra fue definido mediante la fórmula de detección de enfermedad o prevalencia limite (Thrusfield, 1990), con un nivel de confianza del 95% y prevalencia límite de 10.9% (Ojer-Usoz et al., 2013) para la detección de los genes menos frecuentes (TEM y SHV) de enterobacterias productoras de β-lactamasas de espectro extendido (BLEE) en muestras positivas a enterobacterias obtenidas de muestras de carne de pollo. El tamaño de muestra calculado fue de 26; no obstante, se obtuvieron muestras adicionales a fin de garantizar el número mínimo de muestras ante la eventualidad de pérdidas.
Se estableció una base de datos con la lista de mercados de abasto brindada por la Municipalidad de Santiago de Surco (Exp. 1155562019). Se excluyeron los mercados que no contaban con puestos de carne de pollo, quedando disponibles 25 mercados. El muestreo se realizó de manera aleatoria, mediante un sistema computarizado (Tyrer y Heyman, 2016). De estos, nueve (los más grandes) fueron seleccionados para realizar el muestreo en dos puestos, mientras que en los restantes se muestreó un solo puesto por mercado, registrándose un total de 34 muestras. La colección de muestras fue realizada con intervalo de 2 días.
Los operarios en la mayoría de los puestos de los mercados contaban con mandil de plástico y guantes. Las mesas donde se cortaba la carne eran de loseta y en algunos casos se usaban tablas de madera con desperfectos a manera de grietas. Las bandejas donde se almacenaban los cortes para la venta eran de fierro enlozado y, en algunos casos, contaban con bandejas con materiales adecuados desde el punto de vista sanitario. Sin embargo, solo en algunos recintos se observó que se mantenía la cadena de frío. Las herramientas para trozar la carne y utensilios eran adecuadas. Algunos mercados se ubicaban en espacios abiertos no techados. También se observó presencia de plagas (dípteros) en algunos establecimientos.
Las piezas de pollo (ala y pierna) se encontraban trozadas al momento de la compra (toma de muestra). Estas fueron colectadas siguiendo las recomendaciones de Santos et al. (2020) y Weese et al. (2010). Las muestras fueron adquiridas mediante una transacción económica cancelando por ello el costo correspondiente al peso de las piezas adquiridas. Las muestras fueron empacadas directamente en bolsas de plástico por el vendedor, de manera que se asegure el canal tradicional de venta al por menor en el puesto de venta. A su vez, los productos adquiridos se depositaron en bolsas estériles y trasladadas al laboratorio bajo condiciones de refrigeración (5 °C) para el procedimiento microbiológico y bioquímico.
El procedimiento microbiológico utilizado fue un protocolo para el aislamiento de enterobacterias descrito por la Comisión Internacional de Especificaciones para Alimentos (ICMSF, 1978), con algunas modificaciones. Se realizó un homogenizado de los cortes en Stomacher® 400 Circulator a 270 rpm por 3 min, se tomó 10 g de carne homogenizada y se colocó en 90 ml de agua destilada en bolsas estériles para una nueva homogenización con el mismo equipo por 2 min. De la dilución, se sembró 0.1 ml en agar EMB (Agar Eosina y Azul de Metileno) para obtener las colonias características y en TSA (Agar Tripticasa Soya) para las pruebas bioquímicas. En ambos casos, los cultivos fueron incubados a 37 ± 2° C durante 24 ± 2 h. Se seleccionó una colonia de enterobacterias al azar por cada cultivo correspondiente a cada homogenizado. Se realizó la tinción Gram, y la prueba de oxidasa a partir de un cultivo bacteriano en TSA. Ambas pruebas se realizaron para confirmar la detección de bacilos Gram y oxidasa negativos y asegurar el aislamiento de enterobacterias. Luego, se utilizó el kit EnteroPluri-Test® (Liofilchem) para la caracterización bioquímica de enterobacterias, siguiendo las instrucciones del fabricante.
La susceptibilidad antimicrobiana se realizó bajo el método de Kirby-Bauer en agar Müller Hinton. La inoculación de la bacteria se efectuó habiendo llevado la turbidez a 0.5 en la escala de Mc Farland (CLSI, 2015). Debido a la característica principal de las BLEE (sensibilidad disminuida frente a cefalosporinas de amplio espectro y aztreonam), los discos antibióticos utilizados fueron los β-lactámicos: cefotaxima (CTX) 30 µg, ceftazidima (CAZ) 30 µg, ceftriaxona (CRO) 30 µg, aztreonam (ATM) 30 µg y cefpodoxima (CPD) 10 µg, según el protocolo propuesto por autores de referencia y la CLSI ( Seral García et al., 2010; CLSI, 2015; Perozo Mena et al., 2017). Las placas de Müller Hinton cultivadas fueron incubadas por 16 ± 2 h a 35 ± 2 ºC, con el fin de detectar mediante este método de cribado a enterobacterias sospechosas de producir BLEE (CLSI, 2015; Perozo Mena et al., 2017).
Las cepas que resultaron ser resistentes al método de cribado fueron seleccionadas para confirmar el fenotipo de resistencia mediante el método de Jarlier, también llamado método del doble disco (método confirmatorio) (Ojer-Usoz et al., 2013; Perozo Mena et al., 2017; EUCAST, 2018). El método de doble disco fue realizado en agar Müller Hinton, bajo las mismas condiciones del método de cribado utilizando los mismos antibióticos con adición de un disco de amoxicilina/ácido clavulánico (AMC) 30 µg, debido a la sensibilidad disminuida de las BLEE también frente al ácido clavulánico (Seral García et al., 2010; Perozo Mena et al., 2017; EUCAST, 2018). El disco de amoxicilina/ácido clavulánico (AMC) 30 µg se colocó en el centro del antibiograma y los demás discos a 30 mm de distancia (EUCAST, 2018). Los resultados positivos fueron mediados por la manifestación de un efecto de sinergia (también llamado efecto huevo, de cola de pez o de corcho de champagne) entre los discos de amoxicilina/ ácido clavulánico y los discos circundantes (Ojer-Usoz, 2013; Perozo Mena et al., 2017; EUCAST, 2018). Las enterobacterias positivas al método del doble disco fueron seleccionadas para el análisis molecular mediante PCR en punto final para la detección de los genes de resistencia codificantes para el fenotipo de producción de β-lactamasas de espectro extendido (BLEE) (Seral García et al., 2010; Ojer-Usoz, et al., 2013; Perozo Mena et al., 2017).
Para la identificación de los genes BLEE, fueron realizadas reacciones de PCR en punto final. Para eso, se realizó la extracción del ADN bacteriano a partir de colonias previamente aisladas utilizando el kit de extracción GeneJet Genomic DNA purification kit (Thermo Scientific™) según las instrucciones del fabricante.
Para la detección de los genes CTX- M-1, CTX-M-2 y CTX-M-9 se realizó un PCR simple en punto final para cada gen (Woodford et al., 2006) y para la detección de genes TEM y SHV se realizó el PCR dúplex, según protocolo descrito por Colom et al. (2003). Las secuencias de los cebadores se detallan en la Cuadro 1. Los ensayos de PCR se realizaron con un volumen final de reacción de 25 µl, con la siguiente composición: 12.5 µl de DreamTaq Green MasterMix, 2 µl de ADN templado y 0.25 µM de cebadores específicos de cada reacción. Cepas aisladas de bacteriemias de neonatos humanos fueron usadas como controles positivos y agua PCR fue usada como control negativo en todas las reacciones de PCR.
Las condiciones de amplificación fueron una modificación de las metodologías descritas por Colom et al. (2003) y Woodford et al. (2006). Las reacciones para detección de CTX-M-1, CTX-M-2 y CTX-M-9 consistieron en una desnaturalización inicial a 95 ºC por 3 min, seguido de 30 ciclos de desnaturalización de ADN a 95 ºC por 30 s, hibridación a 52 ºC por 40 s, extensión a 72 ºC por 1 min, y una elongación final a 72 ºC por 10 min. Para el PCR para la detección de los genes TEM y SHV, las condiciones de amplificación fueron desnaturalización inicial a 95 ºC por 3 min, seguido de 32 ciclos de desnaturalización de ADN a 95 ºC por 30 s, hibridación a 52 ºC por 30 s, extensión a 72 ºC por 1 min, y una elongación final a 72 ºC por 10 min.
Los productos del PCR fueron analizados mediante electroforesis en 2% de gel de agarosa (Ultrapure Agarosa™) con bromuro de etidio (0,5 µg/ml) y 1X de buffer TBE™. El gel fue fotografiado bajo luz ultravioleta usando un transiluminador (Clear View, Cleaver Scientific).
RESULTADOS
Todas las muestras fueron positivas a la detección de enterobacterias por cultivo bacteriano y superaron el límite de criterios microbiológicos de Escherichia coli para carnes crudas picadas y molidas, según la NTS-071 de la R.M. 591-2008/MINSA/DIGESA-V-01. Asimismo, en el Cuadro 2 se puede observar que hubo presencia de Salmonella spp en las muestras de carne. Se identificaron 34 cepas de enterobacterias, de las cuales 20/34 fueron sospechosas de producir enzimas BLEE bajo el método de Kirby-Bauer, en tanto que 12/34 fueron cepas confirmadas bajo el método de doble disco de producir BLEE. Estas últimas cepas fueron seleccionadas para los ensayos de PCR en punto final y correspondieron a una cepa de Salmonella enterica serovar choleraesuis, ocho cepas de Escherichia coli, una cepa de Serratia odorifera y dos cepas de Klebsiella ozaenae.
Cuadro 2 Perfil de resistencia antimicrobiana y multidrogoresistencia (MDR) de enterobacterias aisladas en carne de pollo vendida en expendio (expresado en milímetros de halo de inhibición)
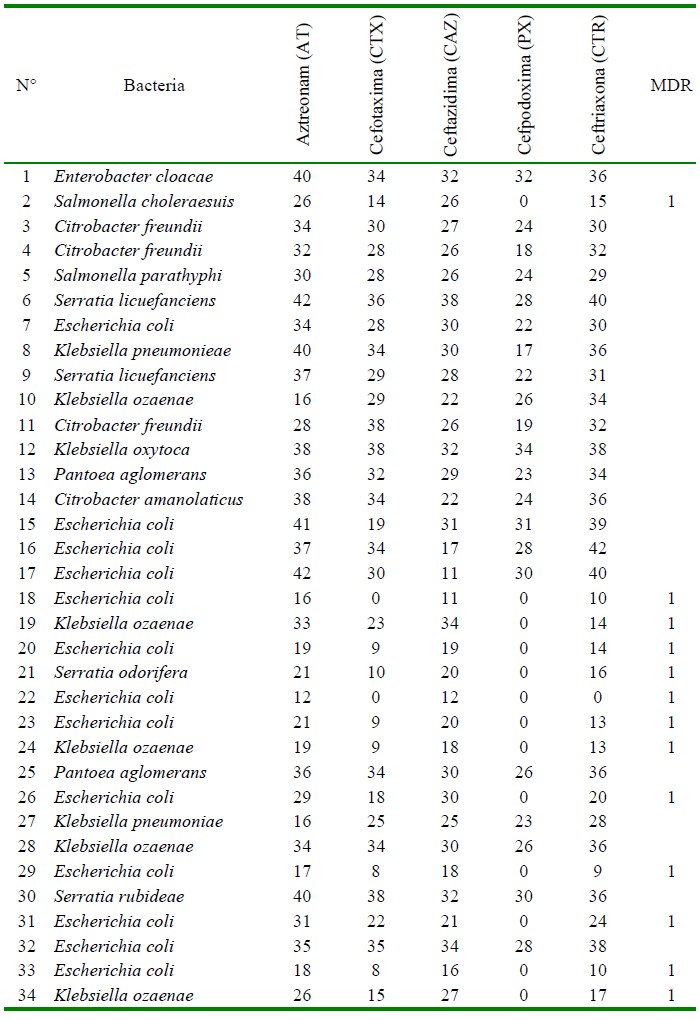
Las cepas resistentes a múltiples antibióticos (MDR) se reportaron en función a la sensibilidad a los antibióticos probados en cada cepa por el método Kirby-Bauer, donde 13/34 fueron positivas (Cuadro 2), siendo ocho cepas de Escherichia coli, tres de Klebsiella ozaenae, una de Serratia odorifera y una de Salmonella enterica serovar choleraesuis. Por otro lado, en 12 cepas de las 34 enterobacterias aisladas se detectaron genes BLEE por el método molecular, siendo CTX- M-1 el gen con mayor frecuencia (12/12), seguido de TEM (10/12) y CTX-M-9 (2/12) (Figuras 1, 2 y(3). No se detectó la presencia de genes CTX-M-2 ni SHV.
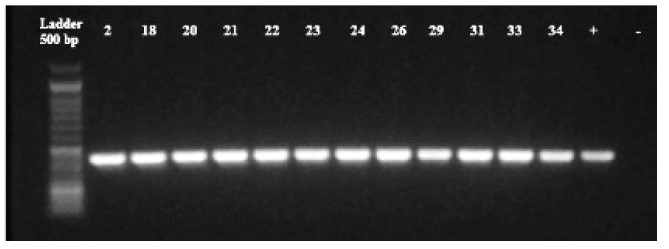
Figura 1 Detección de genes CTX-M-1 (12/12) (415 bp). Control positivo (+), control negativo (-). Muestras: 2 (Salmonella choleraesuis), 18 (Escherichia coli), 20 (Escherichia coli), 21 (Serratia odorifera), 22 (Escherichia coli), 23 (Escherichia coli), 24 (Klebsiella ozaenae), 26 (Escherichia coli), 29 (Escherichia coli), 31 (Escherichia coli), 33 (Escherichia coli), 34 (Klebsiella ozaenae). Todas las muestras resultaron positivas

Figura 2 Detección de genes CTX-M-9 (2/12) (205 bp). Control positivo (+), control negativo (-). Muestras: 2 (Salmonella choleraesuis), 18 (Escherichia coli), 20 (Escherichia coli), 21 (Serratia odorifera), 22 (Escherichia coli), 23 (Escherichia coli), 24 (Klebsiella ozaenae), 26 (Escherichia coli), 29 (Escherichia coli), 31 (Escherichia coli), 33 (Escherichia coli), 34 (Klebsiella ozaenae). Las muestras positivas fueron las cepas 2 (Salmonella choleraesuis) y 34 (Klebsiella ozaenae)
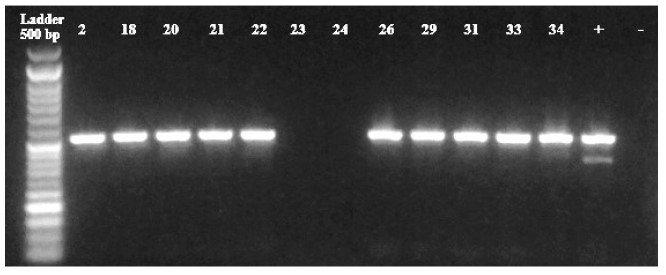
Figura 3 Detección de TEM (516 bp) y SHV (392 bp). Control positivo (+), control negativo (-). Muestras: 2 (Salmonella choleraesuis), 18 (Escherichia coli), 20 (Escherichia coli), 21 (Serratia odorifera), 22 (Escherichia coli), 23 (Escherichia coli), 24 (Klebsiella ozaenae), 26 (Escherichia coli), 29 (Escherichia coli), 31 (Escherichia coli), 33 (Escherichia coli), 34 (Klebsiella ozaenae). Todas las muestras resultaron positivas para el gen TEM con excepción de las cepas 23 (Escherichia coli) y 24 (Klebsiella ozaenae) (83.3%). Ninguna muestra fue positiva para el gen SHV
DISCUSIÓN
Todas las muestras de carne de pollo fueron positivas para la detección de enterobacterias. La NTS N.° 071 aprobada por R.M. 591-2008/MINSA/DIGESA-V-01 no indica un criterio microbiológico específico para enterobacterias en carne de pollo. Sin embargo, si bien el objetivo del estudio no es establecer un recuento bacteriano, se comparó el recuento de UFC/g de enterobacterias con el límite de UFC/g del punto X.6 Carnes crudas picadas y molidas para Escherichia coli (50 - 5x102). El recuento de enterobacterias resultó estar por encima del límite microbiológico, lo cual podría indicar contaminación fecal originaria de algún punto de la cadena productiva por ausencia de buenas prácticas de manipulación durante el eviscerado, falta de refrigeración del producto durante la venta y malas prácticas de higiene del manipulador (Rouger et al., 2017). Así mismo, los cortes que se realizan en la carne favorecen la salida de líquido intracelular (elevada actividad de agua) y compuestos proteicos, los cuales actúan como medio de cultivo para la proliferación de bacterias que se encuentran en la carne (Saenz-García et al., 2020; Rosas, 2007). Todas las cepas de Escherichia coli seleccionadas para la detección molecular de genes BLEE fueron positivas a la detección de al menos un gen. Si bien la prevalencia de aislamiento para Escherichia coli en Lima fue de 95.3% (Ruiz-Roldán et al., 2018), estudios en ciudades europeas como Navarra, España, indican prevalencias del 59.0% (Vitas et al., 2018) y en ciudades asiáticas como Sylhet, Bangladesh de 63.5% (Rahman et al., 2020). Asimismo, Ruiz-Roldán et al. (2018) detectaron 59.4% (19/32) de Escherichia coli productora de BLEE en aislamientos en carne de pollo en mercados de Lima metropolitana, en tanto que en el presente estudio se encontró que todas las cepas fueron productoras de BLEE.
Estudios como el de Ojer-Usoz et al. (2013) y el de Ruiz-Roldán et al. (2018) han podido detectar prevalencias altas de E. coli y otras enterobacterias productoras de BLEE en carne de pollo por encima de otras carnes. Si bien no se puede descartar la contaminación cruzada durante la manipulación en mataderos o puestos de mercados, la alta prevalencia de BLEE en enterobacterias también podría deberse al uso indiscriminado de antimicrobianos en granjas avícolas, una problemática que a través del tiempo se ha agudizado debido a la explotación intensiva de animales de producción (Smet et al., 2010; Aarestrup, 2015). Esta hipótesis no debe quedar aislada si se considera el consumo per cápita de carne de pollo (4.2 kg/hab/mes) de la población peruana (MINAG, 2019) y la crianza intensiva de pollos.
Si bien el recuento de enterobacterias resultó elevado, la relativa baja frecuencia de E. coli (38%, 13/34) podría explicarse por la rigurosidad de las municipalidades en cumplir las Buenas Prácticas de Manipulación (BPM), Buenas Practicas de Higiene (BPH) e inspecciones sanitarias en mercados de distritos como Santiago de Surco, tal como indica la R.M. 282-2003; característica que puede verse afectada en otros distritos como los citados en el estudio de Ruiz-Roldán et al. (2018). Así mismo, los mataderos también deben estar regulados por la autoridad competente (DS Nº 029-2007-AG/SENASA- PERÚ), debido a que también pueden ser fuente de diseminación de enterobacterias productoras de BLEE.
Un hallazgo resaltante del estudio fue la detección de Salmonella enterica serovar choleraesuis y Salmonella enterica serovar paratyphi, ambas patógenas; considerando que cualquier especie de Salmonella spp es un contaminante en la carne de acuerdo con la RM. N° 591-2008-/MINSA/DIGESA-V-01 y por tanto debe de estar ausente. La OMS (2018) atribuye al género Salmonella spp la capacidad de generar enfermedad no solamente por serovares como typhi o paratyphi, sino también por serovariedades clasificadas como específicas para animales, debido a que se ha reportado que serovares como enteritidis, typhimurium y choleraesuis, entre otros, han generado enfermedades en humanos, desde leves a graves sintomatologías gastrointestinales, fiebres e incluso sepsis (Chiu et al., 2004; OMS, 2018).
Los genes de mayor frecuencia fueron CTX-M-1 (12/12; 100%) y TEM (10/12;83.3%). En esta línea, Kola et al. (2012) hallaron 41.2% (77/187) de muestras de carne de pollo en Alemania con el gen CTX-M-1 y 8.6% (16/187) para el gen TEM-52. En el Perú no se reporta la presencia de BLEE en heces de aves de corral; sin embargo, se reportan aislamientos de E. coli provenientes de órganos y vísceras de ave (Gordillo et al., 2019), en donde el gen de mayor prevalencia fue el CTX-M-1 (90.3%) seguido del gen SHV (8.1%). Por otro lado, el gen CTX-M-2 ha sido descrito como un genotipo de campo avícola (Leverstein-van Hall et al., 2011), lo que sugiere que la contaminación de la carne puede darse por heces de animales de explotaciones avícolas.
El estudio de Ojer-Usoz et al. (2013) realizado en España, indica que existe una mayor prevalencia (84.0%) de enterobacterias productoras de BLEE en carne de pollo sobre otras carnes como la de cerdo (55.0%) y res (59.0%). Asimismo, se indicó que los genes de mayor prevalencia detectados en la carne fueron los tipos CTX-M (Ojer-Usoz et al., 2013). Información que fue contrastada con reportes que indican la predominancia de CTX-M en aislamientos de origen animal y aislamientos clínicos (Smet et al., 2010). Esto indica la posibilidad de que aquellas infecciones en humanos por enterobacterias productoras de BLEE podrían tener un origen alimentario a través de la carne de pollo, pues se ha demostrado que las bacterias resistentes a los antibióticos pueden trasmitirse al hombre a través del consumo de carne mal cocida (Hoffmann et al., 2017; Langsrud et al., 2020). Sin embargo, en el Perú hacen falta mayores estudios a nivel epidemiológico para poder detectar algún tipo de correlación o asociación concreta.
Un estudio reciente en el Perú indica que el 92% de cepas Salmonella enterica serovar infantis aisladas de muestras de heces de infantes atendidos en hospitales de Lima fueron cepas productoras de BLEE (Granda et al., 2019). Asimismo, Zamudio et al. (2011) reportaron que Salmonella enterica serovar infantis es el tercer serovar más frecuentemente encontrado en huevos y carne de ave. Si bien en el presente estudio no se detectó este serovar, Granda et al. (2019) indicaron que la mayor prevalencia de genes BLEE detectados en los hospitales fue para el gen CTX-M-65 perteneciente al grupo CTX-M-9.












 uBio
uBio