INTRODUCCIÓN
La pandemia causada por el coronavirus COVID-19 ha afectado a más de 500 000 000 de personas en prácticamente todos los países y territorios del mundo (CSSE, 2022). El agente causal es el Coronavirus tipo 2 causante del Síndrome Respiratorio Agudo Severo (SARS-CoV-2), el cual pertenece a la familia Coronaviridae, subfamilia Orthocoronavirinae, género Betacoronavirus (âCoV) y subgénero Sarbecovirus (Elaswad et al., 2020). Su genoma, formado por un ARN monocatenario de unos 30 000 nucleótidos, codifica cuatro proteínas estructurales: espícula (proteína S), envoltura (E), membrana (M), nucleocápside (N) y 16 proteínas no estructurales de carácter regulador (Brooke y Prischi, 2020).
Se dispone de evidencia circunstancial que respalda dos posibles rutas de transmisión del SARS-CoV-2 desde reservorios naturales hacia el ser humano. La primera vía consiste en una transmisión zoonótica directa, evidenciada por la alta identidad genética entre el SARS-CoV-2 y dos coronavirus tipo SARS aislados de murciélagos: 1. El coronavirus RaTG13 (Bat-CoV-RaTG13), con el cual demostró 96.2% de identidad de secuencia (Zhou et al., 2020); y 2. Un coronavirus denominado RmYN02, con el que comparte una identidad de secuencia del 93.3% (Zhou et al., 2020). La segunda y más probable vía consiste en la transmisión del SARS-CoV-2 desde un animal con alta carga viral que funcionó como reservorio natural, a un hospedero intermedio, generando un efecto de spillover que finalmente permitió la transmisión del virus al ser humano (OMS, 2021a).
Para tener la habilidad de infectar diferentes especies, el virus debe someterse a diversas mutaciones y recombinaciones en el dominio de unión al receptor (RBD) de la proteína S que le permitan tener una afinidad mucho más alta por su receptor, la enzima convertidora de angiotensina 2 (ECA2) del hospedero (Andersen et al., 2020). La afinidad por la ECA2 es fundamental para el ingreso del SARS-CoV-2 a la célula, ya que el virus no tiene la capacidad de infectar células que no expresan la ECA2, mientras que infecta con gran facilidad células que poseen una sobreexpresión de esta (Zhou et al., 2020), sugiriendo que la efectividad de la unión entre la proteína S y la ECA2 refleja la susceptibilidad del hospedero al SARS-CoV-2 (Ma y Gong, 2021). En particular, el perro y el gato poseen la proteína ECA2 más conservada respecto al ser humano y expresan notablemente el gen ECA2 en varios tejidos corporales, confirmando la gran posibilidad de estos animales para funcionar como hospederos del SARS-CoV-2 (Sun et al., 2021).
Considerando que el brote de SARS-CoV-2 puede ser potencializado por la transmisión zoonótica del virus desde animales, incluso los de compañía, hacia el ser humano (Drózdz et al., 2021), es recomendable realizar vigilancia activa de los animales de compañía expuestos a personas COVID-19 positivas con el fin de entender el rol de los caninos y felinos domésticos como reservorios del virus (Hamer et al., 2021). La vigilancia epidemiológica del SARS-CoV-2 en animales se inició luego del primer reporte a la Organización Mundial de Sanidad Animal (OIE) de un canino doméstico infectado naturalmente con el virus (Sit et al., 2020). Desde entonces, se han reportado al Sistema Mundial de Información Zoosanitaria (OIE-WAHIS) más de 500 animales infectados (OIE-WAHIS, 2020). Paralelamente, los esfuerzos actuales de secuenciación genómica masiva del SARS-CoV-2 han permitido que la vigilancia epidemiológica del virus se realice casi en tiempo real, lo cual se ve evidenciado en más de tres millones de secuencias, compartidas en su totalidad en la plataforma GISAID (https://www.gisaid.org/) (Munnink et al., 2021).
Aunque se conoce desde hace décadas que los coronavirus circulan en varias especies animales, no existe suficiente información sobre la epidemiología animal, la existencia de posibles hospederos intermedios y las rutas actuales o potenciales de transmisión del SARS-CoV-2 desde animales hacia el ser humano (Ferri y Yong, 2020). Adicionalmente, en lo que respecta a caninos y felinos domésticos, no existe una revisión sistemática donde se realice vigilancia epidemiológica de las secuencias genéticas del SARS-CoV-2 aisladas de dichas especies alrededor del mundo. El presente estudio tuvo como objetivo realizar una revisión sistemática de la epidemiología del SARS- CoV-2 en caninos y felinos domésticos, así como el análisis de las secuencias genéticas del virus aisladas de perros y gatos reporta- das en la plataforma GISAID, con el fin de identificar las dinámicas de transmisión del virus entre animales de compañía y el ser humano, así como para brindar un punto de partida para una correcta vigilancia epidemiológica.
MATERIALES Y MÉTODOS
Fuentes de Información y Estrategia de Búsqueda
La búsqueda de información se basó en seis plataformas científicas, NCBI (PubMed: https://pubmed.ncbi.nlm.nih.gov/), BioRxiv (https://www.biorxiv.org/), Elsevier Group (ScienceDirect: http://www.sciencedirect.com/), MDPI (https://www.mdpi.com/), Scholar Google (Scholar Google: https://scholar.google.com/) y ResearchGate (https:// www.researchgate.net/). Los términos de búsqueda incluyeron: SARS-CoV-2, COVID- 19, perros, gatos, epidemiología, transmisión animal, mascotas, animales de compañía, reservorios animales, y zoonosis (empleando términos en inglés). La revisión sistemática siguió los protocolos PRISMA para este tipo de estudios (Page et al., 2021).
Criterios de Elegibilidad
Se usaron los siguientes criterios de elegibilidad:
Artículos publicados donde se reportan solo infecciones naturales con el SARS-CoV-2 en perros y gatos, estudios de seroprevalencia e interacciones y saltos interfaces de SARS-CoV-2 entre animales de compañía y el ser humano.
Reportes realizados al OIE-WAHIS de infecciones naturales con el SARS-CoV-2 en caninos y felinos domésticos (https://www.oie.int/es/que-ofrecemos/emergencia-y-resiliencia/covid-19/#ui-id-3).
Secuenciaciones genéticas del SARS-CoV-2 aisladas de caninos y felinos domésticos reportadas en la base de datos EpiCoV™ de la plataforma GISAID.
Los documentos y reportes debieron ser publicados desde el 1 de enero de 2020 hasta el 10 de octubre de 2021. No se estableció límite de idioma del artículo, pero se excluye- ron los documentos que no se relacionaban con los objetivos del estudio. Los resultados de búsqueda se evaluaron de forma independiente por dos investigadores.
Selección de Estudios
Los investigadores formaron dos grupos de trabajo, donde se leyeron los títulos y resúmenes de los artículos de las bases de datos que fueron consultadas de acuerdo con las palabras clave. Luego de comparar la información entre los grupos de trabajo, los artículos e informes se extrajeron y fueron leídos y analizados en su totalidad, examinándose de acuerdo con los criterios de inclusión y exclusión. Se eliminaron los registros duplicados (Figura 1).
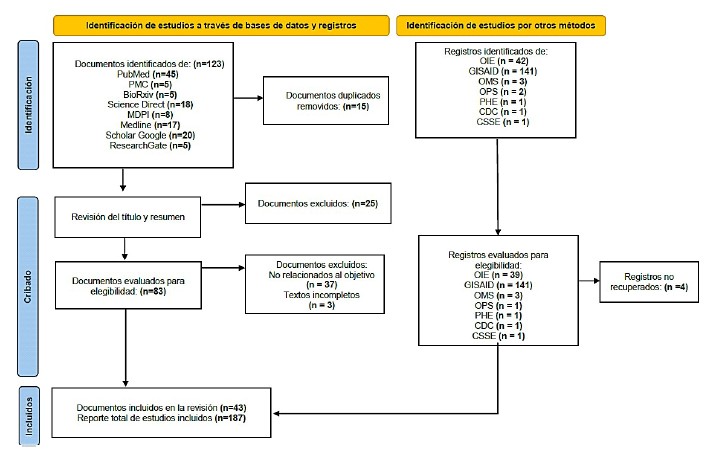
Figura 1 Características y selección de estudios basados en el protocolo PRISMA 2020 para revisiones sistemáticas. OIE: Organización Mundial de Sanidad Animal; GISAID: Global Initiative on Sharing All Influenza Data; OMS: Organización Mundial de la Salud; OPS: Organización Panamericana de la Salud; PHE: Public Health England; CDC: Centers for Disease Control and Prevention; CSSE: Center for Systems Science and Engineering
Análisis Filogenético
Los genomas del SARS-CoV-2 aislados de perros y gatos, secuenciados y depositados en la base de datos EpiCoV™ de la plataforma GISAID fueron analizados a través de la herramienta Nextclade v1.7.1 (https://clades.nextstrain.org/) para la generación de los árboles filogenéticos, donde las relaciones filogenéticas de los clados del SARS- CoV-2 se analizaron según lo definido por Nextstrain Project (https://nextstrain.org/ncov/gisaid/global). Para este estudio, la filogenia tuvo sus raíces en la relación con la estructura genómica de la referencia Wuhan- Hu-1/2019 para SARS-CoV-2. La resolución temporal que se asumió fue de una tasa de sustitución de nucleótidos de 8×10^-4 subs por sitio por año y la aptitud mutacional se calculó utilizando los resultados de Obermeyer et al. (2021).
Los sistemas de nomenclatura para denominar la diversidad genética del SARS-CoV-2 utilizados en este estudio fueron «Phylogenetic Assignment of Named Global Outbreak LINeages (PANGOLIN)» para nombrar los linajes (https://cov-lineages.org/) y «Nextstrain» para la nomenclatura de los clados (https://nextstrain.org/blog/2021-01-06-updated-SARS-CoV-2-clade-naming), siguiendo la metodología propuesta por Alm et al. (2020); donde siempre se asoció cada clado o linaje con las variantes de preocupación en salud pública denominadas por la OMS (https://www.who.int/es/activities/tracking-SARS-CoV-2-variants).
RESULTADOS Y DISCUSIÓN
Se recuperaron 123 artículos por medio de la estrategia de búsqueda. Después de la selección por resumen y título, se seleccionaron 83 artículos para la evaluación de texto completo. De estos, 37 fueron excluidos por no tener relación alguna con los objetivos del estudio y tres fueron excluidos por encontrarse incompletos, quedando finalmente 43 artículos. Por otro lado, se recuperaron 187 reportes de las plataformas incluidas en el estudio, quedando 141 reportes a la plata- forma GISAID de aislamientos genéticos del SARS-CoV-2 de perros y gatos, 39 informes del portal OIE-WAHIS de perros y gatos domésticos positivos para SARS-CoV-2 y 7 reportes de organizaciones de salud pública del mundo (OMS [Organización Mundial de la Salud], OPS [Organización Panamericana de la Salud], PHE [Public Health England], CDC [Centers for Disease Control and Prevention] y CSSE [Center for Systems Science and Engineering]) para analizar los resultados de la revisión sistemática y el análisis filogenético (Figura 1).
Interacciones y Saltos Interfaces del SARS-CoV-2 entre Humanos y Animales de Compañía
El perro (Canis lupus familiaris) y el gato (Felis catus) son las principales especies usadas como animales de compañía y tienen estrecho contacto con el humano (Michelitsch et al., 2021). Al realizar un análisis filogenético de la longitud total de la proteína ECA2 de estas especies, se encontró que la ECA2 del gato doméstico cuenta con una identidad genética del 85.2% a la ECA2 humana, difiriendo en tan solo 4 de sus 20 residuos de contacto (Wu et al., 2020). La ECA2 del perro doméstico difiere en 5 de los 20 residuos de contacto, demostrando una identidad genética del 83.4% a la ECA2 humana (Stout et al., 2020). Asimismo, se encontró que la ACE2 es altamente expresada en la piel, punta de las orejas, pulmones y retina de los gatos, así como en la piel y retina de los perros (Sun et al., 2021). Esto indica que los gatos tienen alta susceptibilidad al virus, siendo mucho mayor a la del perro que, aunque pueden ser susceptibles, atraviesan una infección mucho más leve.
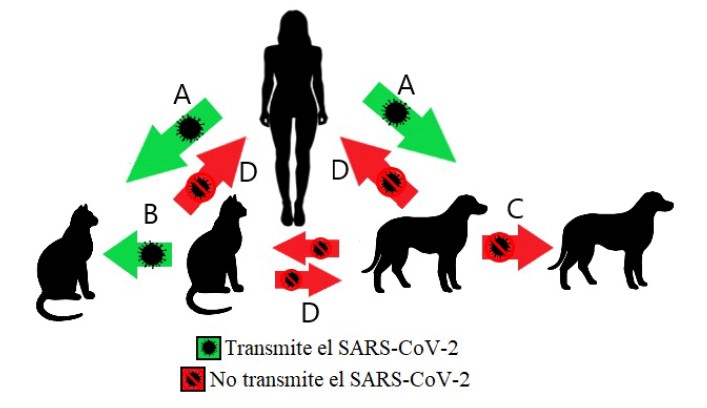
Si bien no se tienen pruebas de que los caninos y felinos domésticos juegan un papel relevante en la infección humana, se ha observado varios casos de zoonosis inversa o transmisión humano-animal (Figura 2) (Bosco-Lauth et al., 2020; Goraichuk et al., 2021). El primer evento epidemiológico de SARS-CoV-2 en un animal de compañía fue reportado el 28 de febrero de 2020 en Hong Kong. Se trató de un perro cuya propietaria fue hospitalizada por COVID-19, el cual dio un positivo leve mediante la técnica RT-qPCR de muestras obtenidas por hisopos nasales y orales (Sit et al., 2020). El primer reporte de un gato positivo a SARS-CoV-2 se realizó en Bélgica el 28 de marzo de 2020, y este caso fue el primer reporte de enfermedad por SARS-CoV-2 en un animal de compañía, ya que presentó signos clínicos, tanto respiratorios como entéricos, luego de estar en contacto con una persona positiva a COVID-19 (McNamara et al., 2020).
Experimentalmente, los felinos domésticos han demostrado ser altamente susceptibles a la infección por SARS-CoV-2, siendo capaces de transmitir por contacto directo el virus a otros gatos (Figura 2) (Bosco-Lauth et al., 2020) y de reinfectarse con el virus (Gaudreault et al., 2021). El primer estudio de infección natural por SARS-CoV-2 en gatos, realizado en Wuhan, reportó que 15 de 102 animales resultaron positivos al virus mediante pruebas de neutralización viral, y 3 de los 15 animales pertenecían a hogares con personas positivas a COVID-19, presentando los mayores títulos de neutralización del virus en el estudio (Zhang et al., 2020). En Francia se encontró una seroprevalencia del SARS-CoV-2 del 23.5% (8/34) en gatos que habitaban hogares COVID-19 positivos (Fritz et al., 2020); en Italia del 5.8% (11/191) (Patterson et al., 2020) y 20.4% (11/54) (Colitti et al., 2021); en Alemania, del 0.69% (6/920) (Michelitsch et al., 2020); en Brasil del 40% (4/10) (Calvet et al., 2021) y en Estados Unidos del 41.2% (7/17), sugiriendo que la transmisión humano-animal del SARS-CoV-2 puede ocurrir en felinos con mayor frecuencia de lo que se había esperado previamente (Hamer et al., 2021).
Los caninos domésticos demuestran susceptibilidad limitada al SARS-CoV-2 al ser inoculados experimentalmente y no desarrollan infección posterior al contacto directo con perros infectados (Figura 2) (Shi et al., 2020). La baja susceptibilidad de los caninos domésticos al SARS-CoV-2 no indica que no puedan infectarse naturalmente con el virus. Se han reportado seroprevalencias del virus en perros que habitan con personas positivas a COVID-19 del 43.9% (34/78) en Croacia (Stevanovic et al., 2021), 31% (9/29) en Brasil (Calvet et al., 2021), 15.4% (2/13) en Francia (Fritz et al., 2020) y 3.3% (15/451) (Patterson et al., 2020) y 3.2% (3/93) en Italia (Colitti et al., 2021), y 1.7% (1/59) en Estados Unidos (Hamer et al., 2021).
En el caso de los caninos y felinos domésticos positivos a SARS-CoV-2 a través de ARN viral en saliva y secreciones nasales, se ha observado que, aunque el virus tenga la capacidad de infectar a ambas especies, la diseminación viral de los animales de compañía puede no ser suficiente como para infectar al ser humano o a otros animales con los que tengan contacto breve (Figura 2) (Gaudreault et al., 2021; Valencak et al., 2021). Asimismo, son pocos los casos en los que se reportan signos de enfermedad por el SARS-CoV-2 en perros o gatos, debido a una pérdida de genes que genera deficiencias en las vías de activación del inflamasoma y la piroptosis, disminuyendo la inflamación excesiva y, como consecuencia, incrementando la tolerancia a la enfermedad (Cui y Zhang, 2021). Por último, hasta la fecha no existe evidencia de que caninos y felinos domésticos sean un factor de propagación del SARS-CoV-2 hacia el ser humano (Morais et al., 2020; Drózdz et al., 2021; Mostafavi et al., 2021).
Epidemiología SARS-CoV-2
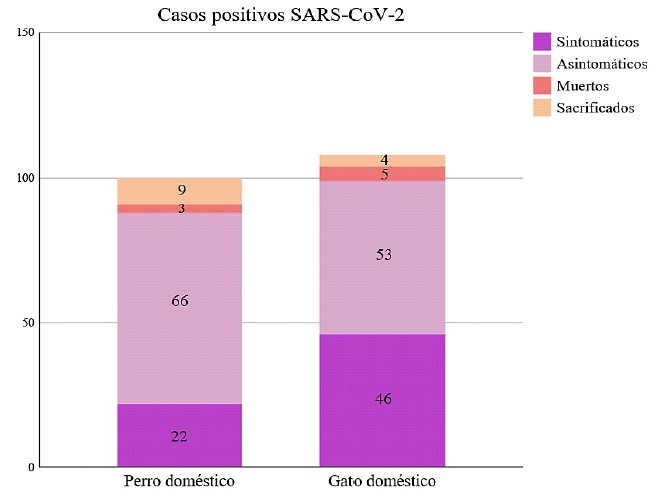
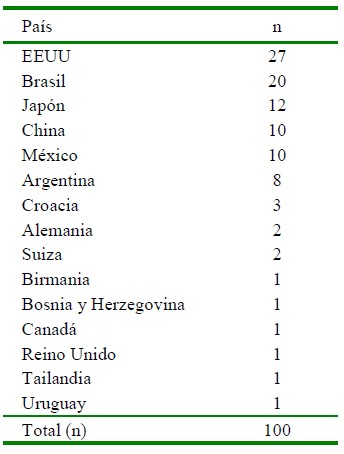
En el portal OIE-WAHIS se encontraron 39 informes que reportaron 407 caninos y felinos domésticos evaluados, de los cuales 208 fueron positivos para SARS-CoV-2. De estos, 100 casos fueron en perros, donde 73% fue diagnosticado en las Américas, principalmente en los Estados Unidos de Norteamérica (EEUU), 20% en Asia, principalmente en Hong Kong, y 13% en Europa (Cuadro 1). Del total de casos reportados, 34% de los caninos presentaron sintomatología respiratoria; mayormente dificultad respiratoria, fiebre, tos, conjuntivitis, depresión y anorexia; no obstante, la mayoría de los casos fueron asintomáticos (Bosco-Lauth et al., 2020; Shi et al., 2020; Perera et al., 2021). Entre EEUU, Alemania, Reino Unido y Argentina se reportó el sacrificio del 9% de los animales ante dificultad respiratoria, apatía y otros debido a la dificultad para hacer los seguimientos de los casos. Por otro lado, en un informe de EEUU y otro de México se expresó 3% de mortalidad debido a complicaciones de los signos clínicos presentados (Figura 3).
Cuadro 1 Reportes de casos positivos a SARS-CoV-2 en caninos domésticos enviados al portal OIE-WAHIS
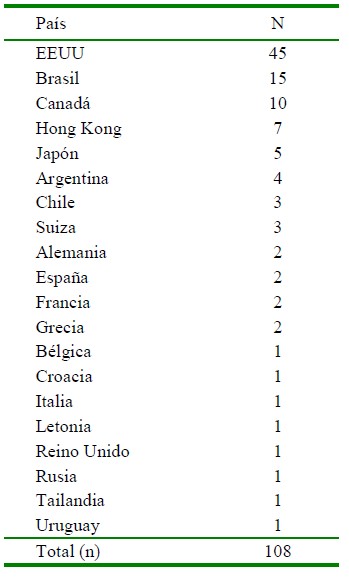
En los gatos se reportaron 108 casos positivos para SARS-CoV-2, siendo el 70% en las Américas, 18% en Europa y 12% en Asia (Cuadro 2). De estos, 55% expresó signos clínicos similares a los presentados por los caninos, además de secreción nasal/ocular purulenta, anorexia y vómito. Asimismo, seis informes no registraron la presentación de signos clínicos, lo que se asocia a lo expresado por varios autores (Newman et al., 2020; Sailleau et al., 2020; Pagani et al., 2021). A diferencia de los caninos, los gatos presentaron una tasa de mortalidad de 4.6% y de 3.7% de animales sacrificados debido a la severidad de los signos clínicos (Figura 3). Por otro lado, la presentación de casos positivos en 2020 fue de 60% para estas especies.
Cuadro 2 Reportes de casos positivos a SARS-CoV-2 en felinos domésticos enviados al portal OIE-WAHIS
Secuencias Genéticas del SARS-CoV-2
La vigilancia mundial de las secuencias genéticas del SARS-CoV-2 y de los metadatos relacionados facilita el seguimiento de la propagación geográfica y temporal del virus, así como la detección y análisis de mutaciones que puedan influir en el poder patógeno del virus o su transmisibilidad (OMS, 2021b). En este estudio se obtuvieron 141 secuencias genéticas del SARS-CoV-2 aisladas de caninos y felinos, de la cuales 50 pertenecían a perros (35.5%) y 91 a gatos (64.5%). Los reportes de estos aislamientos se realizaron desde 24 países a través de la plataforma GISAID (Figura 4). Estos reportes se organizaron en cinco regiones (Europa, Asia, Sudamérica, África y Norteamérica). La región con mayor frecuencia de genomas de SARS-CoV-2 fue en Norteamérica con 43.6%.
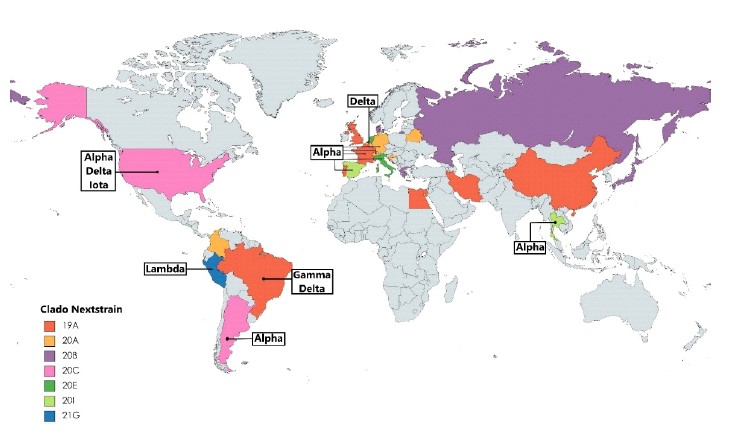
Figura 4 Distribución geoespacial de los aislamientos virales de SARS-CoV-2 de caninos y felinos domésticos, marcados con el clado Nextstrain más frecuente (colores por países) y señalados (recuadros en cada país) con las variantes preocupantes para la salud pública denominadas por la OMS
La caracterización genómica del SARS-CoV-2 se divide en varios clados, donde algunas mutaciones específicas definen los li- najes que circulan actualmente a nivel global (OPS, 2021b). La herramienta PANGOLIN define el conjunto de mutaciones en linajes ayudando a comprender los patrones y los determinantes de la propagación global del SARS-CoV-2; la nomenclatura se designa por letras en orden alfabético para cada linaje y a sus descendientes se les asigna un valor numérico (Rambaut et al., 2020). Por otro lado, el sistema de nomenclatura Nextstrain facilita el análisis de los patrones de diversidad del SARS-CoV-2 a gran escala, nombrando clados (grupo de linajes con un ancestro común) con base en sus combinaciones únicas de mutaciones (firma mutacional). Estos se denominan con el año en que surgen y una letra mayúscula iniciando por la A, siguiendo un orden de tipo alfabético (Hodcroft et al., 2020). Cada nuevo clado es nombrado e incluido si cumple con alguna de las siguientes tres condiciones:
Alcanza una frecuencia global >20% por dos o más meses, 2) Alcanza una frecuencia regional >30% por dos o más meses y 3) Se reconoce como una variante de preocupanción (VOC) (Nextstrain, 2021).
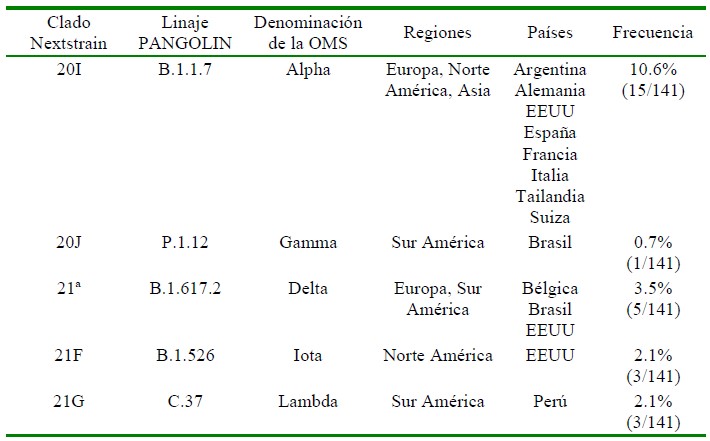
El linaje viral B.1 se aisló con mayor frecuencia de caninos y felinos (13.3%). Este linaje en el humano comprende el gran brote que se dio en Italia a inicios de 2020 y es el linaje global predominantemente conocido, siendo subdividido en más de 70 sublinajes (Rambaut et al., 2020). Los clados encontrados en los aislamientos virales del SARS-CoV-2, así como la frecuencia con la cual se presentan y la cantidad de mutaciones encontradas se presentan en el Cuadro 3 y graficados por medio de árboles filogenéticos en las figuras 5 y 6.
Cuadro 3 Frecuencia de clados SARS-CoV-2 aislados de caninos y felinos domésticos alrededor del mundo, reportados en la plataforma Nextclade1
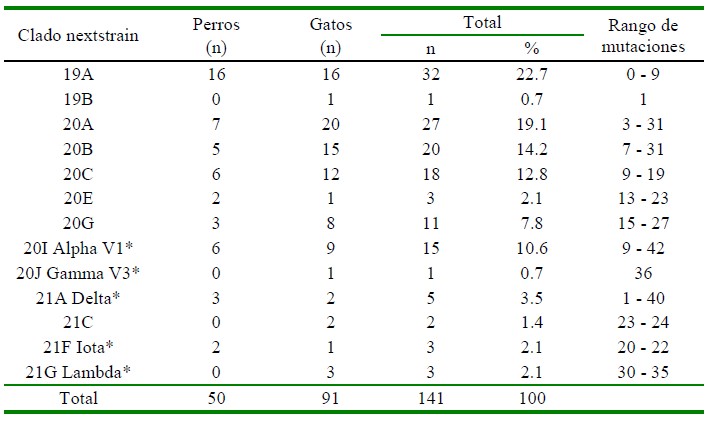
* Denominación de la OMS para el seguimiento de variantes de importancia para la salud pública
1 https://clades.nextstrain.org/
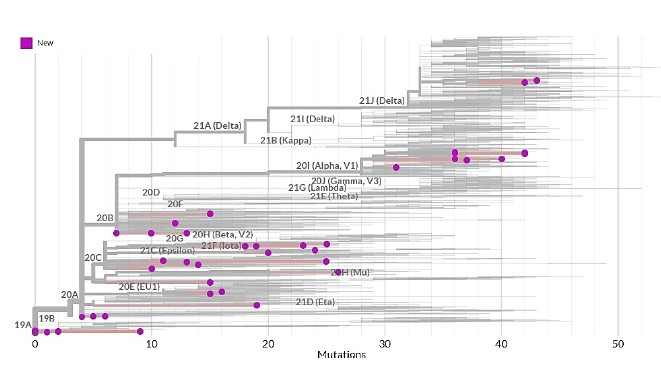
Figura 5 Árbol filogenético de los 50 aislamientos y secuenciaciones del SARS-CoV-2 en caninos registrados en GISAID. El clado raíz (19A) representa la secuencia de referencia Wuhan-Hu-1/2019, el primer caso aislado del SARS-CoV-2 en Wuhan-China. Los puntos violetas corresponden a cada secuencia genética del SARS-CoV-2 aislada de un canino y se ubican dependiendo a su clado correspondiente. Los principales clados que demuestran una frecuencia global actual >5% son: 20A, 20B, 20C, 20E, 20G y 20I (Nextstrain, 2021). El eje horizontal representa la distancia genética (mutaciones, tiempo), por lo que los grupos de secuencias genéticas ubicados hacia la derecha indican el acúmulo de mutaciones adicionales en su genoma a través del tiempo. Las variantes Alpha y Delta generaron mayor cantidad de mutaciones al infectar al canino doméstico, demostrado por más de seis secuencias genéticas que acumulan por encima de 30 mutaciones
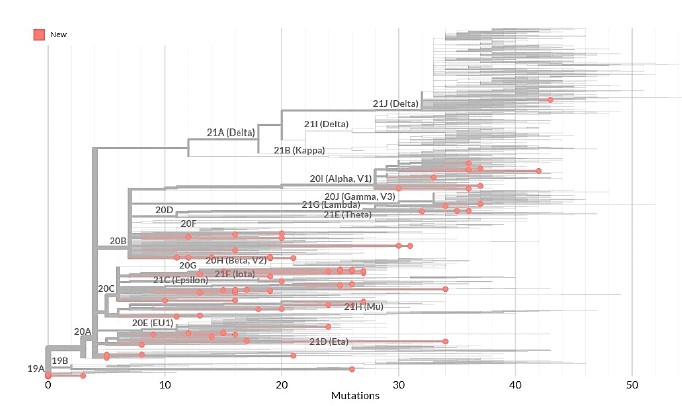
Figura 6 Árbol filogenético de 91 aislamientos y secuenciaciones del SARS-CoV-2 en felinos domésticos registrados en GISAID. El clado raíz (19A) representa la secuencia de referencia Wuhan-Hu-1/2019. Los puntos rosados corresponden a cada secuencia genética del SARS-CoV-2 aislada de un felino doméstico y se ubican dependiendo a su clado correspondiente. Los principales clados que demuestran una frecuencia global actual >5% son: 20A, 20B, 20C, 20E, 20G y 20I (Nextstrain, 2021). El eje horizontal representa la distancia genética (mutaciones, tiempo), por lo que los grupos de secuencias genéticas ubicados hacia la derecha indican el acumulo de mutaciones adicionales en su genoma a través del tiempo. Al infectar a felinos domésticos las cepas con las variantes Alpha, Gamma, Delta y Lambda generaron mayor cantidad de mutaciones (>40) y al menos 15 secuencias genéticas acumularon por encima de 30 mutaciones
Los coronavirus manifiestan gran propensión a la transmisión interespecie, donde la mayoría de las mutaciones genéticas o deleciones/inserciones en los genes de sus proteínas virales ocurren luego de un cambio de huésped (host-shift) o un cambio en el tropismo tisular del virus (Islam et al., 2021). Cuando el genoma viral contiene una o más mutaciones, se puede indicar de una variante del virus. En algunos casos, las variantes con cambios genéticos similares pueden ser designadas por las organizaciones de salud pública como variantes preocupantes (VOC) o variantes de interés (VOI) y serán nombradas con una letra del alfabeto griego, correspondiendo al sistema de nomenclatura propuesto por la OMS para las variantes de importancia (OMS, 2021c). Estas variantes son agrupadas dependiendo a las características compartidas que supongan un riesgo para la salud pública (CDC, 2021), como lo son el aumento de la transmisibilidad y virulencia, la disminución de la eficacia de medidas sociales y de salud pública o de los métodos diagnósticos, las vacunas y los tratamientos disponibles (OPS, 2021a).
Hasta el momento, la variante Alpha (B.1.1.7), una VOC identificada por primera vez en Inglaterra (PHE, 2020), es la más reportada en caninos y felinos domésticos. El primer reporte de la variante en animales de compañía fue realizado por Ferasin (2021) e identificada en dos gatos y un perro. Posteriormente fue reportada en Estados Unidos en un canino y un felino, ambos asintomáticos, de un mismo hogar con propietario positivo a la variante (Hamer et al., 2021), en Francia en un gato que convivía con personas positivas a la variante (Krafft et al., 2021), en España en un canino que presentó cargas virales elevadas en frotis nasales y rectales (Barroso-Arévalo et al., 2021), en Alemania en un gato con neumonía (Keller et al., 2021) y en Italia en un felino cuya familia era positiva a la variante (Zoccola et al., 2021).
El análisis filogenético confirmó que se han aislado cinco variantes monitoreadas por las organizaciones de salud pública: las VOC Alpha, Delta y Gamma, y las VOI Iota y Lambda (Cuadro 4). No obstante, aún no está claro si las variantes del SARS-CoV-2 se asocian a mayor transmisibilidad o virulencia al infectar animales de compañía (Krafft et al., 2021); sin embargo, se ha demostrado que la mutación D614G, presente en todas las VOC, demuestra una eficacia de infección de 8-10 veces mayor que la cepa original del virus, así como mayores títulos de ARN del SARS-CoV-2 en muestras aisladas de pacientes infectados con la cepa que presenta la mutación (Tian et al., 2021). Adicionalmente, se encontró que los virus pseudotipados que presentan mutaciones en la proteína S de las variantes Alpha y Gamma presentaron alteraciones relacionadas a una mayor capacidad de entrada viral en comparación a la proteína S de la cepa original (prototipo Wuhan-1) (Kim et al., 2021).
CONCLUSIONES
La zoonosis reversa es un evento frecuente, como es demostrado en más de 200 caninos y felinos domésticos infectados naturalmente con el SARS-CoV-2 y en las prevalencias de infección en perros y gatos que habitan en hogares positivos a COVID-19 (Fritz et al., 2020).
La ocurrencia de infección por SARS-CoV-2 en caninos y felinos domésticos puede ser el resultado de un efecto de spillover desde el humano (Sharun et al., 2021).
La mayoría de las infecciones cursaron sin signos clínicos, sugiriendo que el riesgo de transmisión perro-humano o gato-humano es prácticamente nulo en este contexto (Krafft et al., 2021).
Se reporta por primera vez la infección de perros y gatos con las VOC Delta y Gamma, y las VOI Iota y Lambda, hallazgo posible gracias al análisis filogenético de la totalidad de secuencias del SARS-CoV-2 aisladas de caninos y felinos domésticos.












 uBio
uBio