INTRODUCCIÓN
La enfermedad infecciosa bursal, también conocida como enfermedad de Gumboro, es una enfermedad viral, altamente contagiosa e inmunosupresora que afecta a aves jóvenes, siendo responsable de importantes pérdidas económicas en la industria avícola a nivel mundial. Es producida por un virus ARN de doble cadena bisegmentado (segmentos A y B) del género Avibirnavirus y familia Birnaviridae (Murphy et al., 1999).
El segmento A contiene dos marcos de lectura que se solapan parcialmente. El marco de lectura abierto 1 (ORF1, por sus siglas en inglés Open Reading Frame) codifica una poliproteína de 110 kDa que se empalma autocatalíticamente en las proteínas virales VP2 (48 kDa), VP3 (33-35 kDa) y VP4 (24 kDa), en tanto que el marco de lectura abierto 2 (ORF2) codifica a una proteína no estructural de 17 kDa, la VP5 (Toskano Hurta- do, 2016). El segmento B tiene 2.8 kDa de longitud y codifica a la proteína VP1, una proteína de 97 kDa con actividad polimerasa dependiente de ARN y se presenta tanto libre como ligada al genoma (Toskano Hurta- do, 2016). Esta última desempeña un papel clave en la encapsulación de las partículas virales e interactúa con VP3 para formar el complejo VP1-VP3 que da integridad estructural a las partículas virales (Mahgoub, 2012; Dey et al., 2019).
La forma de prevención de la enfermedad es mediante la vacunación de las parvadas (vacunas de virus atenuados, inactivados, recombinantes que contienen la proteína VP2) y mediante el empleo de inmunocomplejos antígeno-anticuerpo (Mahgoub, 2012; Toskano Hurtado, 2016; Wu et al., 2020). Pese a la existencia de una gran variedad de vacunas, el control de la enfermedad sigue siendo un reto, debido a estimaciones epidemiológicas inexactas (Sun et al., 2013). Ante esto, la comunidad científica desarrolla nuevas estrategias como la búsqueda de compuestos inhibidores de proteínas para combatir las afecciones de origen viral.
La primera etapa en la búsqueda y desarrollo de nuevas moléculas con potencial uso farmacológico utiliza recursos computacionales enfocados en el análisis de las propiedades fisicoquímicas, toxicológicas, farmacéuticas, ADMET, entre otras, de las moléculas; así como, de las interacciones ligando-proteína en los sitios activos/catalíticos o alostéricos que conlleven hacia una potencial actividad biológica, optimizando de esta manera la inversión de tiempo y costo en el diseño de nuevos fármacos (Anwar et al., 2021). Generalmente la búsqueda se realiza con productos naturales, ya que las plantas son fuentes inagotables de compuestos que presentan actividades antivirales contra una considerable cantidad de virus (Kapoor et al., 2017). Los mecanismos de acción pueden ser varios, como la inhibición de una o varias etapas importantes en el ciclo replicativo, la entrada viral o el proceso de replicación del vi- rus (Kapoor et al., 2017).
La especie Whitania somnifera (L) Dunal es ampliamente empleada principal- mente por sus propiedades antimicrobianas, utilizada para el tratamiento de afecciones como la tuberculosis y enfermedades de transmisión sexual (ETS) (Gupta y Rana, 2007). También se ha demostrado actividades antivirales contra el virus del herpes simplex tipo I (VHS-1) (Kambizi et al., 2007). Asimismo, Mofed et al. (2020) han reportado reducciones de la carga viral del virus de la hepatitis C en el tratamiento con el extracto acuoso de W. somnifera en concentraciones de 25 mg.ml-1.
Pant et al. (2012) trabajaron con el extracto hidroalcohólico de W. somnifera de- mostrando su eficacia al inhibir la replicación del virus de la enfermedad infecciosa bursal (IBDV, por sus siglas en inglés Infectious Bursal Disease Virus) en un 99.9% a con- centraciones seguras (25 µg.mL-1) y no citotóxicas; sin embargo, se desconoce el mecanismo de acción. El objetivo del presente estudio fue identificar los fitoconstituyentes descritos en W. somnifera que presenten afinidades de unión con el sitio activo de la pro- teína viral 1 (VP1) del IBDV y caracterizarlas mediante el análisis computacional de acoplamiento molecular.
MATERIALES Y MÉTODOS
Caracterización de sitios drogables en la región catalítica de la proteína VP1
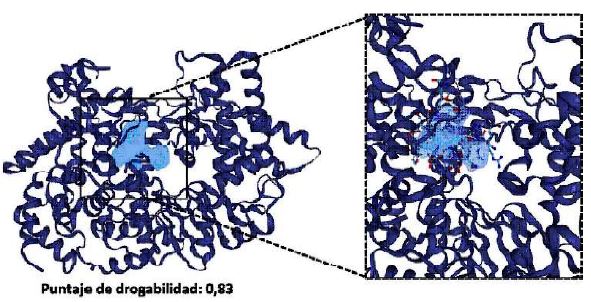
El estudio se realizó siguiendo el esquema metodológico representado en la Figura 1. Para ello, primeramente, se hizo la búsqueda de sitios con perfiles drogables presentes en la región catalítica de la VP1 (PDB: 2R70, 2R72), siendo identificados y caracterizados empleando la herramienta DoGSite- Scorer de ProteinsPlus (Fährrolfes et al., 2017; Schöning-Stierand et al., 2020). La caracterización fue realizada mediante descriptores como las dimensiones del sitio drogable, determinación de grupos funcionales y la composición de residuos; además, se estimó el puntaje de drogabilidad (Volkamer et al., 2012).
Pruebas de acoplamiento molecular entre fitoconstituyentes de W. somnifera y la VP1
La estructura proteica de la VP1 del IBDV fue obtenida de la base de datos Protein Data Bank (PDB: 2R70) (Garriga et al., 2007). La estructura molecular de los fitoconstituyentes descritos en W. somnifera se obtuvieron de la base de datos PubChem (Cuadro 1) (Gupta y Rana, 2007).
Primeramente, se procedió a eliminar las moléculas de agua presente en la estructura proteica, luego se aplicó el método de campo de fuerza UFF (Universal Force Field) junto con el algoritmo de optimización de gradientes conjugados, y a continuación se proporcionaron de cargas parciales y de átomos de hidrógeno polares presentes a pH fi- siológico de 7.4, utilizando el programa Avogadro 1. 2 (Hanwell et al., 2012).
Las pruebas exploratorias de acopla- miento molecular se llevaron a cabo utilizan- do el modelo tipo receptor-ligando, utilizando una caja de acoplamiento con dimensiones de 21.22 x 25.15 x 18.50 Å3 dirigido al sitio drogable identificado en el sitio catalítico. Para ello se emplearon los programas LeDock (Zhao y Caflisch, 2013) y AutoDock Vina (Trott y Olson, 2010). Como controles del sistema de acoplamiento se emplearon a los ribonucleótidos adenosina trifosfato (ATP), guanosina trifosfato (GTP), citosina trifosfato (CTP) y uridina trifosfato (UTP).
De los resultados obtenidos se seleccionaron aquellos ligandos que presentaron va- lores de energía libre de unión (ΔG) más favorables (baja energía). Estos fueron sometidos a pruebas de mayor exhaustividad (exhaustividad = 20) haciendo cincuenta repeticiones mediante el programa AutoDock Vina (Trott y Olson, 2010), a modo de minimizar el grado de incerteza y reducir los erro- res de las simulaciones. La precisión de los procedimientos de acoplamiento molecular se garantizó volviendo a acoplar el ligando cocristalizado con la proteína (PDB: 2R72) y calculando valor de RMSD (raíz de la desviación cuadrática media). La visualización y el análisis de los complejos resultantes de las pruebas de acoplamiento molecular se realizaron utilizando el programa Discovery Studio Visualizer v. 20 (BIOVIA Dassault Systèmes, 2020) y Visual Molecular Dynamics (Humphrey et al., 1996).
Predicción de propiedades ADME de fitoconstituyentes de W. somnifera
Las propiedades ADME (absorción, distribución, metabolismo y excreción) de los fitoconstituyentes que presentaron valores más favorables de energía de unión fueron caracterizadas empleando la regla de Lipinski (Butina et al., 2002; Selick et al., 2002), utilizando la herramienta SwissADME (Daina et al., 204, 2017; Daina y Zoete, 2016).
Análisis estadísticos
Los datos obtenidos en las pruebas de acoplamiento molecular fueron analizados mediante el test de Kruskal-Wallis y la prueba post-hoc de Dunn (5% de nivel de significancia), utilizando el programa Past 4. 08 (Hammer et al., 2001).
RESULTADOS Y DISCUSIÓN
El análisis de las estructuras de la VP1 evidenció la presencia de un sitio con un alto puntaje de drogabilidad igual a 0.83 ubicado en la región catalítica de la proteína, según fue descrito por Garriga et al. (2007), quienes reportaron que los residuos activos del sitio catalítico de la VP1 son Arg175, Lys333, Arg335, Asp402, Asn403, Asp416, Lys419, Glu421, Asn493 y Lys529. Este sitio presenta un volumen de 579,49 Å3, una superficie de 69.31 Å2 y una profundidad de 21.53 Å (Figura 2). Según Volkamer et al. (2012) los índices de drogabilidad pueden tomar valores desde 0 a 1, donde aquellos puntajes cercanos a 1 indican su potencial uso como blanco para la búsqueda de inhibidores, siendo considerado puntajes mayores a 0.50 como valores óptimos (Volkamer et al., 2012).
Cuadro 1. Resultados obtenidos en las pruebas de acoplamiento molecular
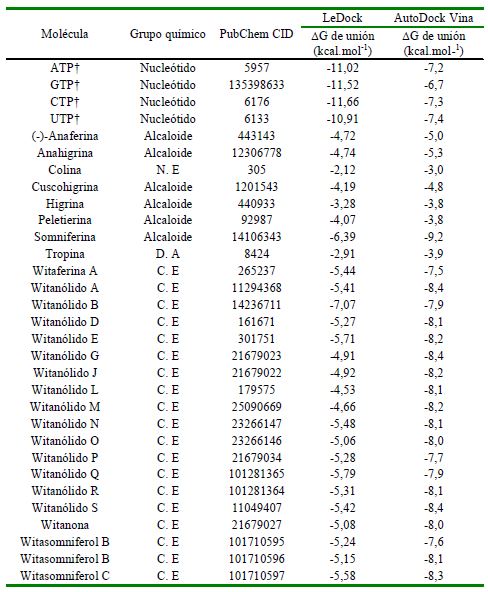
† Controles, N. E: Nutriente esencial, D. A: Derivado alcaloide, C. E: Compuestos esteroídicos
El sitio drogable se encuentra conformado por residuos apolares en un 28%, residuos polares en un 47%, residuos básicos en un 19% y residuos ácidos en un 6%. Los residuos son Tyr59, Arg175, Leu176, Met179, Lys180, Ser256, Ser257, Lys267, Leu318, Lys321, Arg335, Asn336, Ile337, Trp338, Ser339, Ala340, His345, Glu421, Ala422, Val465, Val466, ASP467, Ser468, Ser469, Cys470, Lys478, Thr479, Tyr480, Gly481, Gln482, Gly483, Ser484, Gly485, Asn486, Ala488 y Thr489, de entre los cuales Arg175, Arg335 y Glu421 son residuos que se encuentran cumpliendo funciones importantes en el sitio catalítico de la VP1 (Garriga et al., 2007).
El sitio se encuentra conformado por 22 grupos funcionales capaces de actuar como donadores en puentes de hidrógeno, 54 grupos funcionales aceptores en puentes de hidrógeno y 13 grupos funcionales capaces de participar en la formación de interacciones hidrofóbicas, con una proporción de hidrofobicidad de 0.15.
Los resultados de las pruebas exploratorias de acoplamiento molecular demostraron que los fitoconstituyentes con valores más favorables de energía libre de interacción fueron la somniferina, los witanólidos A, N, R, S, y el witasomniferol C. Estos compuestos fueron seleccionados para llevar a cabo pruebas de mayor exhaustividad (Cuadro 1), pruebas que revelaron que estos fitoconstituyentes presentan valores significativa- mente favorables (p<0.001) de energía libre de interacción con la región catalítica de la VP1 en comparación con a los resultados obtenidos con los controles (Figura 3).
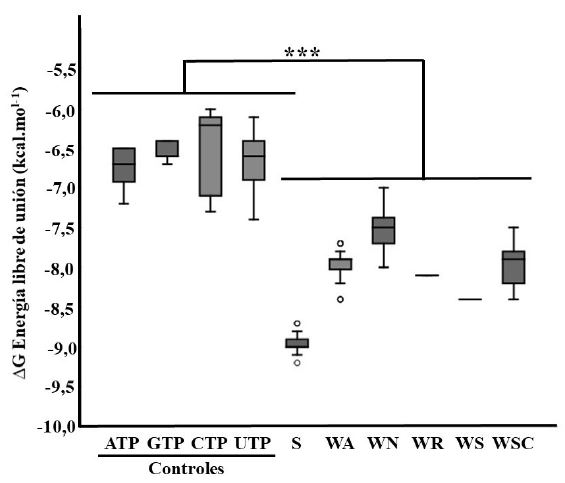
Figura 3 Energía libre de interacción (ΔG) registrada en las pruebas con mayor exhaustividad. S: Somniferina, WA: witanólido A, WN: witanólido N, WR: witanólido R, WS: witanólido S, WSC: witasomniferol C (***p<0.001)
La somniferina presentó una energía libre de unión igual a -8.92±0.15 kcal.mol-1 y resultó ser el ligando con la con la energía más baja detectada en comparación a los otros fitoconstituyentes. Los residuos activos identificados fueron Arg175, Met179, Arg335, Asp402, Lys419, Glu421 y Arg592, donde Arg175, Arg335, Asp402, Lys419 y Glu421 son residuos con roles importantes en el sitio catalítico de la VP1 (Garriga et al., 2007).
Se pudo registrar interacciones del tipo puentes de hidrógeno entre la somniferina y el residuo Arg335, interacciones del tipo car- bono-hidrógeno no convencionales con el Asp402, interacciones electroestáticas con los residuos Arg175 y Arg532 (orbitales π-catión) y con el residuo Glu421 (orbitales π-anión), interacciones entre cadenas de alquilo con la Met179 e interacciones entre orbitales π y las cadenas de alquilo con los residuos Lys419 y Arg335 (Figura 4.A).
El witanólido A reveló una energía libre de unión de -7.98±0.20 kcal.mol-1, donde los residuos activos registrados fueron Arg175, Asn403 y Asn493, con los cuales solo se identificaron la formación de puentes de hidrógeno convencionales, donde Arg175 y Asn493 son aminoácidos con funciones importantes en el sitio catalítico (Garriga et al., 2007) (Fi- gura 4.B). A su vez, el witanólido N evidenció una energía libre de unión de -7.56±0.28 kcal.mol-1. Los residuos que se encontraban participando activamente en la formación del complejo con la VP1 son Arg175, Asp402 y Asn493, con los cuales el ligando se encontraba interaccionando mediante puentes de hidrógeno; no obstante, Arg175, Asp402 y Asn493 son asimismo residuos importantes del sitio catalítico (Garriga et al., 2007) (Figura 4.C).
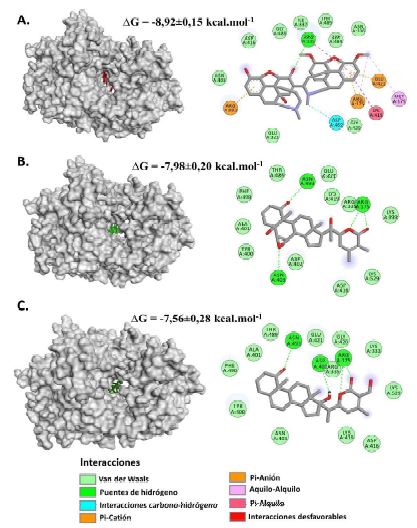
Figura 4 Representación gráfica tridimensional y bidimensional del acoplamiento molecular entre fitoconstituyentes de W. somnifera y la región catalítica de VP1. A. Somniferol. B. Witanólido A. C. Witanólido N
El witanólido R demostró una energía libre de -8.09±0.014 kcal.mol-1 en unión a la región catalítica de la VP1. Los residuos activos registrados fueron Arg175, Asn403 y Asn493, con los cuales se formaron puentes de hidrógeno con el ligando (Figura 5.A).
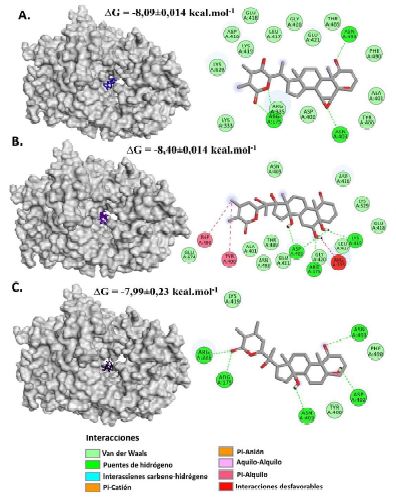
Figura 5 Representación gráfica tridimensional y bidimensional del acoplamiento molecular entre fitoconstituyentes de W. somnifera y la región catalítica de VP1. A. Witanólido R. B. Witanólido S. C. Witasomniferol C
Estos residuos cumplen roles importantes en el sitio catalítico de la VP1 (Garriga et al., 2007).
El witanólido S presentó una energía libre de unión de -8.40±0.014 kcal.mol-1. Los residuos activos fueron la Arg175, Asp402, Lys419, Tyr400 y Phe490. Se detectó la formación de puentes de hidrógeno entre el ligando y los residuos activos Arg175, Asp402 y Lys419. También se registró la formación de interacciones entre los orbitales π de las cadenas laterales de los residuos Tyr400 y la Phe490 y las cadenas de alquilo del ligando. Sin embargo, se identificó la presencia de interacciones desfavorables entre el ligando y el residuo Arg335 lo cual es generado debido a repulsiones existentes entre átomos del residuo y del ligando, lo cual podría reducir la estabilidad del complejo (Dhorajiwala et al., 2019) (Figura 5.B). Entre estos, los residuos Arg175, Asp402 y Lys419 son aminoácidos con funciones importantes en el sitio catalítico de la enzima (Garriga et al., 2007).
Cuadro 2. Propiedades ADME según la regla de Lipinski de los fitoconstituyentes con valores energéticos favorables
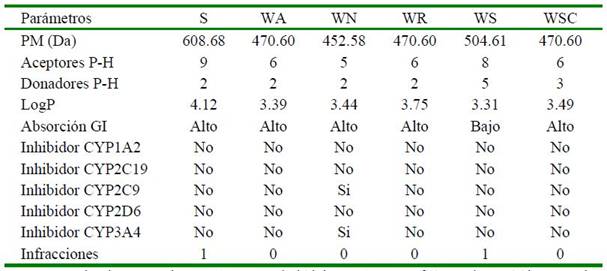
PM: peso molecular, Da: Dalton, P-H: puente de hidrógeno, LogP: coeficiente de partición octanol- agua, GI: gastrointestinal, CYP: citocromo P450. S: Somniferina, WA: witanólido A, WN: witanólido N, WR: witanólido R, WS: witanólido S, WSC: witasomniferol C.
Finalmente, el compuesto witasom- niferol C demostró una energía de interacción de -7.99±0.23 kcal.mol-1. Los residuos activos identificados fueron Arg175, Arg335, Asp402, Asn403 y Asn493, con los cuales el ligando se encontraba formando puentes de hidrógeno convencionales (Figura 5.C). Todos los residuos registrados como activos con el witasomniferol C son aminoácidos que cumplen papeles importantes en el sitio activo de la VP1 (Garriga et al., 2007). La evaluación de la precisión de los ensayos mediante el redocking del ligando cocristalizado reveló un valor de RMSD igual a 2.1 Å, sien- do este valor considerado como óptimo y aceptable para la validación del procedimiento de acoplamiento molecular (Mateev et al., 2022).
La regla de Lipinski, también conocida como regla de los cinco, establece que las moléculas con aptitudes farmacológicas para su administración oral deben poseer un peso molecular no mayor a los 500 Da, al igual que un coeficiente de participación octanolagua (LogP) no mayor a 5. Este ofrece in- formación sobre la lipofilicidad de las molé- culas. Asimismo, es requisito de la regla que las moléculas analizadas no posean un número mayor a 10 grupos aceptores de puentes de hidrógeno y un número no mayor a 5 grupos que actúen como donadores en los puentes de hidrógeno (Lipinski, 1995).
Los fitoconstituyentes analizados revelaron la presencia de solamente una infracción a la regla de Lipinski en los ligandos somniferina y el witanólido S, los cuales poseen un peso molecular mayor a lo recomen- dado por la regla (>500 Da); sin embargo, los productos naturales generalmente rompen la regla de los 5 debido que los mismos son capaces de mantener el equilibrio entre el peso molecular de los compuestos, la cantidad de enlaces rotables, la capacidad de actuar como donadores en puentes de hidrógeno y la hidrofobicidad de la molécula. lo cual hace que los productos naturales presenten ventajas sobre drogas sintéticas (Ganesan, 2008) (Cuadro 2).
Es importante destacar que las predicciones sugieren que todos los fitoconstituyentes analizados presentan un alto perfil de absorción gastrointestinal excepto el witanólido S que demostró una baja absorción. Así también, las predicciones realiza- das revelaron solo a la molécula del witanólido N como posible inhibidor de la proteína citocromo P450 (CYP), específicamente a las isoformas CYP2C9 y CYP3A4; sin embargo, el resto de los fitoconstituyentes analizados no demostraron ser inhibidores del CYP en ninguna de sus isoformas (Cuadro 2).
El análisis predictivo ADME sugiere que todos los fitoconstituyentes analizados presentan aptitudes para el desarrollo farmacológico, presentando resultados favorables para biodisponibilidad en sangre y para la difusión a través de las membranas (Bhal, 2019).
Muchos estudios se han enfocado en la búsqueda de constituyentes vegetales con actividades antivirales que puedan ayudar a combatir el avance del IBDV, tal es el caso de los resultados reportados por Sun et al. (2013), quienes empleando compuestos empleados en la medicina natural china evidenciaron que las moléculas glicirricato de dipotasio y clorhidrato de ligustrazina presentan actividades antivirales contra el virus con EC50 iguales a 663.2±268.4 and 92.52±21.13 mg.ml-1 e índices de selectividad mayores de 4.52 y 21.62, respectivamente, demostrando acciones de inhibición del 70-80%.
Ahmad et al. (2014) reportaron potenciales actividades antivirales del extracto etanólico de raíces de Glycyrrhiza glabra, frutos secos de Phyllanthus emblicus, hojas de Moringa oleifera y Eugenia jambolana contra el IBDV en concentraciones de 12.5, 25, 50 y 100 µg.ml-1, respectivamente. Así también, Anyanwu et al. (2017) demostraron que los alcaloides aislados de la pulpa del fruto de Cucumis metuliferus presentan actividades antivirales contra el IBDV a concentraciones de 6.125 y 100 mg.ml-1, las cuales fueron seguras para el modelo biológico empleado para el estudio (células fibroblásticas embrionarias de pollo). Por otro lado, Pant et al. (2012) hallaron actividades antivirales importantes del extracto hidroalcohólico de las raíces de W. somnífera contra el IBDV, el cual evidenció un 99.9% de inhibición de la replicación viral a una concentración no tóxica de 25 µg.ml-1, aunque no describieron el posible mecanismo de acción.
La proteína viral 1 (VP1) es una proteína importante para el IBDV; sin embargo, el mecanismo de activación depende de la interacción con la proteína viral 3 (VP3), por lo que es considerado como una APDA no canónica (von Einem et al., 2004; Garriga et al., 2007). Entre los residuos activos identificados en los complejos formados con los fitoconstituyentes analizados en este estudio, muchos de ellos son importantes para el correcto funcionamiento de la VP1; entre estos, la Arg175, Arg335, Asp402 y Asn403, los cuales cumplen una función importante en la estabilización de los nucleótidos que van ingresando a la región catalítica de la enzima (Garriga et al., 2007).
Los residuos Asp402 y Glu421 se en- cargan específicamente de recibir a los nucleótidos ingresantes. Así también la Lys419 es uno de los residuos implicados en la activación previa de la VP1 por interacción con la VP3 (Garriga et al., 2007).
Se han reportado numerosas propiedades biológicas de los witanólidos y sus deriados como la capacidad que poseen de inhibir enzimas importantes de microorganismos, al igual que los metabolitos esenciales para el metabolismo del microorganismo (Budhiraja et al., 2000). En parte estas propiedades se deben también a la capacidad que presentan este grupo de moléculas de interaccionar con las membranas celulares facilitando el ingre- so a las células, principalmente aquellos compuestos derivados de estructuras esteroidales, facilitando de esta manera sus acciones como antimicrobianos e inhibidores proteicos (Kopeæ et al., 2013).
CONCLUSIONES
El análisis computacional reveló que los fitoconstituyentes de W. somnifera que presentaron afinidades de interacción significativamente favorables (p<0.001) por el sitio catalítico de la VP1 del IBDV fueron la somniferina, witanólido A, witanólido N, witanólido R, witanólido S, witasomniferol C, los cuales demostraron valores de energía libre de interacción de entre -7.56±0.28 a -8.92±0.15 kcal.mol-1.
Los residuos activos (Arg175, Arg335, Asp402, Asn403 y Glu421) comprometidos en la formación de los complejos con los fitoconstituyentes, poseen funciones importantes en la VP1 durante el proceso de síntesis del nuevo genoma viral.
Los fitoconstituyentes que demostraron interacciones favorables con la región catalítica de la VP1 presentaron predicciones favorables de ADME ajustándose con la regla de los cinco de Lipinski, lo cual favorece la selección de estos compuestos para su potencial investigación farmacológica.
Los hallazgos sugieren que estos compuestos presentes en W. somnifera poseen características de ser candidatos como potenciales inhibidores de la actividad de la VP1 del IBDV y, de esta manera, interferir con el ciclo replicativo del virus; sin embargo, están sujetas a ensayos in vitro para confirmar estas observaciones.












 uBio
uBio