INTRODUCCIÓN
La resistencia antimicrobiana se ha convertido en una problemática importante en salud pública, que involucra tanto humanos como animales, y que ha afectado notablemente los resultados de los tratamientos de una enfermedad específica. Se han establecido factores que propician la resistencia, tales como el uso excesivo de antibióticos, sobre todo en infecciones que no las requieren, dosis incorrectas de los medicamentos o tratamientos incompletos por falta de recursos económicos (OMS, 2001). Ante esto, la Organización Mundial de la Salud (OMS) ha establecido estrategias para contener la resistencia contra los antimicrobianos, tanto los utilizados en el humano como en los animales, una de las cuales consiste en la vigilancia de resistencia antimicrobiana mediante métodos in vivo e in vitro. En este último se encuentran la clasificación fenotípica y molecular (pruebas genéticas) que están sien- do utilizado en diversos países para detectar genes de resistencia antimicrobiana en animales de producción (Boerlin et al., 2005; Brand et al., 2017; Mazurek et al., 2018).
Escherichia coli es una bacteria gram negativa comensal, presente en el tracto digestivo de humanos y animales. También es el agente causal de diversas enfermedades en los porcinos, como la colibacilosis neonatal y posdestete que cursa con diarrea, causan- do altas tasas de morbilidad con disminución en la producción e incremento de los costos en el tratamiento, además de mortalidad, sien- do esta una de las enfermedades de mayor relevancia en la industria porcina (Luppi, 2017; Zimmerman et al., 2019).
Según reportes de uso de antimicrobianos de Canadá (CIPARS, 2018), los principales antibióticos utilizados en la producción porcina para el tratamiento de colibacilosis entéricas son las tetraciclinas, apramicina, sulfonamidas, estreptomicina y amoxicilina-ácido clavulánico, observando cada vez más resistencia contra estos en los protocolos de tratamiento (Luppi, 2017). El problema con esta enfermedad se ha exacerbado debido al uso inadecuado de los antibióticos en los animales (Burow et al., 2014), donde médicos veterinarios y productores reportan casos de colibacilosis porcina y otras enfermedades bacterianas en anima les que son resistentes a diferentes protocolos terapéuticos (Van Boeckel et al., 2015). Ante esto, y con el fin de evaluar la situación local, el objetivo del estudio fue determinar la frecuencia de genes de resistencia antimicrobiana en aislados de Escherichia coli en cerdos con cuadros diarreicos mediante PCR convencional.
MATERIALES Y MÉTODOS
Material Experimental
Se utilizaron 119 aislados de Esche- richia coli obtenidos de muestras de intestino de cerdos con cuadros diarreicos remitidas al Laboratorio de Bacteriología de la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos, en Lima, Perú, Perú por veterinarios y productores de 12 granjas porcinas tecnificadas de la región de Lima entre 2017 y 2020.
Cuadro 2 Frecuencia de resistencia antimicrobiana a cuatro grupos de antibióticos de 119 aislados de E. coli obtenidos de cuadros diarreicos en cerdos (Región Lima, 2017- 2020)
Reactivación de Aislados y Extracción del ADN
Los aislados se reactivaron en caldo Tripticasa de soya (Merck, Alemania) y posteriormente fueron sembrados en agar MacConkey (Merck, Alemania). La extracción de ADN genómico se realizó con el kit de extracción de ADN GeneJET Genomic DNA Purification Kit (Thermo Scientific, USA) de acuerdo con las instrucciones del fabricante. Una vez extraído el ADN se preservó a -20 °C.
Identificación de Genes de Resistencia por PCR
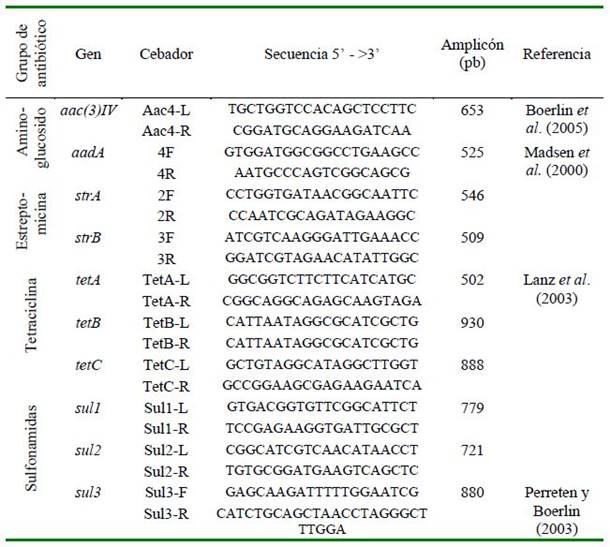
Para la estandarización de los esquemas de PCR para cada gen se tomó como referencia los protocolos y los cebadores empleados por Madsen et al. (2000), Lanz et al. (2003), Perreten y Boerlin (2003) y Boerlin et al. (2005) para cuatro grupos de antibióticos (Cuadro 1).
Se amplificó cada gen por muestra con las siguientes condiciones: en un volumen de 20 µl con una concentración de 0.2 µM de cada cebador y 10 µl de 2X PCR HotStart MasterMix (ABM, Canadá) y 1 µl ADN (30- 70 ng/µl). Las condiciones del termociclador incluyeron una desnaturalización inicial a 95 ºC durante 4 min, seguido por 35 ciclos de 95°C de desnaturalización durante 1 min, hibridación durante 1 min con la temperatura respectiva según el gen (Cuadro 1) y 72 °C para la elongación durante 1 min, con una elongación final de 72 °C durante 7 min.
Para la corrida electroforética, se preparó el gel de agarosa al 1.5% con buffer TBE al 0.5X durante 80 min a 90 v. Los productos fueron analizados con el buffer de carga SafeGreen (ABM, Canadá) en el transiluminador SafeVIEW (Clever Scientific, UK) de luz azul.
Reactivación de Aislados y Extracción del ADN
RESULTADOS
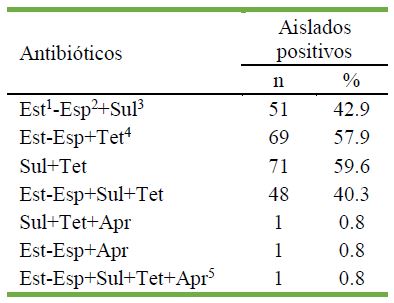
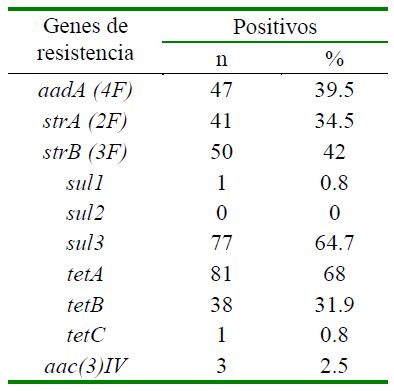
El 98.3% (117/119) de las muestras fueron positivas a por lo menos un gen de resistencia antimicrobiana, destacando el grupo de tetraciclinas (Cuadro 2). Los antibióticos que presentaron resistencia antimicrobiana a más de dos antimicrobianos se presentan en el Cuadro 3, siendo el grupo de las sulfonamidas con las tetraciclinas las de mayor frecuencia. Con respecto a los genes de resistencia antimicrobiana, el gen tetA fue el de mayor frecuencia (Cuadro 4), y en los casos donde hubo la participación de más de un gene de resistencia, destacaron los genes tetA y sul3 (Cuadro 5).
DISCUSIÓN
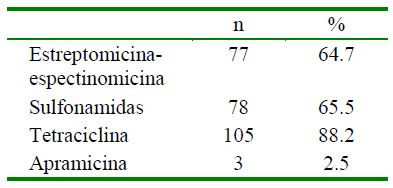
Los resultados obtenidos en este estudio muestran que de los 119 aislados de E. coli, 98.3% (117/119) presentaron al menos un gen de resistencia antimicrobiana, siendo las tetraciclinas el grupo de antibióticos con mayores casos de resistencia (88.2%). Esta alta frecuencia ha sido determinada en estudios previos; así, en Canadá, E. coli provenientes de cerdos mostraron 100% de resistencia en cepas de ETEC (Boerlin et al., 2005). Inclusive, estudios que abarcan varias especies de animales, concluyen que los porcinos presentan mayor frecuencia en la resistencia antimicrobiana (Lanz et al., 2003; Lim et al., 2007). En el Perú, Saldarriaga et al. (2000) y Monterroso et al. (2019) encontraron que el 97% de las 340 aislados y 100% de 36 aislados de E. coli, respectivamente, presentaban resistencia por lo menos a un antibiótico, resultados similares al presente estudio donde 98.3% (117/119) de los aislados fue resistente a por lo menos un antibiótico. Estos hallazgos muestran la alta prevalencia de resistencia antimicrobiana en las granjas porcinas tecnificadas a distintos antimicrobianos.
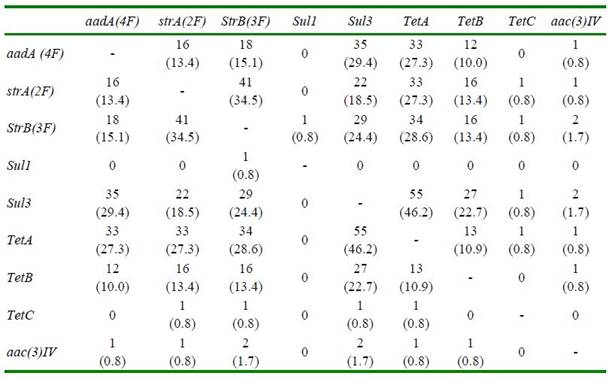
Cuadro 5. Frecuencia a dos de genes de resistencia antimicrobiana en 119 aislados de E. coli obtenidos de cuadros diarreicos en cerdos
Los aislados de E. coli evaluados presentaron mayor resistencia contra las tetraciclinas (88.2%), seguido de las sulfonamidas (65.5%) y estreptomicina- espectinomicina (64.7%). La alta resistencia a las tetraciclinas, antimicrobiano de mayor frecuencia de uso en las granjas porcinas, también ha sido descrita por Boerlin et al. (2005), encontrando 100% de frecuencia en cepas ETEC y por Lim et al. (2007), donde el 96.3% de aislados de E. coli de cerdos aparentemente sanos fueron resistentes a las tetraciclinas. Esto podría explicarse debido al uso de las tetraciclinas como promotores de crecimientos, administrados en subdosis, lo cual permitiría una selección de bacterias resistentes que podrían tener el rol de reservorio de genes de resistencia para otras bacterias comensales y patógenas (Chopra y Roberts, 2001).
El gene tetA fue el de mayor frecuencia de resistencia antimicrobiana, resultado que concuerda con el hallazgo de 92% de frecuencia por Boerlin et al. (2005), donde no solo es el gen predominante en el grupo de las tetraciclinas, sino entre todos los genes de resistencia antimicrobiana evaluados. Resultados similares han sido reportados en otros estudios (Lanz et al., 2003; Schwaiger et al., 2010; Sheikh et al., 2012). Sin embargo, Maynard et al. (2003), encontraron una baja frecuencia del gen tetA (25%) comparado con el gen tetB (80%) en muestras recolectadas entre 1978 al 2000. Estudios iniciales como el Sunde et al. (1998) posicionaron a los genes tetB y tetC como predominantes en la resistencia contra tetraciclinas.
El segundo gen de resistencia antimi- crobiana con mayor frecuencia fue sul3 (64.7%), a diferencia de otros estudios don- de el gen predominante en las sulfonamidas corresponde al gen sul1 (Boerlin et al., 2005; Brand et al., 2017; Mazurek et al., 2018); sin embargo, Sheikh et al. (2012), obtuvieron la misma frecuencia de resistencia en los genes sul1 y sul3 (9.8%) en muestras procedentes de carne de cerdo. Otros autores (Lanz et al., 2003; Smith et al., 2010) no consideraron el gen sul3 en sus estudios, siendo el gen sul1 el predominante. Esto es debido a que recién en 2003 se descubrió el gen sul3 en la población porcina de Suiza (Perreten y Boerlin, 2003), demostrando la capacidad de adaptación bacteriana contra los antimicrobianos. Para el gen sul1 solo se obtuvo una muestra positiva; sin embargo, esta no fue corroborada con el secuenciamiento completo del genoma. Por otro lado, el gen sul2 no fue encontrado en este estudio, mientras que para Schwaiger et al. (2010) fue el gen predominante en el grupo de las sulfonamidas y el gen sul3 el de menor frecuencia.
Los genes strB, seguido del gen aadA, fueron los de mayor frecuencia del grupo de la estreptomicina-espectinomicina, resultado que difiere con otros estudios, donde el gen aadA es el más frecuente dentro del grupo de los aminoglucósidos en porcinos (Lanz et al., 2003; Boerlin et al., 2005; Smith et al., 2010; Sheikh et al., 2012; Mazurek et al., 2018). En el estudio realizado por Saldarriaga et al. (2000) en el país se encontró una resistencia fenotípica más alta hacia la estreptomicina (del 85.91%; 292/340), pese a que dicho antibiótico no fuera administra- do, lo cual supone una resistencia cruzada por el uso de espectinomicina en las marranas previo al parto, debido a su composición química que los asemeja a los aminoglicósidos (Werth, 2020). El gen aac(3)IV fue el de menor frecuencia, encontrándose solo en tres cepas, lo que concuerda con otros estudios (Kozak et al., 2008; Sheikh et al., 2012) don- de la frecuencia de este gen de resistencia fue mínima. Sin embargo, este gen es notablemente mayor en los resultados de Smith et al. (2010), siendo el segundo gen con más frecuencia en el grupo de aminoglucósidos, alcanzando incluso el 85% de aislamientos positivos. Esto puede deberse a la característica de las cepas usadas, pues los serogrupos en el estudio corresponden al patotipo ETEC y son multidrogo resistentes.
La asociación de genes de resistencia fue mayor entre tetA y sul3, dado que ambos fueron los genes con mayor frecuencia en este estudio; sin embargo, otros autores no muestran asociación entre estos (Boerlin et al., 2005; Schwaiger et al., 2010; Sheikh et al., 2012), probablemente debido a la baja frecuencia del gen sul3 en sus resultados. La segunda asociación más frecuente fue de los genes strA y strB, ambos del grupo de los aminoglucósidos, asociación que se ha visto presente en varios estudios (Boerlin et al., 2005; Schwaiger et al., 2010; Zhao y Dang, 2010), puesto que la interacción de ambos genes garantiza una mayor resistencia contra la estreptomicina (Sunde y Norström, 2005). Por otro lado, la asociación de dos genes de resistencia contra las tetraciclinas fue mínima, al igual que Schwaiger et al. (2010), donde el 9% de los aislados presentaban dos genes de resistencia contra las tetraciclinas, lo que podría deberse a una incompatibilidad de plásmidos que contienen genes de resistencia contra las tetraciclinas (Jones et al., 1992; Maynard et al., 2003).
CONCLUSIONES
Se determinó una alta presencia de genes de resistencia antimicrobiana (98.3%) en aislados de E. coli de cuadros diarreicos de cerdos, siendo los genes gen tetA (68%), sul3 (64.7%), strB (42%) y aadA (39.5%) los de mayor frecuencia.
La frecuencia más alta de resistencia antimicrobiana fue al grupo de las tetraciclinas (88.2%), seguido de las sulfonamidas (65.5%) y estreptomicina- espectinomicina (64.7%).












 uBio
uBio