INTRODUCCIÓN
El género Salmonella se caracteriza por tener bacilos Gram negativos que pertenecen a la familia Enterobacteriaceae. Son microorganismos anaerobios facultativos y tienen la capacidad de producir ácido sulfhídrico (H2S). Se conocen dos especies denominadas S. enterica y S. bongori, siendo de interés para la salud humana y animal S. enterica, la cual se subdivide en seis subespecies: S. enterica subsp enterica, S. enterica subsp salamae, S. enterica subsp arizonae, S. enterica subsp diarizonae, S. enterica subsp houtenae y S. enterica subsp indica (Kauffman, 1961).
La taxonomía de Salmonella es compleja debido a que cuenta con más de 2500 variantes serológicas según la clasificación del esquema de White-Kauffmann-Le Minor, la cual se basa en la presencia de antígenos somáticos (O) y flagelares (H) (Switt et al., 2009), de allí la dificultad de utilizar la técnica de diagnóstico más apropiada.
Los biovares específicos causantes de enfermedades en aves son S. Gallinarum que ocasiona la fiebre tifoidea aviar y S. Pullorum causante de pulorosis aviar (Barrow y Freitas Neto, 2011). Por otra parte, los serovares más importantes transmitidos de animales a seres humanos y asociados a enfermedades de transmisión alimentaria (ETA) son: Salmonella enterica serotipo Enteritidis y Salmonella enterica serotipo Typhimurium (OMS, 2018).
La enfermedad que ocasiona esta bacteria es la salmonelosis, definida como una patología infecciosa diarreica y sistémica, que afecta tanto humanos como animales, siendo de reporte obligatorio en algunos países. La notificación de casos y brotes permite dar un seguimiento al desarrollo de la enfermedad y así facilitar su control y prevención (OIE, 2018). En general, las entidades encargadas de la vigilancia y control de Salmonella sp recomiendan diferentes técnicas diagnósticas según las necesidades de cada laboratorio, el tamaño del muestreo, la procedencia y la precisión que se requiere para establecer un diagnóstico. El aislamiento microbiológico mediante cultivo es uno de los métodos más usados debido a su sensibilidad, especificidad, economía y sencillez. El protocolo incluye una etapa de pre-enriquecimiento, seguido de enriquecimientos selectivos, y finalmente se debe confirmar bioquímica y serológicamente con antisueros O y H; sin embargo, estos métodos tardan hasta cinco días para generar un presunto aislamiento positivo y dos días más en dar un resultado verdaderamente positivo con pruebas bioquímicas confirmatorias (OIE, 2018). Adicionalmente, los protocolos microbiológicos no identifican los serovares (Dos Santos et al., 2015).
Por otro lado, los métodos de diagnóstico han mejorado sustancialmente con el desarrollo de técnicas moleculares como la Reacción en Cadena de la Polimerasa (PCR) y la secuenciación, las cuales son rápidas, sensibles, específicas y logran identificar la mayoría de las serovariedades, pero requieren equipos de mayor costo y operadores especializados que limitan su aplicación en entornos con recursos reducidos (Liu et al., 2019). Otros métodos para el diagnóstico de Salmonella sp son los enzimoinmunoanálisis (ELISA), la separación inmunomagnética (IMS), MALDI- TOF (Matrix-Assisted Laser Desorption/Ionization-Time of Flight) o ionización-desorción asistida por matriz con tiempo de vuelo, y el análisis por microchip, entre otros (OIE, 2018).
Según lo expuesto, es necesario recopilar información sobre las técnicas que se encuentran disponibles para el diagnóstico de Salmonella sp, que identifiquen verdaderos positivos y verdaderos negativos, y que se acomoden a los recursos disponibles y a las necesidades de cada laboratorio, dependiendo del tipo de muestra y otras variables que influyen en el diagnóstico. El objetivo de esta revisión sistemática fue proporcionar información sobre las técnicas microbiológicas y moleculares para la identificación de Salmonella sp que son utilizadas a nivel mundial, para ofrecer alternativas a los laboratorios de diagnóstico para optimizar resultados y así aportar a la toma de decisiones en campo.
MATERIALES Y MÉTODOS
Se llevó a cabo una búsqueda sistemática de artículos y trabajos de tesis sobre la identificación y técnicas diagnósticas de Salmonella sp a nivel global y publicados desde enero de 2015 hasta diciembre de 2021. La revisión incluyó técnicas moleculares y de secuenciación del genoma, pruebas serológicas y técnicas de cultivo tradicionales que abarcan la batería bioquímica y los cultivos selectivos. Los artículos se obtuvieron de seis bases de datos: NCBI/PubMed (http://www.ncbi.nlm.nih.gov/PubMed), ScienceDirect (https://www.sciencedirect.com/), -Scopus (https://www.scopus.com/home.uri?edit.scft=1), SciELO - Scientific Electronic Library Online (http://www.scielo.org/), Google Scholar (https://scholar.google.com/) y Springer (https://www.-springer.com/la).
En las búsquedas se excluyeron reseñas de libros, capítulos de libros, resúmenes de conferencias, enciclopedias, correspondencia, informes de patentes, comunicaciones cortas, ensayos clínicos, artículos con reportes de Salmonella sp desarrollada en humanos o resistencia antimicrobiana en especies de aves que no fueran galliformes.
El proceso de búsqueda y selección se realizó haciendo uso de palabras clave en la investigación y de conectores booleanos para construir ecuaciones de búsqueda, abarcando los criterios de inclusión. Algunos ejemplos de las ecuaciones de búsqueda fueron: ((poultry industry) AND (invA gene)) AND (PCR) AND (avian) AND (salmonella) AND (molecular detection); (((Poultry farm) AND (broiler)) AND (avian salmonella)) AND (serotyping))) NOT (clinical). Para la selección e inclusión de los artículos que aportaron a la revisión sistemática se utilizó la metodología PRISMA (http://www.prismastatement.org).
Dado el impacto de Salmonella sp en la industria avícola y considerando el objetivo de esta revisión de alcance se abordaron las siguientes preguntas:
- ¿Cuáles son las técnicas microbiológicas y moleculares para la identificación de Salmonella sp en la industria avícola?
- ¿Cuál es la técnica más utilizada para la detección de salmonelosis aviar según los artículos que se encontraron en las diferentes bases de datos?
- ¿Qué protocolos de microbiología se han empleado en la identificación de Salmonella sp?
- ¿Cuáles genes se proponen para la identificación de Salmonella sp en aves en la identificación molecular?
- ¿Cuál es el origen de las muestras de los artículos revisados?
- ¿Cuál es la importancia del número de muestras analizadas en las diferentes técnicas de los artículos reportados?
RESULTADOS
Se identificaron 860 documentos en las seis bases de datos revisadas. De estos, 1.4% (n=12) fueron duplicados y se excluyeron dentro de la primera fase de identificación. Se revisó por título y Resumen/Abstract el 98.6% (848) restante. Se eliminó el 91.6% (n=777) según los criterios de exclusión iniciales, quedando 71 documentos (8.4%). Luego de revisarlos, 10 documentos tenían datos insuficientes, eran artículos o informes en aves que no describen el trabajo en la especie G. gallus o no eran relevantes en técnicas diagnósticas, de modo que solo 61 documentos cumplieron con los criterios de inclusión establecidos para este estudio. La Figura 1 muestra los detalles del proceso de búsqueda en las bases de datos.

Figura 1 Diagrama del flujo PRISMA (Preferred Reporting Items for Systematic Reviews and Meta-Analyses - Elementos preferenciales para informar sobre revisiones sistemáticas y metanálisis) que describe las etapas de identificación, cribado, elegibilidad e inclusión de los artículos selecciones en el estudio. Adaptado de Page et al. (2021)
El análisis de los artículos incluidos y revisados mostró que el 24% (n=15) fue publicado en 2019, seguido de 2017 (20%, n=12) y 2018 (18%, n=11). Asimismo, la mayor parte de las publicaciones se hizo en revistas del área de veterinaria con 42.6% (n=26), seguido del área de microbiología con 22.9% (n=14). En veterinaria, las investigaciones estuvieron dirigidas a la identificación y diagnóstico de Salmonella sp en la industria avícola (gallinas y pollos), enfocada en el control oportuno de las posibles enfermedades causadas por especies de Salmonella en las aves o que desencadenan enfermedad grave en el humano por la transmisión alimentaria.
Del total de artículos, el 41% fue realizado en Asia, 38% en las Américas, 18% en África y 3% en Europa, no habiéndose registrado estudios en Oceanía. En el continente asiático, el país que predominó fue China con 11 artículos. Asimismo, a nivel de Latinoamérica se obtuvieron 17 artículos de interés para el presente estudio.
Con base en los protocolos de microbiología encontrados en los 61 artículos revisados, 19.6% (n=12) realiza pre-enriquecimiento en agua peptonada incubando a 37 °C durante 18-24 h cuando las muestras provienen de un hisopo, y enriquecimiento selectivo en caldos de tetrationato (TT) y RappaportVassiliadis (RV). Los medios líquidos se incuban durante 24 h a 42 °C. RV es un medio selectivo enriquecido con verde malaquita que inhibe el crecimiento de microorganismos distintos de Salmonella ( Saravanan et al., 2015; Alegria-Moran et al., 2017; Reda et al., 2019; Obe et al., 2020).
Salmonella sp se puede aislar en medios selectivos y diferenciales como el agar xilosa lisina desoxicolato (XLD), agar Salmonella Shigella (SSA), agar Hektoen Entérico y agar MacConkey (Moraes et al., 2016; De Carli et al., 2017; Pereira et al., 2018; Khaltabadi et al., 2019; Mthembu et al., 2019; Zanetti et al., 2019). El 37.7% (n=23) de las publicaciones hicieron uso de los medios selectivos y diferenciales ya mencionados para la identificación de las bacterias, aunque algunos autores emplearon estrictamente la metodología tradicional (8.19%, m=5) (Clemente et al., 2015; Vaddella et al., 2016; Kumar et al., 2019), o combinaron la microbiología con técnicas moleculares (42.6%, n=26) (Soria et al., 2017; Wajid et al., 2019; Shehata et al., 2020).
Asimismo, en 18% (n= 11) de los artículos se hizo uso de una batería bioquímica; lo cual ayuda a confirmar si las presuntas colonias son de Salmonella sp por medio de las reacciones metabólicas que presenta la bacteria en los diferentes medios. En estos casos se hizo uso de agares como los citados a continuación:
- Triple Azúcar Hierro (TSI): indica fermentación de glucosa y no fermentación de lactosa y/o agarosa; puede presentar o no fondo negro, indicando producción de H2S (Tarabees et al., 2017; Zanetti et al., 2019).
- Agar Lisina Hierro (LIA): indica fermentación de glucosa y descarboxilación de lisina (Tarabees et al., 2017).
- Agar Urea: determina si la bacteria hidroliza la urea (De Carli et al., 2017; Alzwghaibi et al., 2018).
- Agar Simons citrato: indica otra vía de obtención de energía, usando carbono como única fuente de energía (Saravanan et al., 2015; Alegria-Moran et al., 2017; Alzwghaibi et al., 2018).
- Caldo triptófano: producción de indol (Saravanan et al., 2015; Yokoyama et al., 2015). - Medio SIM: sulfuro de hidrógeno, formación de indol y motilidad (DonadoGodoy et al., 2015; Yokoyama et al., 2015).
- Caldos de fermentación para carbohidratos: maltosa, lactosa y sacarosa (Saravanan et al., 2015).
El perfil bioquímico para los serovares de mayor importancia en el área avícola se describe en el Cuadro 1.
Cuadro 1 Perfil bioquímico de los serovares Enteritidis, Typhimurium y Gallinarum con sus biovares Gallinarum y Pullorum, que ayuda a identificar diferencias como motilidad o producción de H2S entre serovares
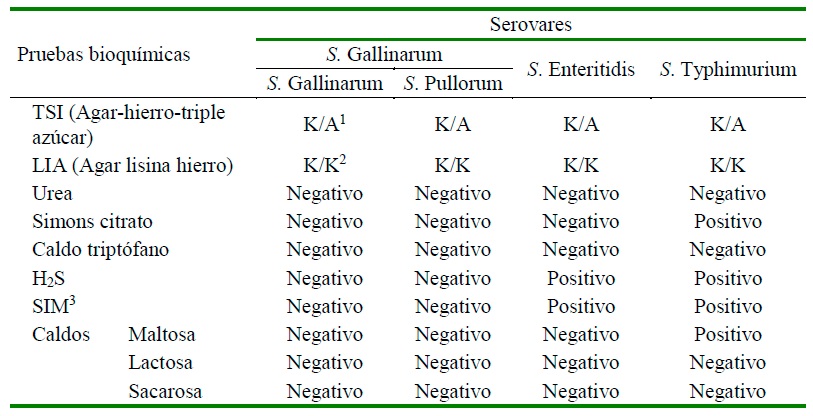
1 K/A: Superficie alcalina/profundidad ácida: Microorganismo que solamente fermenta la glucosa
2 K/K: Indica decarboxilación de la lisina, por ende, es un resultado positivo
1 SIM: producción de sulfuro de hidrógeno (H2S), formación de indol y motilidad
La prueba gold standard para la identificación de serovares de Salmonella sp es la serología, por medio de la metodología White-Kauffmann-Le Minor, que consta de una gama de antisueros que detectan los antígenos específicos de cada serovar de Salmonella sp (Alzwghaibi et al., 2018; Xiong et al., 2018; Sedeik et al., 2019).
En los artículos que se incluyeron en la revisión sistemática, el 27.8% (n=17) emplearon la metodología White-Kauffmann-Le Minor para serotipificar, los artículos que emplearon serotipificación identificaron dos serovares: S. Enteritidis y S. Thyphimurium (Tarabees et al., 2017), mientras que los trabajos que combinaron serotipificación por White-Kauffmann-Le Minor con alguna técnica molecular (9.83%, n=6) caracterizaron serovares como S. Enteritidis, S. Typhimurium, S. Panama, S. Lexington y S. Rissen (Machado et al., 2017; Rodríguez et al., 2015b; Zhao et al., 2016). La Figura 2 muestra las publicaciones que se apoyaron en más de un tipo de técnica para identificar y caracterizar Salmonella sp.
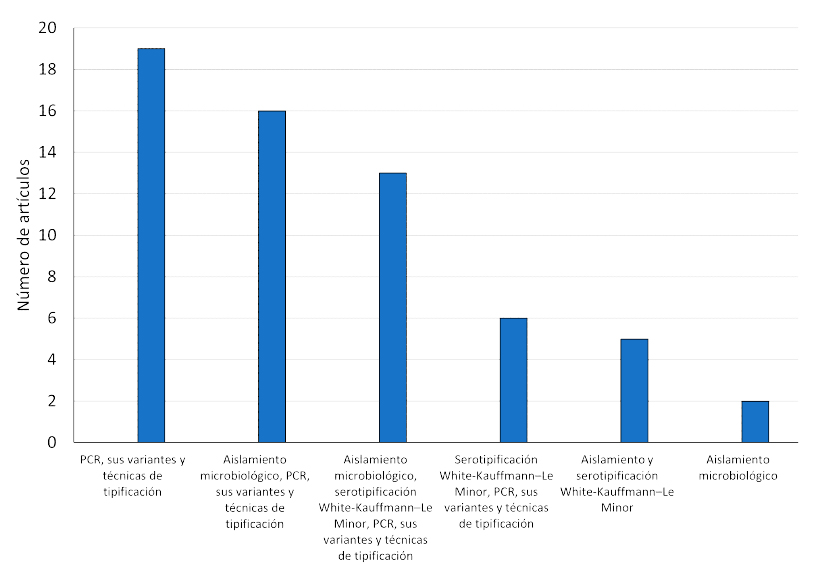
Figura 2 Técnicas de identificación y caracterización para Salmonella sp reportadas en 61 artículos científicos publicados entre 2015 y 2021
En el 88.5% (n=54) de los artículos emplearon herramientas moleculares, siendo PCR la técnica más empleada (72.1%). Algunos autores utilizaron variaciones de la PCR como la PCR multiplex (Ibrahim et al., 2016; Xiong et al., 2018), PCR-tiempo real (qPCR) (Heymans et al. 2018) y Consenso Intergénico Repetitivo de Enterobacterias - PCR (ERIC-PCR Enterobacterial Repetitive Intergenic Consensus - PCR) (Zhao et al., 2016) con 13.1, 8.1 y 1.6%, respectivamente. Para identificar y caracterizar los serovares de Salmonella sp se usó una amplia gama de genes específicos como el gen invA (Abd-Elghany et al., 2015; Wajid et al., 2019), sepC y glgC para biovares como S. Gallinarum y S. Pullorum (Zanetti et al., 2019) (Ver Cuadro 2).
Cuadro 2 Genes y cebadores reportados en artículos (n) por diversos autores para la identificación de Salmonella sp

NE: No especifica
Como alternativa de la PCR se llevaron a cabo técnicas como amplificación isotérmica mediada por Loop (LAMP - LoopMediated Isothermal Amplification) (Liu et al., 2019) y Amplificación por recombinasa y polimerasa (RPA - Recombinase Polymerase Amplification) (Liu et al., 2017). Las técnicas de caracterización reportadas fueron la tipificación multilocus de secuencias (MLST - Multi-Locus Sequence Typing) (Wang et al., 2020), ribotipificación (ISR - dkgB-linked intergenic sequence ribotyping) (De Carli et al., 2017) y el análisis filogenético (Yokoyama et al., 2015). También se empleó la electroforesis en campo pulsado (PFGE - Pulsed Field Gel Electrophoresis) (Saravanan et al., 2015; Machado et al., 2017; Ha et al., 2018) con el fin de rastrear los serovares responsables de los brotes, diferenciar entre serovares patógenos y establecer la relación genética de los serovares para construir la filogenia (Wang et al., 2020).
Con las herramientas moleculares, especialmente con la PCR y sus variantes y con las técnicas de tipificación multilocus como MLST, PFGE e ISR se identificaron serovares como S. Typhimurium, S. Heidelberg, S. Bongori, S. Enteritidis, S. Paratyphi y S. Newport (Ramatla et al., 2020), S. Kentucky (Tasmin et al. 2017), S. Schwarzengrund (Moraes et al. 2016), S. Hvittingfoss y S. Muenster (Rodríguez et al., 2015a), S. Pullorum y S. Gallinarum (Ren et al., 2017). El serovar más frecuente identificado con herramientas moleculares fue S. Typhimurium (47.5%, n=29) seguido de S. Enteritidis (39.3%, n=24).
El tipo de producción incluido en esta investigación abarcó pollos de engorde (21.3%, n=13), ponedoras (8.2, n=5) y reproductoras (8.2%, n=5), mientras que en el 62.3% (n=38) de los estudios no especificaron el tipo de ave. El serovar más frecuente en pollos de engorde fue Enteritidis (muestras fecales) (Zhao et al., 2016), y Typhimurium y Kentucky en canales de pollos de engorde (Tasmin et al., 2017). Por otra parte, los serovares encontrados con mayor frecuencia en las aves ponedoras fueron Schwarzengrund y Enteritidis aislados de la cáscara del huevo, albúmina y la yema (Moraes et al., 2016). Asimismo, Im et al. (2015) encontraron serovares menos comunes en heces, polvo y cáscaras de huevo de aves ponedoras (S. Bareilly, S. Mbandaka, S. Rissen). En forma similar, en muestras de reproductoras se encontraron los serovares S. Gallinarum, S. Typhimurium y S. Enteritidis que fueron aislados de huevo y de hisopado cloacal (Gómez et al., 2015).
El tipo de muestra para la identificación de Salmonella sp fue muy variable. Entre las muestras de tejidos (n=9) se utilizó preferentemente el huevo (cáscara, albúmina y yema) y en las muestras ambientales (n=11) (Saravanan et al., 2015; Wajid et al., 2019) se utilizó mayormente el hisopado de arrastre. En las muestras alimentarias se utilizaron partes de aves como alas, pechugas y muslos (Park y Ricke, 2015; Abd-Elghany et al., 2015; Tarabees et al., 2017; Machado et al., 2020). Por otro lado, en 10 estudios (Kumar et al., 2019; Zanetti et al., 2019; Machado et al., 2020) se empleó directamente aislamientos bacterianos para trabajar con serotipificación, PCR y ELISA. En 20 estudios se trabajó con vísceras como intestino, hígado, riñón, saco vitelino y bazo (Gómez et al., 2015; Zhao et al., 2016).
Un punto importante de analizar es el número de muestras utilizadas en los estudios. De los 61 artículos, 75.4% (n=46) tomaron 100 o más muestras, mientras que 24.5% (n=15) tomaron menos de 100 muestras o no especificaron el tamaño de la muestra.
Posterior a la revisión y análisis de las técnicas reportadas, se elaboró una guía de identificación y caracterización para Salmonella sp. Se partió del tipo de muestras que se pueden tomar en avicultura: a) muestras ambientales (hisopos de arrastre, muestras de polvo, ropa de trabajadores, muestras directas de galpones, reservas de agua y alimento para los pollos); b) hisopados cloacales y heces secas y c) muestras de órganos (huevos o vísceras) (Machado et al., 2017). Se debe considerar que, en las muestras ambientales, de huevos o de heces solo se puede recuperar salmonellas no tifoideas, mientras que en las muestras obtenidas de órganos post mortem se recuperan las salmonellas tifoideas (S. Gallinarum y S. Pullorum). A partir de las muestras ambientales e hisopados cloacales se realiza un preenriquecimiento no selectivo en agua peptonada a 37 °C durante 18-20 h, luego se realiza un enriquecimiento selectivo en caldo o agar semisólido de TT y RV a 42 °C durante 18-24 h (Saravanan et al., 2015), y se continua con la etapa de aislamiento en medios selectivos y diferenciales, observando las características bioquímicas de las cepas aisladas. Entre los medios más utilizados se encuentra MacConkey, Hektoen Entérico, Salmonella Shigella y XLD, que se incuban a 37 °C durante 18-24 h (Soria et al., 2017)
Para confirmar las presuntas colonias de Salmonella sp se tienen dos protocolos. El primero consiste en la metodología tradicional, realizando la batería bioquímica básica, seguido de la serotipificación con el esquema White-Kauffmann-Le Minor. La segunda metodología consiste en aplicar herramientas moleculares como la PCR. Para esto último es preferible tener células bacterianas viables en un medio de cultivo líquido como el Luria Bertani (LB) para facilitar la extracción de ADN, dado que no contiene inhibidores de la PCR. Los cebadores específicos que se requieren en la PCR permiten determinar algunos de los serovares de Salmonella sp, al igual que las técnicas de tipificación molecular como MLST, ISR o PFGE (Ver Figura 3)
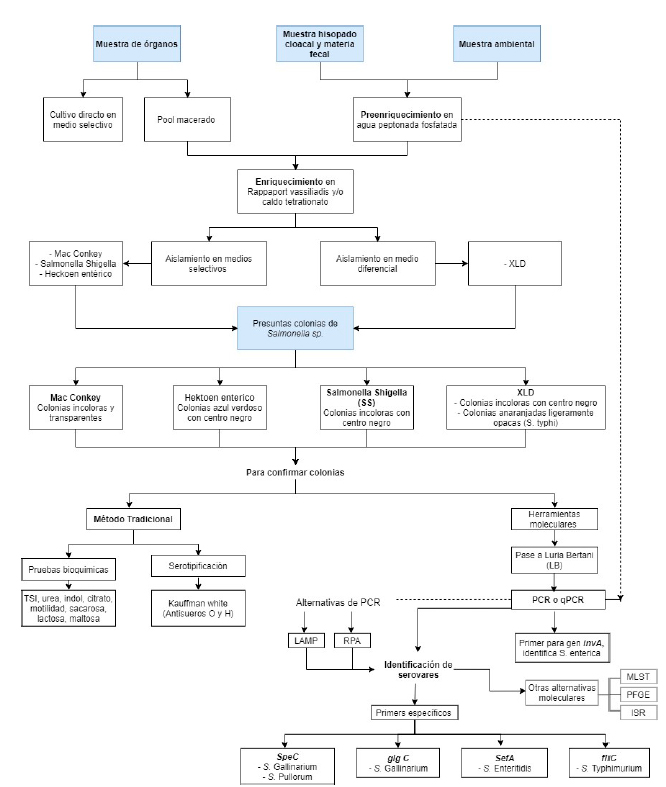
Figura 3 Protocolo de identificación y caracterización para Salmonella sp a partir de muestras ambientales, hisopados cloacales, heces y órganos de aves (Gallus gallus) obtenidas en granjas avícolas. LAMP: Loop-Mediated Isothermal Amplification; RPA: Recombinase Polymerase Amplification; MLST: Multi-Locus Sequence Typing; PFGE: Pulsed Field Gel Electrophoresis; ISR: dkgB-linked intergenic sequence ribotyping
DISCUSIÓN
Las técnicas tradicionales basadas en la microbiología y serotipificación prevalecen como el gold standard debido a su practicidad, bajo costo y sensibilidad (Moraes et al., 2016). Además, se reconoce que la mayoría de los laboratorios prefieren realizar pre-enriquecimiento, enriquecimiento y aislamiento antes de pasar a técnicas moleculares como la PCR (Ramatla et al., 2020). El pre-enriquecimiento permite restaurar las bacterias dañadas, logrando de esta manera una condición fisiológica estable (Wajid et al., 2019) y el enriquecimiento aporta en la selectividad creciente del patógeno. Cabe aclarar que dentro de las técnicas microbiológicas, los medios selectivos y diferenciales son de gran ayuda para la identificación de colonias, pero requieren tiempo, ya que tardan hasta cinco días en dar un resultado (Ibrahim et al., 2016). Por otro lado, la extracción del ADN para su uso en PCR puede ser complicado cuando se usan medios de cultivo que contienen inhibidores como sales biliares y rojo de fenol en el caso del medio XLD, por lo que debe hacerse un pase intermedio a medios que no contengan inhibidores de la PCR antes de proceder a la extracción del ADN. Aun así, los medios selectivos más utilizados en las publicaciones revisadas fueron XLD y MacConkey (Donado-Godoy et al., 2015; Sedeik et al., 2019; Wang et al., 2020) para la identificación de Salmonella sp.
La metodología White-Kauffmann-Le Minor es sensible para subtipificar los diferentes aislamientos según su serovariedad, habiéndose descrito cerca de 2600 serotipos con este esquema (Lozano-Villegas et al., 2019). Esto ha sido posible gracias a la identificación de antígenos somáticos (O) y flagelares (H) presentes en la pared celular de Salmonella sp (Alzwghaibi et al., 2018). No obstante, la serotipificación es un proceso costoso y laborioso, que no identifica el serovar con la fase flagelar como S. enterica serovar Gallinarum y no distingue entre las serovariedades de S. Gallinarum y S. Pullorum (Xiong et al. 2018). Los anterior es un problema para la industria avícola ya que estos biovares son los causantes de la fiebre tifoidea aviar y la pulorosis aviar.
En la presente revisión, técnicas moleculares como PCR multiplex y PCR convencional fueron las más utilizadas. La PCR multiplex es una herramienta prometedora para el diagnóstico clínico al tener una alta sensibilidad y especificidad en los resultados (Xin et al., 2021). También, amplifica secuencias específicas y discrimina en forma simultánea a microorganismos, cepas y serotipos (Mthembu et al., 2019). En contraparte, tiene la desventaja de requerir una gran cantidad de cebadores, lo cual aumenta el riesgo de un alineamiento incorrecto (Li et al., 2017). La PCR convencional amplifica solo una región del ADN, limitando su uso (Rodríguez Hernández et al., 2021). El poder discriminatorio de esta técnica está basado en el tamaño del cebador y su especificidad depende de los cebadores empleados (Soria et al., 2017).
Otra limitación que presenta el PCR multiplex y PCR convencional es el requerimiento de procesarla luego de una PCR para visualizar los productos por medio de electroforesis (Ha et al. 2018). Por otro lado, dada la elevada sensibilidad y especificidad de la qPCR, se puede identificar Salmonella sp en muestras con escasa cantidad de ADN (Park y Ricke, 2015). logrando amplificar y detectar los productos en una misma etapa (Moraes et al., 2016). Aun así, una limitación de esta técnica es el uso de tres dianas (dos cebadores y las sondas) que sean conservadas, ya que una mutación en cualquier segmento impedirá la detección de alguna variante (Nova et al., 2017).
Otras técnicas reportadas basadas en los principios de la biología molecular para realizar la caracterización de Salmonella sp fueron: ERIC-PCR, técnica económica y sencilla, que permite tipificar y discriminar serotipos estrechamente relacionados (Sedeik et al., 2019), así como las técnicas de amplificación isotérmica como RPA y LAMP, las cuales no requieren de extracción de ADN y presentan alta sensibilidad, robustez, rapidez y automatización (Liu et al., 2019). De igual manera, ISR y MLST son empleadas para tipificar aislados de Salmonella sp permitiendo el seguimiento filogenético (Guard et al., 2012; Wang et al., 2020), además de PFGE que es una técnica discriminatoria y reproducible, útil en los estudios de seguimiento epidemiológico para identificar serovares o cepas estrechamente relacionadas (VossRech et al., 2015). No obstante, estas técnicas fueron muy poco utilizadas en las publicaciones revisadas, ya que los autores consideran como limitantes la falta de protocolos estandarizados y la capacitación del personal (Li et al., 2017), además de los costos de los equipos (Kubo et al., 2020).
En los 61 artículos revisados, el gen invA (Abd-Elghany et al., 2015) fue el más utilizado para la identificación de S. enterica, ya que es específico de este género, aunque no permite la discriminación de serovares. Asimismo, en la revisión se detectaron dos genes utilizados para identificar Salmonella sp, que van más dirigidos a sus mecanismos de virulencia: hilA (Shang et al., 2019) y spv (Vidic et al., 2017). Los otros genes que se destacaron en la identificación de serovares específicos de S. enterica fueron speC para S. Gallinarum y S. Pullorum (Alzwghaibi et al., 2018), glgC para S. Gallinarum (De Carli et al., 2017), fliC que codifica la flagelina de S. Typhimurium (Khaltabadi et al., 2019) y sefA que codifica el antígeno fimbrial de S. Enteritidis (Alzwghaibi et al., 2018).
A pesar de que el gold standard para la identificación y caracterización de Salmonella sp en la industria avícola sigue siendo la microbiología y la serotipificación, la mayoría de los países ha implementado PCR y sus variantes. Sin embargo, implementar herramientas moleculares de rutina en los laboratorios es un reto para algunos países, ya que a pesar del bajo costo que tiene procesar las muestras con biología molecular, equipar un laboratorio con los instrumentos necesarios para llevar a cabo estas nuevas tecnologías es costoso. Adicionalmente, lograr estandarizar un ensayo en la mayoría de los laboratorios toma tiempo y trabajo.
CONCLUSIONES
Es esencial que en la industria avícola existan técnicas capaces de identificar los serovares de Salmonella sp causantes de enfermedad en aves de corral y en humanos.
La serotipificación por White-Kauffmann-Le Minor es el gold standard para caracterizar las serovarie-dades de Salmonella sp; sin embargo, esta técnica puede ser costosa y laboriosa, de allí que las técnicas moleculares permiten caracterizar los serovares de Salmonella sp de una forma más sencilla y con mayor sensibilidad.
Las técnicas moleculares más empleadas por los autores son PCR convencional y PCR multiplex, donde la especificidad de los cebadores permite identificar y caracterizar genes específicos de los serovares de Salmonella sp, obteniendo resultados más sensibles, específicos y rápidos.
La PCR y PCR multiplex se pueden realizar a partir de resuspensión de células bacterianas viables de aislamientos en medios selectivos, de allí que se sigue requiriendo el uso de técnicas microbiológicas.
Las herramientas moleculares presentan una gran ventaja frente a los métodos tradicionales, ya que estas técnicas de identificación genotípica permiten diferenciar serovares fenotípicamente similares.












 uBio
uBio 


