INTRODUCCIÓN
La cría de rumiantes menores crece constantemente a nivel mundial (FAO, 2017). Sin embargo, en Colombia esta actividad aún muestra un bajo desarrollo, debido entre otras razones al desconocimiento de los parámetros productivos de los ovinos por la limitada generación de registros de datos, la baja disponibilidad de unidades productivas tecnificadas (Parra et al., 2014), o simplemente por la resistencia de los productores al cambio.
La población ovina en Colombia se concentra principalmente en la Región Caribe con 1 339 480 ovinos correspondientes al 74.2% del inventario nacional (ICA, 2022). El sistema de producción de ovinos en esta región es predominantemente extensivo, de gran aporte a la agricultura familiar y manejado generalmente por pequeños productores de bajo acceso tecnológico, por lo que se ha considerado tradicionalmente como una "economía de subsistencia" (Bustamante et al., 2022). De hecho, Mestra-Vargas et al. (2019) identificaron que un ejercicio de manejo técnico y seguimiento de información permitiría alcanzar un sistema ovino eficiente y rentable. No obstante, la variabilidad ambiental juega un papel preponderante en la Región Caribe y, por tanto, el seguimiento productivo favorecería el reconocimiento de las características genéticas de cada población o raza ovina para su sostenibilidad.
A nivel de campo se observa una alta heterogeneidad fenotípica y posiblemente presencia de altos niveles de consanguinidad en las poblaciones, debido al interés de los productores por mantener núcleos de animales foráneos con fenotipos raciales asociados a Dorper, Katahdin, Santa Inés, Sudán y Pelibuey, entre otros (Leymaster, 2002). Esto puede ser contraproducente puesto que, la presencia de fenotipos "aparentes", la alta heterogeneidad y consanguinidad pueden llevar a una disminución drástica de los parámetros productivos, disminución de la alzada, menores pesos y mayor susceptibilidad a parásitos y enfermedades (Wilson et al., 2022).
La ausencia de una estructura de recolección de información, del manejo de registros de carácter productivo y reproductivo, además de la falta de caracterizaciones de tipo molecular y morfológico de la especie, han llevado a la imposibilidad de estructurar algún tipo de estrategia de manejo genético que permita mejorar la eficiencia productiva de los ovinos y, con ello, generar alternativas económicas para proteger la seguridad alimentaria del país. Con esta problemática, el presente estudio tuvo como objetivo estimar la diversidad genética y la estructura poblacional en las poblaciones ovinas de pelo de la Región Caribe colombiana.
MATERIALES Y MÉTODOS
Animales y Muestreo
Mediante conversaciones con representantes de asociaciones de productores se identificaron municipios que permitieran generar una representación de la diversidad de las poblaciones ovinas de pelo dentro de la región geográfica del Caribe colombiano. Posteriormente, se realizaron visitas técnicas donde se evaluaron fenotípicamente los animales y se definieron a partir de sus rasgos y la comunicación con el criador, la raza o posible grupo poblacional al que pertenecían. No se contaba con un número determinado de grupos poblacionales o de animales dentro de grupo, dado que se buscó identificar la diversidad genética de los ovinos en los departamentos visitados. Finalmente, se seleccionaron 459 animales en 26 granjas distribuidas en 19 municipios ubicados en los departamentos de Córdoba, Sucre, Bolívar, Cesar, La Guajira y Magdalena. Se colectaron entre 10 y 30 muestras por aprisco y se consideró la relación de parentesco entre individuos a fin de reducir los sesgos por representación de marcadores idénticos por descendencia.
A cada individuo se recolectó una muestra de sangre en tubos con EDTA con anticoagulante que fueron remitidas al Laboratorio de Genética Molecular del Centro de Investigación Tibaitatá de AGROSAVIA para los análisis moleculares. Las 459 muestras de sangre recolectadas correspondieron a los siguientes grupos poblacionales: Criollo (n=302, 65.8%), Mestizo (n=72, 15.7%), Pelibuey (n=34, 7.4%), Santa Inés (n=32, 7.0%), Dorper (n=12, 2.6%) y Katahdin (n=7, 1.5%)
Extracción y Cuantificación de ADN
Los procesos de extracción y cuantificación del ADN, al igual que el genotipado, se realizaron en el laboratorio de Genética Molecular del Centro de Investigación Tibaitatá de AGROSAVIA (Mosquera, Colombia).
La extracción de ADN se realizó a partir de 300 µl de sangre utilizando el kit comercial UltraClean(r) Blood DNA Isolation Kit de Mo-BIO (Cat. 12000-100) acorde al procedimiento descrito por el fabricante. Las muestras de ADN fueron cuantificadas por el método de espectrofotometría con el equipo Nanodrop 2000 (Thermo Scientific). Las muestras de ADN con menor concentración a 100 ng/µl y calidad 1.8-2.0 en las relaciones de absorbancia 260/280 y 260/230, fueron concentradas en el equipo Speedvac (Vacufuge plus, Eppendorf) y diluidas a una concentración de 100 ng/µl, con agua grado molecular.
El genotipado se realizó con el chip Ovine LD 15K (Illumina. https://www.illumina.com/), obteniéndose los genotipos de 15 000 Polimorfismos de Nucleótido Simple (SNPs) distribuidos en el genoma. Los chips fueron escaneados en el equipo HiScan de Illumina a través del módulo IScan, y la lectura se realizó con el programa Hiseq Control v. 1.5.15.1. La calidad de la lectura fue evaluada en el programa Genome Studio v2011.1 (Illumina. https://www.illumina.com/).
Control de Calidad de la Información
Se incluyeron SNPs asignados a los cromosomas autosómicos a partir de información de posición de los marcadores en el genoma ovino (https://www.ensembl.org/Ovis_aries/Info/Annotation). Se eliminaron marcadores monomórficos y aquellos que presentaron una misma ubicación genómica. De este modo, la base de datos empleada en el análisis de diversidad y estructura poblacional incluyó 10 929 SNP en 459 muestras.
Adicionalmente, se incluyeron 23 muestras de la raza Wiltshire (Kijas et al., 2012) como grupo control (outgroup), para un total de 482 muestras de ovinos. Con la información agrupada se estimó: 1) Porcentaje de marcadores genotipados en más del 95% de las muestras; 2) Porcentaje de marcadores con una frecuencia de alelos menores (MAF) >0.05; 3) Heterocigosidad esperada; 4) Porcentaje de loci en el equilibrio de Hardy-Weinberg (p>0.01) utilizando una prueba exacta de equilibrio de Hardy-Weinberg (Wigginton et al., 2005). Finalmente, se descartó un animal del grupo poblacional de Criollo debido a bajo call rate y se eliminaron los SNPs que presentaron más de 5% de datos perdidos, dejándose para los análisis 10 908 SNP en 481 muestras. Todos los controles de calidad y depuración de datos se realizaron con el software PLINK v. 1.9 (Purcell et al., 2007).
Varianza Molecular y Divergencia entre Poblaciones
Los niveles de variación genética dentro y entre poblaciones se estimaron mediante un análisis de varianza molecular (AMOVA; Excoffier et al., 1992), con el softwareArlequin v. 3.5 (Excoffier & Lischer, 2010). La significancia de los componentes de varianza se estimó mediante 1000 permutaciones no paramétricas.
Para evaluar la divergencia entre los grupos poblacionales se calcularon dos medidas de distancia utilizando las frecuencias alélicas de las poblaciones. En primer lugar, los índices de fijación de diferenciación genética entre poblaciones (FST) se calcularon a través del software Arlequin v. 3.5 (Excoffier y Lischer, 2010), utilizando 20 000 permutaciones y con un nivel de significación de 0.05. A continuación, se estimaron las distancias genéticas D de Nei (Nei, 1972) entre todos los pares de grupos poblacionales y se construyó un dendrograma con el método de agrupamiento neighbor joining (NJ) (Saitou y Nei, 1987) con ayuda de los paquetes seqboot, gendist del software PHYLIP (Felsenstein, 2005). Para evaluar la consistencia en las relaciones de las poblaciones a través del dendrograma, se realizó un bootstrap de 1000 repeticiones. El dendrograma se trazó en un entorno R (R Development Core Team, 2018) con los paquetes ape (Paradis et al., 2004) y ggtree v. 1.12.7 (Yu et al., 2017).
Componentes Principales y Análisis de Ancestría
Para lograr un enfoque diferente con el fin de caracterizar la divergencia, se aplicó un análisis de componentes principales (ACP) a la matriz de relaciones construida a partir de la identidad por estado (IBS) entre todos los individuos. Todas las estimaciones y diagramas se realizaron a través de scripts de lenguaje bash y R (R Development Core Team, 2018) con el paquete ggplot2 (Wickham, 2016).
Se realizó un análisis de ancestría con el software ADMIXTURE v. 1.3 (Alexander et al., 2009) para identificar la conformación de grupos genéticos en todas las poblaciones. El programa implementa un método de máxima probabilidad para inferir la ascendencia genética de cada individuo a partir de una mezcla de K grupos ancestrales predefinidos. El número de agrupaciones (K) analizadas varió de 2 a 7. Un valor preferible de K exhibirá un bajo error de validación cruzada en comparación con otros valores de K.
RESULTADOS Y DISCUSIÓN
Diversidad Genética
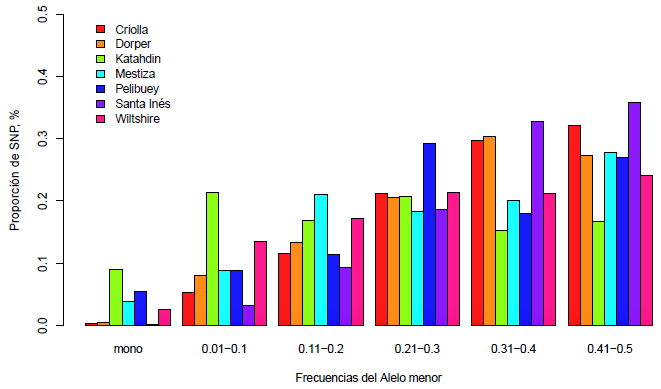
La proporción de marcadores genotipados en más del 95% de las muestras fue superior al 99% en todos los grupos poblacionales, con excepción de Katahdin que fue de 97.95% (Cuadro 1). Esto sugiere la idoneidad del chip para genotipar las poblaciones estudiadas. La proporción de SNP con un MAF >0.05 osciló entre 92 y 98% (con excepción de los animales Wiltshire) lo que indica que la mayoría de los SNP se están segregando en todos los grupos poblacionales.
Cuadro 1 Diversidad genética dentro de poblaciones ovinas de pelo en la Región Caribe colombiana
| Grupo poblacional | Marcadores genotipados en más del 95% de las muestras (%) | Marcadores con un MAF1 >0.05 (%) | Marcadores en equilibrio de Hardy-Weinberg (p>0.01) (%) | Heterocigosidad observada y desviación estándar (DE) | Heterocigosidad esperada y desviación estándar (DE) |
|---|---|---|---|---|---|
| Criolla | 99.78 | 98.20 | 96.27 | 0.401 (0.102) | 0.416 (0.102) |
| Dorper | 99.53 | 92.10 | 99.77 | 0.371 (0.184) | 0.382 (0.138) |
| Katahdin | 97.95 | 94.49 | 100.0 | 0.398 (0.216) | 0.365 (0.142) |
| Mestiza | 99.69 | 98.92 | 98.96 | 0.431 (0.106) | 0.433 (0.090) |
| Pelibuey | 99.83 | 92.25 | 99.44 | 0.374 (0.162) | 0.362 (0.143) |
| Santa Inés | 99.65 | 96.83 | 99.13 | 0.412 (0.141) | 0.401 (0.113) |
| Wiltshire | 99.99 | 81.89 | 99.30 | 0.311 (0.190) | 0.303 (0.167) |
1 MAF = Frecuencia de alelos menores (Minor allele frequency)
No se observaron desviaciones significativas del equilibrio de Hardy-Weinberg de los marcadores en las poblaciones evaluadas, pues el porcentaje de marcadores en el equilibrio de Hardy-Weinberg (p>0.01) fue superior al 98%, a excepción de los animales criollos, los cuales presentaron un porcentaje del 96%.
La heterocigosidad esperada en todas las poblaciones fue 0.378, con estimaciones que van desde 0.303 en la raza Wiltshire (empleada como outgroup) hasta 0.431 en animales mestizos, lo que indica que existe una gran diversidad dentro de los grupos poblacionales. En ovinos criollos de pelo colombiano (OPC), Ortiz et al. (2021) encontraron una heterocigosidad esperada de 0.374, inferior al valor de 0.416 para la población criolla en este estudio. Los valores más altos de heterocigosidad se determinaron en las poblaciones criollas y mestizas, pudiendo ser el resultado del limitado proceso de selección en estos grupos genéticos. Por tanto, la gestión de dicha variabilidad a través de apareamientos dirigidos puede contribuir a definir esquemas de contribución optima, donde se conserve la variabilidad al mismo tiempo que se identificarían animales con mérito genético superior. No obstante, es necesario definir poblaciones de grupos genéticos relacionados para establecer aspectos ambientales que mejoren la respuesta productiva de los individuos.
Para conocer con más detalle el grado de polimorfismo observado en todas las poblaciones, se representó la proporción promedio de SNP para diferentes rangos de MAF. El porcentaje de loci monomórficos fue inferior al 10% en todos los grupos poblacionales, siendo Katahdin, Pelibuey y las mestizas las poblaciones con los valores más altos de alelos fijados (Figura 1).
Varianza Molecular e Índices de Fijación
La parte más importante de la variación molecular (90.58%; p<0.001) fue atribuible a la variación dentro de los individuos. La variación entre poblaciones representó el 7.45% (p<0.001) y la variación entre individuos dentro de las poblaciones fue incluso menor (1.97%; p<0.05) (Cuadro 2). Los resultados indican que estos grupos tienen bajos niveles de variación genética entre poblaciones, lo que podría ser explicado por el alto mestizaje entre poblaciones.
Cuadro 2 Análisis de varianza molecular (AMOVA) en seis grupos poblacionales de la región Caribe Colombiana1
| Fuente de variación | gl | Componentes de varianza | Porcentaje de variación | Índices de fijación |
|---|---|---|---|---|
| Entre poblaciones | 6 | 174.827 | 7.45 | FST = 0.075*** |
| Entre individuos dentro de poblaciones | 474 | 46.100 | 1.97 | FIS = 0.021* |
| Dentro de individuos | 481 | 2124.299 | 90.58 | FIT = 0.094*** |
| Total | 961 | 2345.226 |
1 La raza Wiltshire fue empleada como outgroup
* p value < 0.001
El AMOVA permitió estimar los índices de fijación FIT, FIS y FST en todos los loci (Cuadro 2). Se encontró una endogamia total (valor FIT) de 0.094, una endogamia dentro de la población (valor FIS) de 0.021 y una diferenciación genética entre razas (valor FST) de 0.075. El bajo valor de FIS confirma que las frecuencias alélicas de las poblaciones se encuentran en las proporciones esperadas de Hardy-Weinberg. El valor FST de 0.075 indicó la existencia de una estructura poblacional limitada entre los grupos poblacionales estudiados, según la calificación de Wright (1965). Esta baja diferenciación entre poblaciones podría atribuirse a una falta de presión de selección o la existencia de un flujo genético (mestizaje) entre estas.
De acuerdo con Meyermans et al. (2020), la evaluación de las relaciones entre las poblaciones permite identificar si los reproductores tienen la misma oportunidad de contribuir al acervo genético de la población. El resultado muestra que existe oportunidad para realizar gestión de la población, pero en especial, definir con mayor precisión la combinación de razas o grupos de individuos dentro de cada población para mantener la variabilidad. En un muestreo de 362 individuos de tres poblaciones criollas en Colombia 2017, empleando marcadores microsatélites, se encontraron valores promedio de FIS = 0.107 y FST = 0.054, presentando razas de ovejas con niveles moderados de consanguinidad y bajo grado de diferenciación genética entre las distintas razas (Ocampo et al., 2017).
Los índices FIS específicos en cada grupo poblacional mostraron valores cercanos a 0, que van desde -0.018 en Pelibuey a 0.036 en la población Criolla (Cuadro 3). Estos resultados coinciden con las diferencias encontradas entre la heterocigosidad observada y esperada en el Cuadro 1, e indican una mayor proporción de heterocigotos principalmente en las poblaciones Pelibuey y Katahdin, y mayor proporción de homocigotos en las poblaciones Criolla y Dorper.
Cuadro 3 Estimaciones de los valores de FIS (coeficiente de endogamia) para seis gripos poblacionales de ovinos de pelo en la Región Caribe colombiana (La raza Wiltshire fue empleada como outgroup) (1023 permutaciones)
| Grupo poblacional | Valor de FIS | Valor P |
|---|---|---|
| Criolla | 0.0357 | 0.0000 |
| Dorper | 0.0123 | 0.4448 |
| Katahdin | -0.0165 | 0.5699 |
| Mestiza | 0.0003 | 0.5044 |
| Pelibuey | -0.0181 | 0.6862 |
| Santa Inés | -0.0124 | 0.6266 |
| Wiltshire | -0.0058 | 0.5337 |
El exceso de homocigotos en la población Criolla fue similar, aunque no tan altos, a los valores reportados por Ortiz et al. (2021) en el OPC de entre 0.075 y 0.089, lo que puede ser indicio de algún grado de consanguinidad en estas poblaciones.
Medidas de Distancia y Árbol Filogenético
Los estadísticos FST entre pares de poblaciones fueron empleados como una medida de la distancia genética entre los grupos poblacionales evaluados. Estos mostraron valores que variaron de 0.015 a 0.251 (Cuadro 4). Como era de esperarse, las estimaciones más bajas de FST se observaron entre animales mestizos y las demás poblaciones y las más altas se encontraron entre la raza empleada como outgroup y todas las demás. La distancia genética D de Nei (1972) mostró un patrón similar al obtenido con las estadísticas de FST con valores que van desde 0.011 a 0.069.
Cuadro 4 Estimaciones de las estadísticas de FST (por debajo de la diagonal) y las distancias genéticas D de Nei (por encima de la diagonal) entre pares de poblaciones (20022 permutaciones) de ovinos de pelo de la Región Caribe colombiana
| Grupo poblacional | Criolla | Dorper | Katahdin | Mestiza | Pelibuey | Santa Inés | Wiltshire |
|---|---|---|---|---|---|---|---|
| Criolla | 0.0455 | 0.0477 | 0.0110 | 0.0334 | 0.0290 | 0.0674 | |
| Dorper | 0.0985 | 0.0559 | 0.0418 | 0.0546 | 0.0477 | 0.0657 | |
| Katahdin | 0.0877 | 0.1374 | 0.0409 | 0.0518 | 0.0492 | 0.0620 | |
| Mestiza | 0.0147 | 0.0712 | 0.0448 | 0.0366 | 0.0227 | 0.0694 | |
| Pelibuey | 0.0627 | 0.1602 | 0.1214 | 0.0647 | 0.0457 | 0.0651 | |
| Santa Inés | 0.0586 | 0.1103 | 0.1015 | 0.0342 | 0.1170 | 0.0660 | |
| Wiltshire1 | 0.1883 | 0.2488 | 0.2173 | 0.1700 | 0.2506 | 0.2142 |
1 Empleada como outgroup
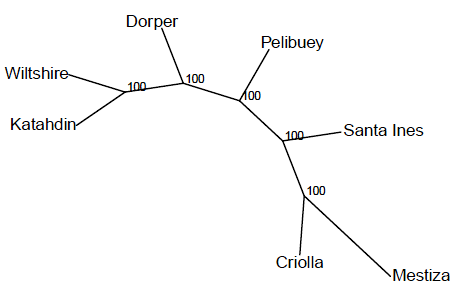
En el dendrograma, a partir de las distancias genéticas (Figura 2), se observa como el outgroup (raza Wiltshire) y la población de Katahdin fueron colocadas muy separadas de los demás grupos. En un punto medio se ubicaron las poblaciones Dorper y Pelibuey, en tanto que los grupos poblacionales Santa Inés, Criolla y los mestizos se ubicaron con mucha cercanía indicando una estrecha relación entre ellas. Es importante resaltar que los porcentajes de bootstrap representados en los nodos internos de la Figura 2, mostraron valores del 100%. El dendrograma muestra como los animales mestizos y criollos comparten el mismo nodo, lo cual puede asociarse a que la mayoría de los cruzamientos emplean la población Criolla como base.
Análisis de Componentes Principales
El ACP para datos de SNPs (Figura 3) se ha utilizado ampliamente para resumir la estructura de la variación genética (Wang et al., 2012) y puede guardar relación con la distribución geográfica de las muestras. Por esta razón, se realizó un ACP empleando la matriz de distancia construida a partir de la matriz de relaciones conocida como Idénticos por Estado (IBS, por sus siglas en inglés). Este análisis permitió representar a cada animal en particular sobre la base de las coordenadas del ACP.
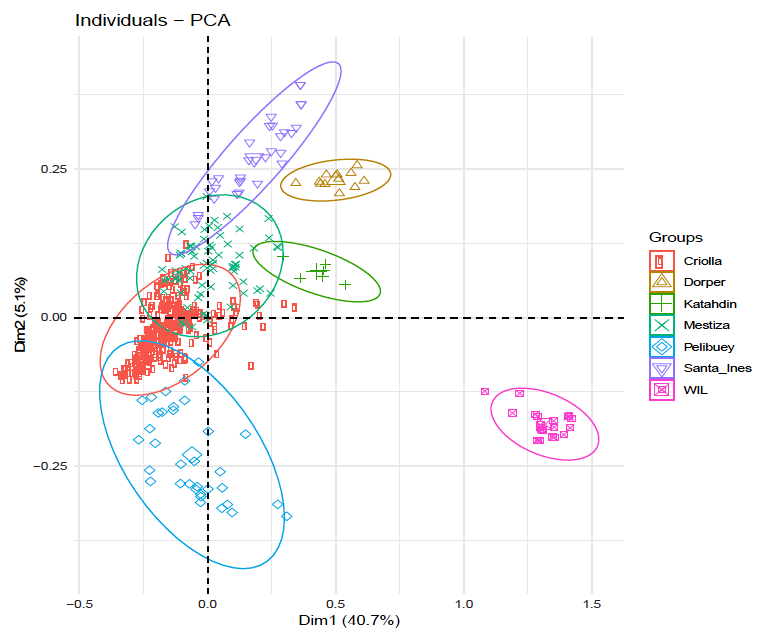
Figura 3 Representación de siete grupos poblacionales ovinos de la Región Caribe colombiana definidos por medio del análisis de componentes principales
El primer y segundo eje de ACP representaron el 40.7 y 5.1% de la varianza total, respectivamente. En general, las agrupaciones de APC sugirieron un patrón de clasificación similar al observado en el árbol N-J. Sin embargo, se deben resaltar varias características de los resultados. Primero, los animales Wiltshire se distanciaron en una alta proporción de los demás grupos raciales, lo que confirma los valores altos de FST y las distancias D de Nei. En segundo lugar, los animales mestizos presentan alto grado de mezcla con individuos de las poblaciones Criollas y Santa Inés, indicando cercanía genética. Tercero, hay una clara dispersión de los animales del grupo poblacional Pelibuey, coincidiendo justamente con la alta proporción de heterocigotos reflejado en el valor más bajo de FIS de todas las poblaciones (Cuadro 3).
Estructura Genética y Niveles de Mestizaje
El análisis de conglomerados permitió caracterizar la divergencia entre los grupos poblacionales, método de máxima probabilidad que infiere la ascendencia genética de cada individuo a partir de grupos ancestrales predefinidos.
Los grupos ancestrales probados variaron de K=2 a K=7. Los errores de validación cruzada más bajos obtenidos fueron de 0.62558 para K=7 y de 0.62899 para K=6, lo que indica que estos fueron los grupos más parsimoniosos. Tanto para K=6 como para K=7, la estimación de probabilidad máxima de ancestros asignó a todos los individuos a grupos que coinciden con la población de origen, aunque también se reveló alto grado de mezcla entre las poblaciones, lo que se observa en gran medida en la población de mestizos (Figura 4). Para K=6, se observó una proximidad en las poblaciones Criollo, Santa Inés y Mestiza, lo cual confirma los resultados de la ACP y NJ (Figuras 2 y 3).
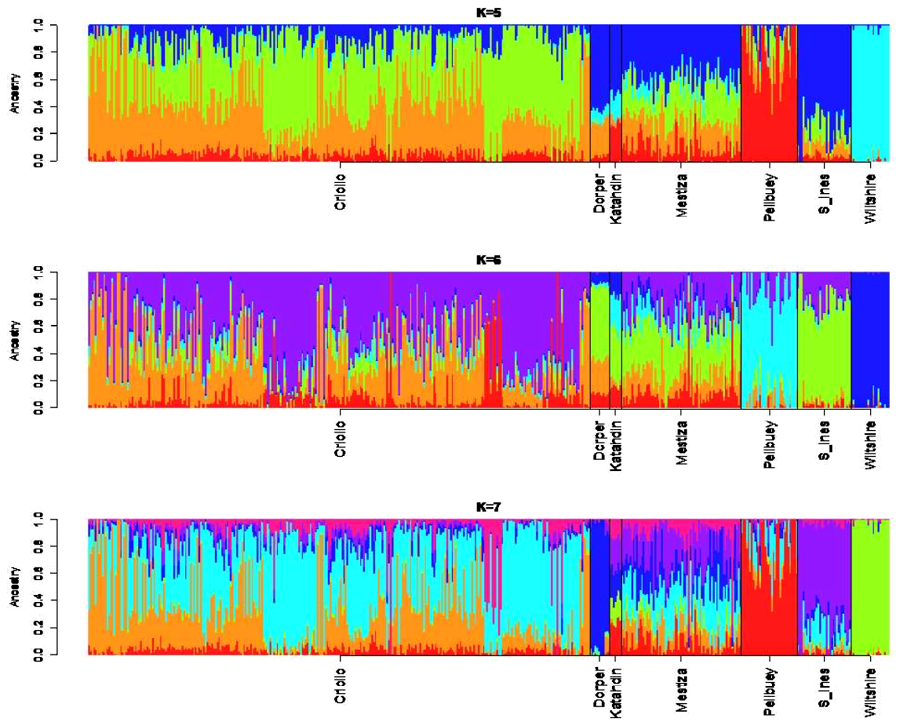
Figura 4 Coeficientes de ancestría estimados para cada individuo con K=5-7 en los siete grupos poblacionales ovinos de la Región Caribe colombiana (La raza Wiltshire fue empleada como outgroup)
En la población Criolla, a pesar de que la mayoría de los animales presentan diferenciación con los demás grupos poblacionales, se observa alto grado de mestizaje, lo que pudo generar la asignación errónea frente al grupo genético.
CONCLUSIONES
Se encontró un alto grado de diversidad genética dentro de los individuos y bajos niveles de variación genética entre grupos poblacionales.
La estructura poblacional fue limitada entre las poblaciones debido principalmente a la falta de presión de selección y a la existencia de un alto grado de mestizaje.
Los análisis de distancias genéticas mostraron una proximidad en las poblaciones Criollo, Santa Inés y animales mestizos con alto grado de mezcla entre los individuos, indicando cercanía genética.












 uBio
uBio