INTRODUCCIÓN
El desarrollo del folículo piloso tiene lugar durante el desarrollo fetal y está estrechamente regulado por las interacciones ectodérmicas-mesodérmicas. Se caracteriza por un crecimiento periódico (Alonso y Fuch, 2006; Roca et al., 2009). Después del nacimiento, los folículos pilosos maduros y en crecimiento activo acaban anclándose en el tejido subcutáneo y se regeneran periódicamente sometiéndose espontáneamente a ciclos repetitivos (Zhang et al., 2020). El ciclo de crecimiento del pelo, de la lana en las ovejas y de la fibra en los camélidos sudamericanos puede definirse típicamente en una fase anágena que ocurre cuando se produce un nuevo pelo, una fase catágena durante la cual cesa la elongación del pelo y una fase telógena cuando el folículo está en reposo (Alonso y Fuch, 2006). Se asume que el supuesto "reloj del ciclo del cabello" está compuesto por uno o más pares de genes activadores/inhibidores que actúan para controlar las transiciones de fase del ciclo (Chen et al., 2015; Wang et al., 2017).
El factor de crecimiento de fibroblastos (FGF5) es un miembro de la familia FGF, y consta de tres exones separados por dos intrones (Hébert et al., 1994). La proteína FGF5 de longitud completa induce la transición de la fase anágena a la catágena en el crecimiento del cabello, mientras que la proteína FGF5c (corta) antagoniza la proteína FGF5 (Suzuki et al., 1998, 2000), lo que sugiere que su expresión conduce a la terminación anágena y a la inducción catágena (Zhang et al., 2020). Tras la identificación del FGF5 en el fenotipo del ratón de Angora, se ha demostrado que las variantes genéticas de este gen afectan a la longitud del pelo en el gato (Drögemüller et al., 2007), perro (Dierks et al., 2013), cabra de Angora (Wang et al., 2015), oveja (Hu et al., 2017), cuy (Yu et al., 2018), llama (Daverio et al., 2017), alpaca (Wang et al., 2015; Pallotti et al., 2018), camello (Maraqa et al., 2021) y en el humano (Higgins et al., 2014).
La alpaca, Vicugna pacos, presenta dos razas: Suri y Huacaya, que se diferencian por las características fenotípicas de las fibras. La Suri tiene fibras largas organizadas en rulos que caen a los lados del cuerpo, de manera similar a lo que se observa en la raza de ovejas Lincoln; lo cual da al animal una apariencia angular (Fernández-Baca, 2005). En cambio, la alpaca Huacaya tiene un vellón esponjoso, con fibras más cortas, similar al vellón de las ovejas Corriedale, dándole al animal una apariencia más voluminosa (Fernández-Baca, 2005). En las llamas también existen dos razas, una de fibra corta (Q'ara) con un alto porcentaje de fibras meduladas (Martínez et al., 1997) y otra de fibra larga (Chak'u), que tiene un crecimiento de fibra de 15.5 a 16.7 cm (Ayala, 1992; Frank, 1999). El guanaco, ancestro de la llama, tiene fibras cortas de 1.4 a 3.8 cm (Von Thüngen et al., 2005). Tanto la cachemira como las fibras finas de los camélidos silvestres se producen en vellones denominados de "doble capa" o heterogéneos, ya que las fibras finas y valiosas están mezcladas y bajo una cubierta de pelos largos y gruesos sin valor (Sacchero y Mueller, 2005). El crecimiento de la fibra de la vicuña no es rápido, con una media anual de 3.3 cm de longitud en el manto y que oscila entre 2.9 y 4.2 cm en los adultos (Quispe et al., 2009).
Poco se sabe sobre los genes implicados en la longitud de la fibra en los camélidos sudamericanos. El objetivo del presente estudio fue analizar el gen FGF5 y sus variantes en las cuatro especies de camélidos sudamericanos, así como su papel en la longitud de la fibra para animales de fibra corta y larga.
MATERIALES Y MÉTODOS
Se utilizaron 16 alpacas (9 Huacaya y 7 Suri), 6 llamas (4 Q'aras y 2 Chak'us) procedentes de las regiones de Puno, Cusco, Arequipa y Cerro de Pasco (Perú), y solo una vicuña y un guanaco por estar protegidos por Ley en el Artículo 66° de la Constitución Política del Estado Peruano, teniendo autorizaciónAUT-IFS-2018-046 emitida por SERFOR-MINAGRI para el muestreo de camélidos silvestres.
Se tomaron muestras de sangre por venopunción yugular en tubos tipo vacutainer con EDTA. Las muestras en el caso de la vicuña y el guanaco se tomaron durante los chaccus programados en las comunidades de Maranganí (Cusco) y de Huaycho (Puno). Las muestras se almacenaron a -20 ºC y fueron trasladadas al Centro de Investigación La Raya de la Universidad Nacional de San Antonio Abad del Cusco, Perú. El ADN fue extraído de la sangre total utilizando el Purelink Genomic DNA Mini kit siguiendo las indicaciones del protocolo del fabricante (Thermo Fisher Scientific).
Se preparó una biblioteca de ADN genómico utilizando un secuenciador BGISEQ-500, con un inserto de ~300 pb para cada muestra analizada. Se obtuvo una cobertura de 50× en el secuenciador BGISEQ-500. Las lecturas de las secuencias se asignaron al ensamblaje del genoma de referencia de alpaca VicPac3.1 (GCF_000164845.3). Se aplicó el algoritmo Burroughs Wheeler Aligner MEM para alinear las lecturas. Los procesos de detección de polimorfismos de un solo nucleótido (SNP) y de InDels se realizaron con el software GATK (https://www.broadinstitute.org/gatk/). Además, se utilizó el programa Annovar (Wang y Hakonarson, 2010) para obtener anotaciones y clasificaciones. Para el modelamiento de las proteínas se utilizó el programa Swiss Model (Waterhouse et al., 2018).
La información de la secuencia analizada de las cuatro especies para el gen FGF5 se presentó en GenBank con los números de acceso OM238194, OM238195, OM238196, OM238197, OM238198, OM238199 y OM238200. También se utilizó el número de acceso del NCBI, NW_021964157.1 para la referencia del genoma de alpaca.
RESULTADOS
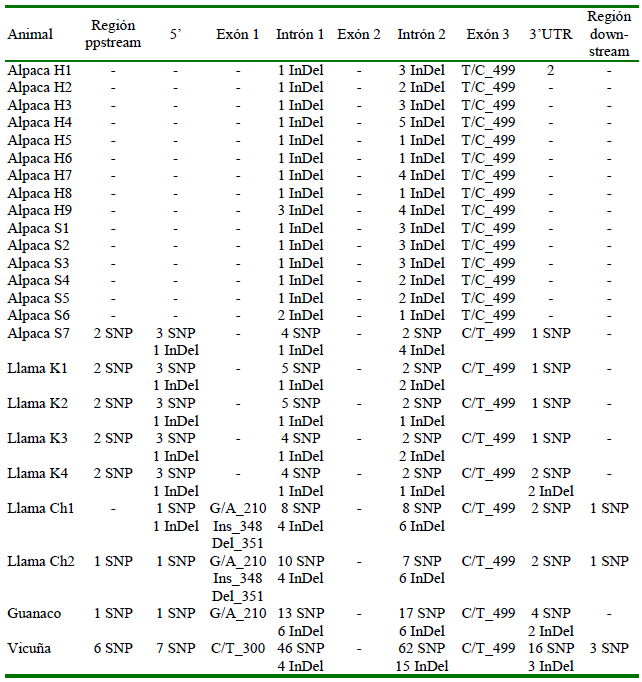
Quince de las 16 alpacas tenían un cambio de aminoácido del nucleótido T en la posición 499 T>C. Este cambio acortó la longitud de la proteína a 166 aminoácidos (Figura 1) que estaban presentes en el exón 3. En las llamas Q'ara, vicuña, guanaco se determinó la proteína completa con 270 aminoácidos (Figura 1, panel inferior). En las llamas Chak'u se determinó una inserción de 12 nucleótidos (GCCATATAACAT_Ins) en la posición 348 y una deleción (C del) en la posición 351. Esta deleción causó un codón de parada prematuro en la posición de 118 aminoácidos (Figura 1). Ambas mutaciones estaban presentes en el exón 1.

Figura 1. Secuencia de aminoácidos de 24 camélidos sudamericanos. Los números entre paréntesis indican el número de animales con la misma secuencia de aminoácidos. Un tamaño corto en las llamas Chak'us (118 aminoácidos), en las alpacas Huacaya y Suri (166 aminoácidos) y una proteína de tamaño completo (270 aminoácidos) en las llamas Q'ara, guanaco, vicuña y en una alpaca Suri
Tres variantes de la proteína FGF5 estaban presentes (Figuras 2, 3, 4 panel A). La isoforma A, presente en las llamas Chak'u (Figura 2), la isoforma B, presente en las alpacas Huacaya y Suri (Figura 3) y, la isoforma C, presente en las llamas Q'ara, vicuña y guanaco (Figura 4). En la vicuña se determinaron 142 SNPs (polimorfismo de un solo nucleótido) + 22 InDels (inserciones y deleciones), y en el guanaco 38 SNPs +14 InDels (Cuadro 1).

Figura 2. Composición de la isoforma corta de la proteína FGF5 (A) presente solo en las llamas Chak'u. (B). Cuando la proteína está afectada se observa un mayor crecimiento del vellón en comparación con su ancestro, el guanaco

Figura 3. Composición de la isoforma corta de la proteína FGF5 (A) presente en las alpacas Suri (B) y Huacaya (C). La presencia de esta isoforma afectó el crecimiento de la fibra larga con respecto a su ancestro vicuña

Figura 4. Una composición de la proteína FGF5 completa (A) presente en vicuña (B), guanaco (C), llamas Q'ara (D), y en una alpaca Suri. La regulación de la longitud de la fibra no permite un vellón largo en estas especies
DISCUSIÓN
El análisis completo del gen FGF5, y su función sobre la longitud de la fibra, reportado en este estudio, fue utilizado para identificar todas las variantes presentes en las cuatro especies de camélidos sudamericanos y sus razas. Cabe recordar que estos animales están sometidos a una esquila anual, especialmente en las alpacas y llamas Chak'u. Por otro lado, la comparación entre especies permitió conocer mejor el gen FGF5.
Quince de las 16 muestras de alpacas presentaban polimorfismos en la posición 490 con un cambio de timina por citocina que a su vez afectaba al tamaño de la proteína. Esta posición representaba un codón de parada prematuro en el exón 3. Esta isoforma corta también fue reportada por Pallotti et al. (2018) en las razas Huacaya y Suri. Las variantes encontradas en las llamas Chak'u en el exón 1, donde la inserción de 12 nucleótidos en la posición 348, y una deleción de citocina en la posición 351 representaron un codón de parada prematura que acortó la proteína. En este sentido, Daverio et al., (2017), informaron de un cambio diferente; es decir, una deleción en la posición 348 y una inserción de 12 nucleótidos en la posición 351. Esta contradicción podría deberse a una repetición de nucleótidos que permitiría un cambio en las posiciones antes mencionadas y, debido posiblemente al método de secuenciación utilizado, siendo en el presente caso información de los genomas completos. Además, la isoforma corta solo está presente en llamas Chak'u, que son completamente diferentes en cuanto a la longitud de la fibra de las llamas Q'ara. En el guanaco, la secuencia reportada por Daverio et al. (2017) es similar a los resultados del presente reporte, presentando la proteína de 276 aminoácidos que regula el crecimiento de la fibra.
Según estudios previos, el gen FGF5 podría inhibir el crecimiento normal del pelo en ratones, y su mutación de pérdida de función provocó un crecimiento anormal del pelo con lo cual se extiende el ciclo de crecimiento del pelo (Sundberg et al., 1997; Kim et al., 2020). En humanos se ha identificado al gen FGF5 como regulador del crecimiento y la caída del cabello (Kim et al., 2020). En cabras y ovejas, utilizando la tecnología CRISPR Cas9 se pudo producir mutaciones de deleción funcional en el gen FGF5, apreciándose un crecimiento más largo del vellón que en animales con un FGF5 funcional (Hu et al., 2017; Li et al., 2017; Su et al., 2020). En el dromedario el gen FGF5 a nivel de la posición 779, el cambio de T por C afecta también el crecimiento del vellón, pero con un modo de herencia de dominancia incompleta. Estos resultados indican que la presencia del gen FGF5 puede afectar el crecimiento del pelo, donde un mayor crecimiento podría promoverse si se altera la expresión del gen FGF5, tal como se puede apreciar en el vellón de alpacas Huacaya y Suri, así como en llamas Chak'u, los cuales tienen un crecimiento de fibra más largo que sus ancestros silvestres y que la llama de K'ara donde el gen FGF5 es funcional.
En alpacas se observa que algunas de ellas no tienen un buen crecimiento de vellón, teniendo una esquila cada dos años, como fue el caso de la muestra de alpaca Suri, con lo que una evaluación de más animales podría indicar que las alpacas con una proteína FGF5 funcional tendrán un menor crecimiento en el vellón.
Los resultados revelaron que los camélidos sudamericanos silvestres (guanaco y vicuña) tienen un mayor número de polimorfismos en el gen FGF5 que sus homólogos domésticos (llama y alpaca). Las vicuñas presentaron 142 SNPs y 22 InDels, en tanto que el guanaco presentó 38 SNPs y 14 InDels. Esta mayor presencia de SNPs sería un indicio de adaptación a la altitud, tal como se informó en las cabras nepalesas que viven a gran altura y que también presentaron una mayor frecuencia de polimorfismos del gen FGF5 (Sasazaki et al., 2021). Se ha demostrado también una presencia de SNPs alta en el gen FGF5 en las ovejas de Etiopía (Edea et al., 2019).
Los resultados del presente trabajo en camélidos sudamericanos silvestres (Cuadro 1), mostraron muchos polimorfismos que indicarían adaptación a lugares altoandinos. Sin embargo, estos resultados deben tomarse con cautela, debido al pequeño número de animales muestreados, en parte por las dificultades para obtener muestras de especies en peligro de extinción y bajo un estricto control gubernamental. Por lo tanto, los resultados que sugieren que el gen FGF5 participa en la adaptación a la altitud es interesante y una función novedosa que debe ser tomada en consideración.
CONCLUSIONES
Un SNP está presente en las alpacas (T/ C_499) causando un codón de parada prematuro en el aminoácido en la posición 166. En llamas Chak'u, hay un cambio de aminoácido en la posición 118, la inserción de 12 nucleótidos y la supresión de una C en la posición 351. Estas variantes causan una longitud de fibra definitivamente mayor en alpacas Huacaya y Suri, así como, en llamas Chak'u.
En las llamas Q'ara, vicuña y guanaco ningún SNP afecta la estructura del gen FGF5 que es de 270 aminoácidos. Sin embargo, existe un mayor número de variantes del gen FGF5 que podrían estar relacionadas con la adaptación a la hipoxia.












 uBio
uBio