Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Ecología Aplicada
versión impresa ISSN 1726-2216
Ecol. apl. v.5 n.1-2 Lima dic. 2006
ARTICULOS ORIGINALES
Análisis de la variabilidad molecular de una colección peruana de Smallanthus sonchifolius (Poepp & Endl) H. Robinson Yacón
Molecular variabiliy analysis of a peruvian smallanthus sonchifolius (Poepp & Endl) H. Robinson "Yacon" Collection
Roberto C. Mansilla S.1; César López B.1; Raúl Blas S.1; Julio Chia W.1 ; J. Baudoin2
1 Instituto de Biotecnología de la Universidad Nacional Agraria la Molina. Apartado postal 12056 Lima 12 - Perú. rmansilla@lamolina.edu.pe / cflb@lamolina.edu.pe
2 Faculté Universitaire des Sciences Agronomiques de Gembloux, Bélgica.
RESUMEN
El yacón es un cultivo de origen andino caracterizado por almacenar principalmente fructooligosacáridos en sus raíces; sin embargo, no se conoce aún cuanta diversidad genética a nivel molecular existe en la especie. En el presente estudio se caracterizó 30 accesiones de yacones cultivados provenientes del norte, centro y sur del Perú mediante la técnica de RAPDs, utilizando 34 iniciadores decaméricos; con los cuales se muestrearon 166 fragmentos genómicos, de las cuales el 30.7% fueron polimórficos. Al 0.58 de similitud se formaron 7 grupos de yacones. El primero de ellos con 22 accesiones agrupa a todas las provenientes del norte y del sur, y algunas del centro, los grupos restantes solamente contienen accesiones del centro. No se logró encontrar material duplicado y la mayor diversidad estuvo concentrada en las accesiones provenientes del centro del Perú. El AMOVA reportó un 21.14% de variación genética explicada por el componente interregional y la mayor variabilidad estuvo concentrada entre las accesiones de cada región (78.86%).
Palabras clave: yacón, germoplasma, marcador molecular, RAPD, diversidad, raíz andina, Smallanthus sonchifolius
ABSTRACT
Yacon is an Andean origin crop, characterized by the storage of mainly fructooligosaccharides in its roots. In spite of advancements in yacon morphological characterizations, the genetic diversity of the crop in molecular terms is still unknown. In the present study 30 cultivated accessions from the northern, center and southern regions of Peru were characterized by RAPDs. With 34 decameric primers 166 bands were generated, of which 30.7% were polymorphic. An index of similarity of 0.58 was chosen and 7 well differentiated subgroups or clusters were formed. The first cluster groups 22 accessions from the northern and southern regions with some from the center. The remaining clusters contain exclusively center region accessions. Duplicated individuals were not found and the greatest diversity was found in the center region of Peru. AMOVA reported 21.14% of variation explained by the among regions component and higher variability was concentrated among the accessions of each region (78.86%).
key words: yacón, germplasm, molecular marker, RAPD, diversity, andean root, Smallanthus sonchifolius
INTRODUCCIÓN
El Yacón es una planta con raíces reservantes; cultivada en la zona andina desde épocas prehispánicas y consumida habitualmente en forma cruda, como fruta en las zonas rurales, (Herman et al., 1997). Se extiende desde Venezuela hasta el noreste de Argentina; además, en la actualidad se encuentra en algunos países tales como Nueva Zelanda, Japón, Corea, Brasil, República Checa, China, Estado Unidos, Paraguay, Taiwán (Seminario et al., 2003).
El Yacón en sus raíces almacena diferentes carbohidratos como fructosa, glucosa y principalmente oligosacaridos de bajo grado de polimerización (mayormente de 3 a 10 oligofructanos), trazas de almidón e inulina. Los fructooligosacáridos pueden llegar a representar el 67% de materia en la raíz (Asami, 1990).
La escasa información de la biología reproductiva indica que predomina la propagación clonal; mientras que a nivel de la reproducción sexual se reporta reducida producción de flores y baja viabilidad de semillas (Grau & Rea, 1997). En cuanto a la estructura cromosómica este cultivo seria octoploide (6A + 2B con 2n = 58), probablemente resultado del cruce interespecífico entre el S. macroscyphus y el S. riparius (Ishike et al., 1997).
A través de la historia el cultivo del yacón ha permanecido restringido al campesinado, lográndose avances en el proceso de domesticación en algunas zonas andinas (Rea, 1998). Sin embargo, en los últimos años se ha venido incrementando el consumo, probablemente debido a la difusión de las propiedades antidiabéticas que tradicionalmente se la ha atribuido; lo que le ha permitido la salida de las frontera andinas hacia otras latitudes. Así mismo, debido al incremento de las áreas de cultivo, se hace necesario identificar y estudiar diversos caracteres y estrategias de mejora que permitan encontrar genotipos adecuados para la industria alimentaria. El proceso de mejora requiere de evaluar e identificar la variabilidad genotípica y fenotípica; es por ello que en relación a la variación fenotípica, se han identificado 9 morfotipos de yacón en la colección del Instituto Nacional de Investigación Agraria (INIA), 10 en la del Centro Internacional de la Papa (CIP), 8 en la Universidad de Cajamarca y 8 en la Universidad del Cusco (Arbizu 1999, comunicación personal). En el presente estudio nuestro objetivo fue analizar la diversidad genética a nivel molecular mediante marcadores Random Amplified Polymorphic DNA (RAPDs) en una colección de germoplasma como modelo aproximativo para colecciones más grandes; considerando que esta técnica se ha empleado para estudiar la diversidad genética, la elaboración de mapas de ligamento entre otros, para diversas especies, así lo indican Bruford et al. (1998) y Ferreira et al. (1998).
MATERIALES Y MÉTODOS
Material Vegetal
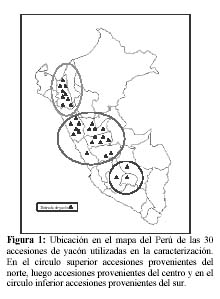
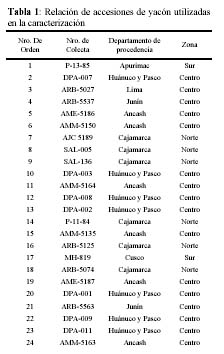
Se emplearon 30 accesiones de yacón mantenidos en el banco de germoplasma del CIP, provenientes de diferentes departamentos del Perú, expresadas en 3 zonas geográficas: norte, centro y sur (Tabla 1 y Figura 1).
Aislamiento del ADN
El ADN fue extraído de hojas jóvenes (tejido fresco), colectado en el invernadero del CIP. El protocolo de extracción consistió en una modificación del método cetil-trimethylammonium bromide (CTAB) de Doyle & Doyle (1990). La concentración y pureza del ADN se determinó mediante espectrofotómetría (Maniatis et al., 1984).
Análisis RAPD
La metodología se basó en los reportes de Williams (1989) y Williams et al. (1990). Para ello se utilizaron 34 iniciadores decaméricos aleatorios, adquiridos de Operon Technologies (Alameda, CA). La amplificación Polymerase Chain Reaction (PCR), se realizó en 15 ul de volumen de reacción, y se optimizó usando el perfil de amplificación mas adecuado en un termociclador Perkin Elmer modelo 9700.
Los productos de la reacción fueron separados por electroforesis en geles de agarosa al 2%, utilizando solución tampón NEB (Tris 1M, EDTA 10mM y Acetato de Sodio 125mM) pH 8.1. Los fragmentos de ADN fueron visualizados por tinción con bromuro de etidio (0.5ug/ul) por 40 minutos.
Análisis de los datos
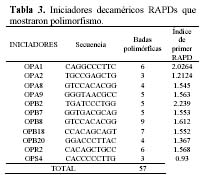
Para la elección de los iniciadores por su capacidad de reportar polimorfismo, se determinó el índice del iniciador, definido como: ∑ (1 – pi2 – qi2), donde pi y qi son las frecuencias de presencias o ausencias de las bandas producidas por el iniciador.
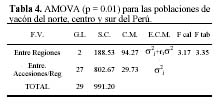
Las accesiones de yacones peruanos fueron agrupadas en 3 regiones, en accesiones/región y todas las accesiones a la vez. Los marcadores RAPDs polimórficos fueron escoriados como presencia (1), ausencia (0) y datos dudosos (9). Con esta matriz básica de datos, se construyó la matriz de similitud utilizando el coeficiente de similitud de Jaccard, citado por Crisci et al. (1983), que permite estimar el nivel de semejanza genética a través de su ADN; y finalmente el análisis de agrupamiento (Cluster analysis) para conocer la estructura poblacional de la colección, mediante la técnica de ligamento medio o Unweighted Pair Group with Arithmetic Average (UPGMA), para ello se utilizó el programa NTSYS pc2.1 (Rohlf, 2000), Se ha estimado la varianza de los componentes intrarregionales e interregionales mediante el análisis molecular de variancia (AMOVA).
RESULTADOS
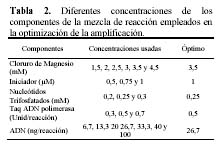
En el Tabla 2 se muestran las diferentes concentraciones de MgCl2, oligonucleótidos iniciadores, nucleótidos trifosfatados, enzima ADN taq polimerasa y de ADN genómico de yacón que se probaron para la estandarización de componentes de PCR. Se obtuvieron mejores resultados con la combinación 3.5 mM de MgCl2, 1µM de oligonucleótido iniciador, 0.25mM de cada nucleótido trifosfatado y 0.5 Unidades de ADN Taq polimerasa y 26.7 ng de ADN en 15 µl de volumen de reacción. Se probaron diferentes perfiles de amplificación, obteniéndose mejores resultados y al menor tiempo de amplificación con el perfil consistente en: 1 ciclo a 94ºC por 2 minutos, luego de 3 ciclos de 94ºC por 15 segundos, 35ºC por 15 segundos y 72ºC por 75 segundos; seguido de 40 ciclos de 94º por 15 segundos, 40º por 15 segundos y 72º por 75 segundos; y finalmente de 1 ciclo de 72º por 2 minutos y 4º hasta sacar las muestras del termociclador.
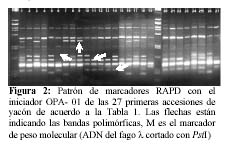
De los 34 iniciadores empleados, 11 mostraron polimorfismo, representando el 32.4% del total. El tamaño de los fragmentos varió entre 402 y 2292 pares de bases (pb). En total se han generado 166 bandas y de las cuales 57 fueron confiables y polimórficas, representando el 30.7%. La figura 2 ilustra la presencia de bandas polimórficas obtenidas con el iniciador OPA1, el cual mostró mayor índice de primer RAPD, mientras que OPS4 produjo el menor valor, Tabla 3.
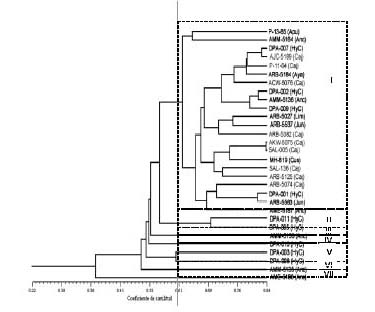
Del análisis de agrupamiento que se aprecia en la figura 3 (en apéndice), no se registran grupos con similitud 1 por lo que esa colección aparentemente no contendría material duplicado. A 0.58 de nivel de similitud se forman 7 grupos de yacones bien diferenciados. El primero de ellos con 22 accesiones agrupa a todas las provenientes del norte y del sur con algunas del centro. Los grupos restantes involucran solamente accesiones del centro.
En cuanto al AMOVA se observa variabilidad de 78.86% en el componente intrarregional y 21.47% en el componente interregional (Tablas 4 y 5)
DISCUSIÓN
De acuerdo al dendograma y a la matriz de similitud, no se registraría material duplicado en la colección de yacones del CIP, sin embargo morfológicamente existirían solamente 10 morfotipos en dicha colección (Dr. C. Arbizu 2005, comunicación personal). También se deduce que habría mayor variabilidad en el material proveniente del centro y sur, mientras que las accesiones del norte resultan ser más parecidos entre ellos. Esta tendencia se ha encontrado en accesiones de yacón ecuatorianos, donde en general habría muy poca variabilidad, mostrando mayores diferencias el material proveniente del sur de ese país (Morillo, 2001). Esto llevaría a pensar que cuanto mas al norte en los andes disminuiría la variabilidad de las accesiones provenientes de esas zonas, y al contrario hacia el sur la variabilidad continuaría esa tendencia hasta el centro y sur del Perú, y tal vez norte de Bolivia a partir de donde disminuiría la variabilidad conforme continua avanzándose hacia el sur, lo cual concuerda con lo reportado por Grau & Rea (1997) y Rea (1998) quienes sostienen que la zona donde haya mayor diversidad de yacones es al norte de Bolivia y sur del Perú. Esto, se debería al mayor movimiento de pocos materiales en el norte del Perú en comparación al centro y sur, donde se ha mantenido en condición de semicultivo y pudo tener mayor intercambio genético con sus parientes silvestres si se tiene en cuenta que en el Perú hay 7 de las 21 especies Smallanthus, convirtiéndose en la zona del mayor número de especies de ese género.
Debido al bajo número de accesiones provenientes de cada zona con que se cuenta en la colección, se complica la determinación de la diversidad que puedan hallarse en dichas zonas.
En cuanto al análisis molecular de varianza, la variabilidad intrarregional de 78% podría atribuirse a la mayor contribución que pudieron dar las diferencias entre accesiones de las zonas centro y sur. Sin embargo la variabilidad interregional 21.14% es de magnitud media y puede ser explicada teniendo en cuenta su reproducción clonal y los problemas para la reproducción sexual (muy común en las raíces andinas). Zhang et al. (1998) trabajando con Ipomoea batatas; al igual que el yacón, una raíz reservante andina poliploide, con accesiones provenientes de dos regiones (Americanas y Papua Nueva Guinea), al evaluar la diversidad genética con métodos similares a los empleados en este trabajo obtuvo 9.4% de variabilidad en el componente interregional. Considerando que el yacón es un poliploide con escasa reproducción sexual comparada a la I. batatas, se ha encontrado un nivel medio bajo de variabilidad interregional en el presente trabajo. Sin embargo, debido al estado de semicultivo el yacón ha podido sufrir proceso de selección natural en cada zona ecológica, por lo tanto es posible hallar en un menor área genotipos que puedan ser diferentes debido a la gran variabilidad ecológica de la región andina. Por lo tanto en las colectas que se realicen en el futuro deben de tenerse en cuenta esos niveles de variación intra e interregional.
CONCLUSIONES
Los resultados de caracterización molecular de 30 accesiones de yacón, muestran que no habría duplicación de genotipos en la colección y aparentemente habría bajos niveles de polimorfismo. Así mismo, la variación interregional seria de 21.14% mientras que la intraregional 78%, con una mayor diversidad de yacones la región central del Perú. Esto nos indicaría realizar futuras prospecciones en esta parte del país, con la finalidad de estudiar mejor la diversidad y establecer centros de conservación in-situ de yacón.
AGRADECIMIENTO
El presente trabajo de investigación fue financiado por Conseil Interuniversitaire de la Communauté Française (CIUF).
LITERATURA CITADA
Asami T. 1990. Composition of storage carbohydrate in tuber roots of yacón (Polymnia sonchifolia). Soil Sci. Plant. Nutr. 36: 167-171. [ Links ]
Bruford B. & Beaumont M. 1998. Binary data analysis. En Molecular tools for screening biodiversity. Ed. A. Karp, P. Isaac & D. Ingram. Thamsen Publishing. London U. K. P. : 329 – 331. [ Links ]
Crisci J. & López M. 1983. Introducción a la teoría y práctica de la taxonomía numérica. Serie de biología No.26, Secretaria General de La Organización de los Estados Americanos (OEA). Programa Regional de Desarrollo Científico y Tecnológico Washington, D.C. : 132. [ Links ]
Demeke T. & Adams R. 1994. The use of PCR – RAPD analysis in plant taxonomy and evolution. En PCR Technology current Innovations, editado por H. Griffin & A Griffin. CRC Press. : 179–192. [ Links ]
Doyle J.J. & Doyle J.L. 1990. Isolation of DNA from small amounts of plant tissues. BRL Focus. 12: 13-15. [ Links ]
Ferreira M.E. & Grattapaglia D. 1998. Introducción al uso de marcadores moleculares en el análisis genético. Embrapa – cenargen. Brasilia : 220. [ Links ]
Grau A. & Rea J. 1997. Yacón. En Hermann, M. & J. Heller, Editores. 1995. Andean Roots and Tubers: Ahipa, Arracacha, Maca and Yacón. IPGRI. Roma Italia. : 199–242. [ Links ]
Hermann M., Freire I. & Pazos C. 1997. Compositional diversity of the yacon storage root. CIP Program Report 1997-1998, Lima Peru.: 425-432. [ Links ]
Ishike K., Salgado Moreno V.X. & Arellano J. 1997. Revision of chromosome number and Karyotype of Yacon (Polymnia sonchifolia). Resúmenes del primer taller Internacional sobre recursos fitogenéticos del Noreste Argentino. INTA, Salta, Argentina. [ Links ]
López C. & Torne J. 1995. Optimización de la técnica Random Amplified polymorphic DNA (RAPD) en poblaciones de respuesta androgénica en maíz (Zea mays L.). Tesis de maestría en genética, Universidad Autónoma de Barcelona. : 15. [ Links ]
Maniatis Fritsch T.E. & Sambrook J. 1984. Molecular Cloning a Laboratory Manual. Cold Spring Harbor Laboratory. New York. [ Links ]
Morillo V. & Luis E. 2001. Análisis de polimorfismo en las colecciones de Jicama (Polymnia sonchifolia P. & E.) y Miso (Miriabilis expasa R. & P.) del Banco de Germoplasma del INIAP. Pontificia Universidad Católica del Ecuador departamento de Ciencias Biológicas. Quito. : 101. [ Links ]
Rea J. 1998. Recursos genéticos del yacón. En producción de raíces andinas, Manual de Capacitación. Compilador Juan Seminario. Lima- Perú. : 27. [ Links ]
Rohlf F. J. 2000. Numerical taxonomy and multivariate analysis system. Port Jefferson: Applied Biostatistics. [ Links ]
Seminario J., Valderrama M. & Manrique I. 2003. El Yacón: fundamentos para el aprovechamiento de un recurso promisorio. Centro Internacional de la Papa (CIP), Universidad Nacional de Cajamarca, Agencia Suiza para el Desarrollo y la Cooperación (COSUDE), Lima, Perú. [ Links ]
Williams F. 1989. Optimization strategies for the polymerase chain reaction. biotechniques –perquin elmer Product Application. Focus .7(7) : 762–767. [ Links ]
Williams J.G.K., Kubelik A.R., Livak K.J., Rafalski J.A. & Tingey S.V. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research. 18: 6531-6535. [ Links ]
Zhang D., Ghislain M. & Huaman Z. 1998. RAPD Variation in Sweet PotatoIpomoea batatas (L.)Lam. - Cultivars from South America and Papua New Guinea. Genetic Resources and Crop Evolution. 45: 271-77. [ Links ]
Apéndice
Figura 3: Dendograma que muestra niveles de similitud entre las accesiones del norte (en gris) del centro y las del sur. En paréntesis los departamentos de procedencia: Anc = Ancash, Apu = Apurimac, Aya = Ayacucho, Caj = Cajamarca, Cus = Cusco, HyC = Huánuco y Pasco, Jun = Junín y Lim = Lima.
Presentado: 21/07/2006
Aceptado: 25/09/2006