Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Ecología Aplicada
versión impresa ISSN 1726-2216
Ecol. apl. v.5 n.1-2 Lima dic. 2006
ARTICULOS ORIGINALES
Diversidad genética molecular de Mirabilis expansa mediante RAPD
Molecular genetic diversity of Mirabilis expansa using RAP
Julio A. Chia W.1; César F. López B.1; Raúl Blas S.1; J. Seminario2; R. Mansilla1 ; J. P. Baudoin3
1 Instituto de Biotecnología, Universidad Nacional Agraria La Molina. Apartado postal 12056 Lima 12 - Perú. julio.chiawong@gmail.com / cflb@lamolina.edu.pe
2 Facultad de Ciencias Agrarias y Forestales, Universidad Nacional de Cajamarca.
3 Faculté Universitaire des Sciences Agronomiques de Gembloux, Bélgica.
RESUMEN
Se estudió la diversidad genética mediante la técnica de RAPD en 37 accesiones de una colección del norte del Perú de chago o mauka Mirabilis expansa; Se obtuvieron 60 marcadores polimórficos con 9 de 19 iniciadores decaméricos. Se calcularon los índices de iniciador RAPD, obteniéndose los valores más altos con los iniciadores OPA04, OPA09 y OPA13, lo cual sugiere su uso valioso en futuras investigaciones con RAPD en colecciones más grandes o para especies de Mirabilis. Con el coeficiente de Simple Matching y el algoritmo UPGMA se obtuvo un dendograma del cual, a un coeficiente de 1, se observan 31 grupos. Esto indicaría unos 16.216 % de posibles duplicados en la colección de Germoplasma. Con un índice de similitud de 0.85 se encontró que se forman 8 clusters o grupos, sin coincidir en su mayoría con los 5 morfotipos reportados. Además se realizó un Análisis Molecular de Variancia con 2 componentes: interregional y entre accesiones/ región, cuyos valores fueron 21.69% y 78.3%, respectivamente; valores que sugieren una considerable contribución de variación genética gracias a las muestras de diversas partes del país.
Palabras clave: RAPD, Mirabilis expansa, raíz tuberosa, diversidad genética, AMOVA
ABSTRACT
A collection of 37 accessions of Mirabilis expansa, chago or mauka from northern Perú, was analyzed by RAPD assays and the molecular genetic diversity determined. In order to know which primer was more informative, the PIC values were summed up and a RAPD primer index calculated, resulting in the primers OPA04, OPA09 y OPA13 with the highest indexes, suggesting their potential utility in further research in Mirabilis species. From 9 of the 19 RAPD primers selected, we obtained 60 polymorphic markers, which yielded a Simple Matching Coefficient Dendogram, which shows 31 groups at a similarity index of 1. It also indicates a 16.216% of possible duplicates in the collection and with a similarity index of 0,85 we observed 8 clusters, with a significant difference compared to the morphological characterization data. An Analysis of Molecular Variance was performed, separating 2 variation components: among regions and among accessions/within region. The values obtained were 21,69% and 78,3% respectively, being both relatively high and showing considerable contribution of genetic variation from samples of diverse collection places.
Key words: RAPD, Mirabilis expansa, Tuber roots, genetic diversity, AMOVA
INTRODUCCIÓN
Mirabilis expansa (Ruiz & Pavón) Standley conocida como chago en Cajamarca y como mauka en el sur del Perú o Bolivia. Es una Nyctaginaceae, planta promisoria distribuida desde el Ecuador, Perú hasta Bolivia; encontrándose parientes silvestres desde Venezuela hasta Chile (Seminario, 1993).
Su importancia radica en que posee raíces reservantes con mayor contenido de proteínas, calcio y fósforo con respecto a otras tuberosas (Franco & Uceda, 1996), pudiendo complementar las deficiencias nutricionales del poblador andino. En otros estudios se ha encontrado que plantas del género Mirabilis poseen proteínas inactivadoras de ribosomas (Ribosome Inactivating Proteins) con propiedades antivirales, que impiden la infección de virus y viroides transmitidos mecánicamente en cultivos como papa y tomate previamente aplicados con extractos crudos de tales plantas (Vivanco, 1999).
El cultivo es prácticamente desconocido para el consumidor común de la costa y en menor magnitud al de la sierra, ya que sólo los campesinos que aprecian sus cualidades la siembran para autoconsumo, incluso no posee las características agronómicas y comerciales que le permita competir con otros productos de mayor consumo (Ejemplo: la papa), por ello el productor grande no le ve importancia en estudiar ni mejorar el cultivo. De esta manera, el cultivo está siendo olvidado o desplazado, perdiéndose como recurso que podría ser aprovechado en el futuro por su potencial en la alimentación ganadera y como fuente de genes.
Se han realizado caracterizaciones morfológicas preliminares de la colección en Cajamarca (Seminario & Seminario, 1995); sin embargo, los rasgos fenotípicos aislados no proporcionan evaluaciones minuciosas de la diversidad genética dado que los caracteres morfológicos pueden representar loci limitados dentro de todo genoma (Zhang et al., 1998); adicionalmente, los factores medioambientales influyen mucho en los resultados (Spooner et al., 1996), por lo que es conveniente añadir caracterizaciones a nivel bioquímico y/o molecular, que permitan definir con mayor certeza las relaciones de parentesco entre cultivares y silvestres. Para este estudio planteamos los objetivos de estudiar la diversidad genética a nivel genotípico mediante los marcadores RAPD y conocer la distribución de la variación genética a nivel intra e interregional.
MATERIALES Y MÉTODOS
Material Vegetal
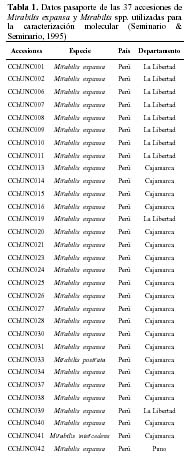
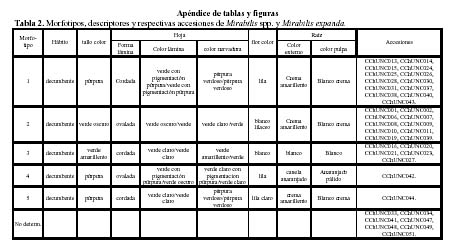
Se analizaron 37 accesiones pertenecientes al Banco de Germoplasma del Programa de Biodiversidad de Raíces y Tubérculos Andinos de la Universidad Nacional de Cajamarca. Según los datos pasaporte proporcionados por Seminario (Tabla 1), 1995. 24 accesiones pertenecen al departamento de Cajamarca, 12 a La Libertad y una accesión a Puno. La accesión CCHuNC033 es Mirabilis postrata (Ruiz & Pavón) Heimerl y la accesión CCHuNC041 Mirabilis intercedens Heimerl son parientes silvestres del M. expansa; la accesión CCHuNC034, es una M. expansa silvestre; las accesiones CCHuNC047, CCHuNC048, CCHuNC049, CCHuNC051 no han sido tipificadas en ningún morfotipo (Tabla 5), siendo agrupadas entonces como Mirabilis spp.
Extracción del ADN genómico
Se modificó el protocolo de Tai & Tanskley (1990) utilizando una concentración mayor de NaCl y sin nitrógeno liquido, para la extracción de ADN de muestras de hojas jóvenes desecadas, tomadas al azar de 3 individuos. Se priorizó la obtención de una mayor calidad y pureza de ADN, basándose en el índice de pureza (Absorbancia a 260 nm / Absorbancia a 280 nm: A260/A280) cercano al 1.8 – 2.0 y la corrida electroforética para verificar la calidad del ADN. Sin embargo, las muestras aún presentaron gran cantidad de poli fenoles, proteínas y ADN degradado. Se realizaron las medidas a través de un espectrofotómetro Beckman UV-VIS y se diluyeron las soluciones stock de ADN hasta una concentración de 9 a 12 ng/ul.
Ensayos RAPD
Se estandarizó en el laboratorio la técnica RAPD, Williams et al. (1990) y Welsh & McClelland (1990); y se ensayaron 19 iniciadores RAPD decaméricos (Operon Technologies Inc.), usando 4 accesiones tomadas al azar.
Todas las pruebas PCR se realizaron en un termociclador Perkin Elmer 9700 en un volumen total de 15 ul de mezcla de reacción compuesto por: buffer de actividad 1X, MgCl2 3.5 mM, dNTPs Sigma® 0.25 mM, iniciador 1 uM, ADN molde 9-12 ng, Bovine Serum Albumin Sigma® (BSA) 0.1 ug/ul, Taq polimerasa (Perkin Elmer®) 1 unidad, agua libre de nucleasas c.s.p.; adicionando 20 ul de aceite mineral para evitar la excesiva evaporación. El programa de PCR usado comprendió un ciclo inicial de 94°C x 1 minuto; luego 3 ciclos de (94°C x 15 segundos, 35°C x 15 segundos, 72°C x 75 segundos); seguido de 40 ciclos de (94°C x 15 segundos, 40°C x 15 segundos, 72°C x 75 segundos); 1 ciclo de 72°C x 7 minutos y finalmente 1 ciclo de 4°C.
Las corridas electroforéticas se realizaron en geles de agarosa al 2.5%, posteriormente teñidas con bromuro de etidio 1% por 10 minutos. La visualización se realizó en un transiluminador y mediante una cámara digital se guardaron las imágenes en archivos gráficos. Estos fueron analizados mediante inspección visualasignando valores de 1 (uno) a la presencia de banda y 0 (cero) a la ausencia. Las bandas de presencia dudosa se le asignaron el valor de 9 (nueve). Se asume que el patrón de todas las bandas muestra dominancia, es decir, la presencia de la banda representa el genotipo homocigótico dominante y heterocigótico, y la ausencia corresponde al genotipo recesivo.
Análisis de los datos
Se determinó el índice de contenido polimórfico PIC (Senior et al., 1998) para cada marcador. Los PIC fueron sumados para cada marcador RAPD generado obteniéndose el Índice de iniciador RAPD (Ghislain et al., 1999).
Para el análisis de datos se ingresaron éstos a una matriz básica de datos con el paquete estadístico NTSYS v2.1p y se corrió la opción de análisis multivariado, obteniendo una matriz de similitud utilizando el Coeficiente de Concordancia Simple o Simple Matching Coeficient (SMC) (Crisci & López, 1983). Para el análisis de agrupamientos se utilizó el programa NTSyspc v2.1p, basado en el UPGMA (Unweighted Pair Group Method using Arithmetic Averages), con el objetivo de obtener el dendograma. Para estimar la variancia de los componentes interregional e intra regional se utilizó el análisis molecular de variancia o AMOVA (Zhang et al., 1998).
RESULTADOS Y DISCUSIÓN
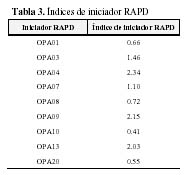
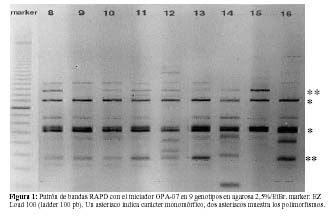
De la prueba con los 19 iniciadores RAPD finalmente se seleccionaron solo 9 considerando los que produjeron fragmentos de ADN bien definidos y de intensidad considerable, estos fueron: OPA01 5-CAGGCCCTTC-3, OPA03 5-AGTCAGCCAC-3, OPA04 5-AATCGGGCTG-3, OPA07 5-GAAACGGGTG-3, OPA08 5-GTGACGTAGG-3, OPA09 5-GGGTAACGCC-3, OPA10 5-GTGATCGCAG-3, OPA13 5-CAGCACCCAC-3, OPA20 5-GTTGCGATCC-3; con los cuales se obtuvieron 60 marcadores polimórficos, algunos de los cuales se aprecian en la Figura 1. La Tabla 3 contiene los índices de iniciadores y se observa que los valores más altos los tienen los iniciadores OPA04, OPA09 y OPA13, pudiendo sugerir su gran valor para futuras investigaciones en identificación genotípica de M. expansa y Mirabilis spp.
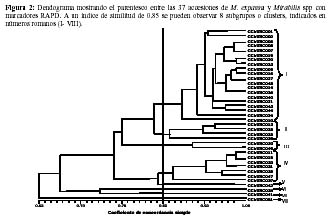
Al realizar el análisis de similaridad se comparó el dendograma basado en el coeficiente de concordancia simple o Simple Matching con el obtenido con el coeficiente Jaccard; aunque la topología de ambos árboles resultantes es similar, se escogió el primero debido a que tiene mayor índice de similitud que el segundo y, se ajusta más a los datos pasaporte y morfológicos; define una mayor similaridad entre las accesiones y ;finalmente, considera a la accesión CCHuNC042 como una OTU separada. Además, se consideraron a las accesiones con un índice de similitud mayor (con respecto al coeficiente de Jaccard) por los siguientes factores: provienen de una pequeña área geográfica de colecta, la cual se circunscribe a localidades de Cajamarca, La Libertad y una de Puno; este material genético ha estado sometido a erosión genética (Seminario, 1993; Franco & Uceda, 1996) como consecuencia hay una pérdida de variabilidad genética. En relación a su sistema reproductor, el cultivo es principalmente autógamo (Valderrama et al., 1999); además su propagación es principalmente clonal y escasamente por semilla, lo cual no permite un incremento de la variabilidad genética. Sobre las accesiones silvestres no se cuenta con mucha información acerca de su forma de propagación, pero es muy posible que sea a través de semillas, y esto implica una mayor base genética en este material. Hay pocos morfotipos (cinco hasta el momento) descritos para los cultivares de M. expansa (Seminario & Seminario, 1995), sugiriendo que la variabilidad a nivel de genes cualitativos es poca, explicándose también por el estado primitivo de este cultivo.
A un coeficiente de 1 se observan 31 grupos. Esto indicaría un 16.216 % de posibles duplicados en el Banco de Germoplasma. Por lo tanto, las accesiones duplicadas serían CChUNC006 = CChUNC008 CChUNC007 = CChUNC019, CChUNC027 = CChUNC048, CChUNC014 = CChUNC026 = CChUNC040, CChUNC015 = CChUNC030.
Entre los posibles duplicados encontrados el denominador común es que la mayoría pertenece al mismo grupo morfológico (Tabla 2, en apéndice), a excepción del par CChUNC027 = CChUNC048, siendo éste ultimo aun no identificado a nivel de especie ni de morfotipo. A diferencia del Cluster II, cuyos elementos comparten el mismo morfotipo y provienen del departamento de Cajamarca, los grupos de accesiones restantes formadas en el dendograma no guardan estricta relación con los datos geográficos o los datos morfológicos (Tabla 2).
Al tomar un índice de similitud de 0.85 en el dendograma (Figura 2), se pueden observar 8 clusters cuyos elementos, en la mayoría de los casos no comparten características morfológicas entre sí; lo cual sugiere una mayor variación a nivel molecular entre las accesiones. Las accesiones CChUNC001, CChUNC002, CChUNC006, CChUNC007, CChUNC008, CChUNC009, CChUNC010, CChUNC011, CChUNC019, CChUNC039 son identificadas en el mismo morfotipo, pero a nivel molecular aún presentan variabilidad. Lo mismo ocurre con las accesiones CChUNC013, CChUNC014, CChUNC015, CChUNC025, CChUNC026, CChUNC028, CChUNC030, CChUNC031, CChUNC037, CChUNC038, CChUNC040,.
Las accesiones, CChUNC016, CChUNC020, CChUNC021, CChUNC023 y CChUNC027, pertenecientes al morfotipo 3, se agrupan junto con accesiones del morfotipo 2 en el Cluster I. La accesión silvestre CChUNC034 no pertenece a ningún cluster. CChUNC033, Mirabilis postrata y CChUNC049, Mirabilis spp. forman el cluster III.
La accesión CChUNC047 considerada como Mirabilis spp. (procedente de Cajamarca) está agrupado en el cluster IV junto con otras M. expansa (por ejemplo, con CChUNC039 de La Libertad) sugiriendo que la similitud genética puede no tener relación con la zona de cultivo y se presumiría que podría pertenecer a la especie M. expansa.
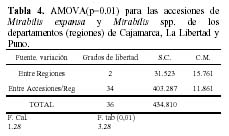
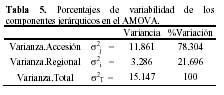
El Análisis Molecular de variancia (AMOVA), usando p=0.01, mediante el cual se desagregaron 2 componentes de variación: interregional e ínter accesiones por región, mostró valores de 21.69% y 78.3%, respectivamente y se exhiben en las Tablas 4 y 5. Ambos valores son medios y altos en relación a trabajos parecidos (Zhang et.al., 1998), por lo que se recomienda hacer énfasis en las colectas a nivel intra e interregional, ya que la participación de ambos componentes es importante por incrementar la variabilidad de la colección. Sin embargo, la variación interregional obtenida en la presente trabajo, tiene el inconveniente de tener en la región Puno solo una accesión, que podría generar un sesgo estadístico en el análisis.
CONCLUSIONES
Se obtuvo un porcentaje de individuos posiblemente duplicados de aproximadamente 16% en la colección estudiada.
No se apreció correspondencia entre los datos de la caracterización morfológica (fenotípica) y los datos de la caracterización molecular.
Se puede considerar el análisis molecular para definir la identidad taxonómica de especie en aquellas accesiones aun desconocidas o de identificación morfológica ambigua.
La variación intrarregional fue más significativa, posiblemente explicada por la reproducción sexual del cultivo.
AGRADECIMIENTOS
Trabajo posible gracias al financiamiento del Conseuil de la InterUniversitaire Communauté Française (CIUF) de Bélgica.
Agradecemos toda la colaboración de los miembros del Instituto de Biotecnología de la Universidad Nacional Agraria La Molina.
Agradecemos al Ing. Miguel Valderrama (Cajamarca) por su ayuda y su aporte en la realización del trabajo.
LITERATURA CITADA
Crisci J. & López M. 1983. Introducción a la teoría y práctica de la taxonomía numérica. Serie n°26 OEA Washington D.C. [ Links ]
Franco P. & Uceda J. 1996. El Chago o Yuca Inca Mirabilis expansa: Raíz andina en peligro de extinción. Informe Técnico – Estación experimental y forestal Baños del Inca n°1. [ Links ]
Ghislain M., Dapeng Z., Fajardo D., Huaman Z. & Hijmans R.J. 1999. Marker assisted sampling of the cultivated Andean potato Solanum phureja collection using RAPD markers. Genetic Resources and Crop Evolution. 46: 547-555. [ Links ]
Seminario C.J. 1993. Aspectos etnobotánicos y morfológicos del chago, miso o mauca (Mirabilis expansa R. y P.) en el Perú. Boletin de Lima. 15(86): 71-79. [ Links ]
Seminario C.J. & Seminario A. 1995. Colección Regional de Germoplasma de Raíces Andinas. Mimeografiado. Universidad Nacional de Cajamarca. Programa Colaborativo Biodiversidad de Raíces y Tubérculos Andinos (Convenio CIP-COTESU). Cajamarca. [ Links ]
Senior M.L., Murphy J.P., Murphy M.M. & Struber C.W. 1998. Utility of SSR for determining genetic similarities and relationships in Maize using an Agarose gel system. Crop Science. 38: 1088-1098. [ Links ]
Spooner D. M., Tivang J., Nienhuis J., Miller J. T., Douches D.S. & Contreras-M. A. 1996. Comparisons of four molecular markers in measuring relationships among the wild potato relatives Solanum section Etuberosum (subgenus Potatoe). Theoretical and Applied Genetics .92: 532-540. [ Links ]
Tai T.H. & Tanksley S.D. 1990. A rapid and inexpensive method for isolation of total DNA from dehydrated plant tissue. Plant Molecular Biology Reporter .8(4): 297-303. [ Links ]
Valderrama M., Seminario J. & Cabanillas J. 1999. Estudio de la Biología Floral de cuatro cultivares y tres parientes silvestres de chago; Mirabilis expansa (R. y P.) Standley. Articulo Científico. Universidad Nacional de Cajamarca. [ Links ]
Vivanco J.M. 1999. Studies on the Biochemistry and Physiology of root-specific ribosome inactivating proteins (RIPs) in Mirabilis expansa and related species.Tesis para optar por el grado de Doctor en Filosofia. Pennsilvania State University. EE.UU. [ Links ]
Welsh J. & McClelland M. 1990. Fingerprinting genomes using PCR with arbitrary primers. Nucleic Acids Research. 18: 7213-7218. [ Links ]
Williams J.G.K., Kubelik A.R., Livak K.J., Rafalski J.A. & Tingey S.V. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research. 18: 6531-6535. [ Links ]
Zhang D., Ghislain M., Huaman Z., Golmirzaie A. & Hijmans R. 1998. RAPD variation in sweetpotato (Ipomoea batatas L.) cultivars from South America and Papua New Guinea. Genetic Resources and Crop Evolution. 45: 271-277. [ Links ]
Presentado: 21/07/2006
Aceptado: 25/09/2006
Apéndice