Introducción
El tarwi (Lupinus mutabilis Sweet) es una leguminosa originaria de la sierra de Bolivia, Ecuador y Perú (Eastwood et al., 2008). En los últimos años, se ha incrementado el interés hacia el lupino como alternativa al consumo de soya, principalmente la de origen transgénico, debido a su alto contenido de proteínas y ácidos grasos (Carvajal-Larenas et al., 2016). Además, es considerado un cultivo muy beneficioso para la agricultura en el altiplano porque sus raíces fijan una alta cantidad de nitrógeno atmosférico (400 kg/ha), mejorando así la fertilidad del suelo (Baldomero, 2015); se adapta a diferentes climas de los Andes, encontrándose incluso campos de tarwi por debajo de los -4°C (Tapia, 2015) y tiene una gran tolerancia al estrés hídrico, similar a la de la quinua (Canahua & Roman, 2016). Sin embargo, su consumo está siendo disminuido paulatinamente en los países andinos, no sólo por falta de difusión de las formas de uso, sino, también, por el desinterés de las instituciones encargadas de promoverlo (Jacobsen & Mujica, 2006).
El suelo es un ecosistema con una gran variedad de microorganismos benéficos. En el caso de las bacterias, a aquellas que son beneficiosas para el crecimiento vegetal y son de vida libre se les ha denominado Plant Growth Promoting Rhizobacteria o, por sus siglas en inglés, PGPR (Kloepper, 1993). Éstas influyen directamente en el desarrollo vegetal, a través de la síntesis de distintos compuestos o, indirectamente, evitando el ataque de organismos patógenos.
Múltiples estudios han demostrado la presencia de microorganismos endófitos en interacción con los nódulos del género Lupinus, tales como Micromonospora, actinobacteria fijadora coexistiendo con Bradyrhizobium (Trujillo et al., 2007); Paenibacillus lupini (Carro et al., 2014) y Cohnella lupini (Flores-Félix et al., 2014) en Lupinus albus; Bosea lupini, alfa proteobacteria asociada con L. polyphyllus (De Meyer & Willems, 2012); Kribbella lupini, Micromonospora lupini y Micromonospora saelicesensis, actinobacterias aisladas a partir de nódulos de Lupinus angustifolius (Trujillo et al., 2006a, 2010). La ocurrencia de estas bacterias en nódulos de Lupinus, y la carencia de reportes dirigidos al estudio de la interrelación microorganismo-planta con Lupinus mutabilis, nos lleva a plantear la necesidad de conocer el estado de la microbiota presente en el tarwi, la cual puede jugar un papel importante en la promoción de crecimiento vegetal.
El objetivo de esta investigación fue aislar cepas de rizósfera, raíces, tallos y nódulos de Lupinus mutabilis y caracterizarlas por sus capacidades PGPR. Del mismo modo planteó identificar molecularmente a los aislamientos con mejores capacidades y evaluar la influencia de ellos en plántulas de Lupinus mutabilis bajo condiciones de invernadero.
Materiales y Métodos
Muestreo y desinfección de material vegetal
Los plantones de Lupinus mutabilis (Sweet) se colectaron de dos campos pertenecientes a la Universidad Nacional Agraria La Molina: el IRD-Sierra (11°51’22” S 75°23’38” W) y al Programa de Cereales y Granos Nativos (12°04’40.8” S 76°56’37.7” W).
Para extraer los plantones se delimitaron superficies cuadradas de 20x20 cm2 en cada base y se excavaron hasta obtener la raíz completa (~50 cm). El suelo no rizosférico fue separado agitando moderadamente la muestra. Luego, se recolectó el suelo más próximo a las raíces en bolsas estériles, con la ayuda de una brocha desinfectada en alcohol 96°. Se colectaron también 10 raíces, 10 tallos y 10 nódulos por planta, los cuales fueron lavados con agua potable y colocados en recipientes con agua estéril (Pérez et al., 2010). La desinfección superficial consistió en un enjuague de 12 horas con agua estéril, un remojo de 2 minutos en solución de bicloruro de mercurio (HgCl2) al 2.5% y 5 enjuagues en agua destilada estéril para remover todo el bicloruro de la superficie (Trujillo et al., 2006b). Se tomaron al azar 5 raíces, 5 tallos y 5 nódulos que fueron transferidos a una placa con medio agar nutritivo para confirmar la ausencia de microorganismos en la superficie.
Aislamiento de microorganismos
Se tomaron 10 g de suelo rizosférico que fueron transferidos a un matraz con 90 ml de solución salina 0.85% (León, 2014) agitándose de manera vigorosa (dilución 10-1). Los tallos y raíces desinfectados fueron cortados en secciones de 4-5 cm y macerados en 30 ml de solución salina 0.85% mientras que los nódulos fueron triturados con 1 ml de solución salina 0.85% (Claros & Ortuño, 2013). En un eppendorf estéril se recuperó 1 ml del macerado de cada muestra. Se realizaron diluciones seriales para cada muestra y se inocularon 0.1 ml de las diluciones 10-4 a 10-6 en medio AN (agar nutritivo) para bacterias, y medio OGY (agar oxitetraciclina-glucosa-extracto de levadura) suplementado con cloranfenicol 150 ppm, para hongos.
Antagonismo frente a un patógeno
Las pruebas de enfrentamiento con el patógeno se realizaron mediante la técnica de cultivo dual in vitro (Fernández & Suárez, 2009; Martínez et al., 2014). Se cortaron fragmentos de 1 cm2 del micelio de los aislamientos, previamente sembrados en medio PDA (agar papa-dextrosa) e incubados a 28 °C durante 4 días. Cada fragmento fue colocado en un extremo de la placa Petri y se incubó en oscuridad durante 12 horas. Transcurrido ese tiempo, se colocó, en el otro extremo de la placa, un fragmento de 1 cm2 del micelio de la cepa patógena (Fusarium oxysporum), que también fue sembrada previamente en medio PDA a 28 °C durante 4 días (Zúñiga, 2007). Como control se sembró en medio PDA únicamente al hongo patógeno, considerando su radio como “crecimiento libre (CL)”. Las cepas que inhibieron el crecimiento del micelio fueron consideradas como positivas y se midió el radio del hongo, considerándolo como “crecimiento influenciado (CI)”.
El porcentaje de inhibición se calculó mediante la Fórmula 1, descrita por Fernández & Suárez (2009).
% de inhibición = ((CL − CI)÷CL)×100
Fórmula 1. Porcentaje de inhibición.
Identificación microscópica
Sólo las que resultaron positivas para la prueba de antagonismo fueron identificadas mediante microscopía hasta nivel de género. Para ello, fueron sembradas en cámara húmeda con medio de cultivo OGY suplementado con cloranfenicol 150 ppm, se incubaron durante 5 días y se tiñeron las estructuras fúngicas con azul de metileno. Cada preparación fue observada en un microscopio óptico a 40X y la identificación se basó en claves taxonómicas de la base de datos online MycoBank (2016).
Producción de ácido indol acético (AIA)
Esta prueba fue un tamizaje para reducir la cantidad de aislamientos bacterianos provenientes de raíces, tallos y rizósfera, aquellos que resultaron negativos o con baja producción de AIA fueron descartados.
La técnica colorimétrica utilizada fue la descrita por (Gordon & Weber, 1950). Se inocularon las cepas bacterianas a una concentración aproximada de 108 ufc·ml-1 en tubos con 4 ml de medio LMC (caldo manitol-levadura) suplementado con L-triptófano 1 g·l-1 (Marca CDH). El control positivo consistió en LMC con adición de AIA 40 mg·l-1 (Marca CDH) y el control negativo únicamente en LMC. Los tubos se incubaron en oscuridad a 28 °C durante 5 días. Culminado el tiempo se centrifugaron a 4 000 rpm durante 15 min (Clavijo et al., 2012). La reacción se realizó en microplacas con una proporción muestra:reactivo de 1:2 (Lara et al., 2011): 100 μl del sobrenadante con 200 μl del reactivo de Salkowsky (FeCl3, agua destilada y HSO4 en proporción volumétrica 1:50:30), y se incubó 30 minutos a temperatura ambiente, luego se evaluó el viraje de color de la solución a tonalidades rojizas. Se consideró tres niveles cualitativos de producción: nivel bajo (+), nivel medio (++) y nivel alto (+++).
Solubilización de fosfato tricálcico
Los aislados fueron sembrados en medio NBRIP (National Botanical Research Institute’s Phosphate, composición para 1 l : 10 g glucosa, 5 g Ca3(PO4)2, 5 g MgCl2.6H2O, 0.25 g MgSO4.7H2O, 0.2 g KCl, 0.1 g (NH4)2SO4,) e incubados a 28 °C durante 5 días. La positividad de la prueba se evidenció por la presencia de un halo transparente alrededor de las cepas (Nautiyal, 1999). Se midió el diámetro del halo hasta observar un valor constante y se calculó la ERS (eficiencia relativa de solubilización) mediante la Fórmula 2, en donde DH es el Diámetro del halo y DC el Diámetro de la colonia. Este índice indica el radio de acción de la bacteria sobre el sustrato en relación al crecimiento de la colonia, expresado en “número de veces mayor a”.
ERS=DH÷DC
Fórmula 2. Eficiencia Relativa de Solubilización.
Antagonismo frente a un patógeno
pruebas de enfrentamiento con el patógeno se realizaron siguiendo el protocolo descrito anteriormente para hongos, con la diferencia de que las cepas bacterianas se sembraron inicialmente en medio PDA y se incubaron a 28 °C durante 24 horas. El porcentaje de inhibición se calculó mediante la Fórmula 1.
Producción de sideróforos
La producción de sideróforos se determinó siguiendo el método descrito por Schwyn & Neilands (1987). Las cepas bacterianas fueron sembradas en medio CAS (Agar Cromoazurol Sulfonato; composición para 1 l de base: 20 g proteasa, 1.5 g MgSO4.7H2O, 1.5 K2HPO4, composición para 100 ml de tinte azul: 0.06 g CAS en 50 ml de agua destilada, 0.0027 g FeCl3.6H2O en 10 ml de HCl 10 mM, 0.0073 g de CTAB en 40 ml de agua destilada) e incubadas a 28 °C durante 5 días. La formación de un halo naranja a amarillo alrededor de las colonias confirmó la producción de sideróforos. Se midió el diámetro de los halos a los 6 días de incubación y se evaluó la ERP (eficiencia relativa de producción) mediante la Fórmula 3, donde DP es el Diámetro de producción y DC es el Diámetro de crecimiento.
ERP=DP÷DC
Fórmula 3. Eficiencia Relativa de Producción.
Fijación de nitrógeno in vitro
Esta prueba se realizó únicamente en las cepas bacterianas aisladas de nódulos. Se las inoculó en tubos con 3 ml de LMC e incubadas a temperatura ambiente por 72 horas en agitación constante (200 rpm). Transcurrido el tiempo, se agregaron 9 ml de KCl 2M con agitación a 150 rpm durante 1 hora. Se dejó reposar una hora adicional y se tomó 10 ml del sobrenadante para centrifugarlos (3 000 rpm) durante 5 minutos. Los sobrenadantes se vertieron en tubos de dilución, se añadieron 0.4 ml de solución alcohólica de fenol 10%, 0.4 ml de nitroprusiato de sodio al 0.5% y 1 ml de solución oxidante, reposando por 1 hora. La positividad de la fijación de nitrógeno in vitro se observó mediante una coloración azul la cual se mensuró mediante absorbancia en espectrofotómetro (UV-VIS GenesysTM 10 Bio, THERMO) a 633 nm. Las concentraciones de nitrógeno fijado como amonio fueron calculadas en una recta patrón obtenida de diluciones sucesivas a partir de una solución madre de 100 ppm de cloruro de amonio (Lara et al., 2007).
Identificación de aislamientos
Los aislamientos bacterianos que tuvieron un potencial como promotores de crecimiento fueron identificados en base a la región 16S del ADNr. Para la extracción del material genético se siguió el protocolo de Wilson (1997) y se diluyó el ADN extraído en agua estéril Mili-Q antes de continuar con la amplificación. Los fragmentos de la región 16S del ADNr (aproximadamente 1500 pb) de cada bacteria fueron separados usando los primers universales descritos por Weisburg et al. (1991), fD1 (5’ - CCGAATTCGTCGACAACAGAGTTTGATCCTGGCTCAG - 3’) y rD1 (5’ - CCCGGGATCCAAGCTTAAGGAGGTGATCCAGCC - 3’). Las condiciones de reacción fueron: buffer de PCR + (NH4)2SO4 - MgCl2 al 1X, MgCl2 1.25 mM, mix de nucleótidos 0.2 mM, 12 pmoles de cada cebador, 1.25 U Taq polimerasa y 2.4 µl de muestra (dilución 1:100); se aplicó el programa: 3 minutos de precalentamiento a 93 °C, 30 ciclos de 45 segundos de desnaturalización a 93 °C, 45 segundos de hibridación del cebador a 62 °C, 2 minutos de elongación 72 °C, y 10 minutos de extensión final a 72 °C. Los productos de la amplificación fueron examinados mediante electroforesis, cargando 5 µl del amplificado en gel de agarosa al 1.5% con buffer de corrida TAE 1X, durante aproximadamente 2 horas a 80 V. Luego, fueron purificados utilizando el GeneJET PCR Purification kit (ThermoScientific, USA) y secuenciados por la empresa MacroGen©. La comparación de secuencias se realizó en la página web del Centro Internacional de la Información Biológica (NCBI) empleando el algoritmo BLAST (Altschul et al., 1990).
Inoculación en invernadero
El sustrato y el material vegetal se obtuvieron del banco de germoplasma de tarwi del Programa de Cereales y Granos Nativos - UNALM. Las semillas de tarwi fueron desinfectadas con Benomil 1g·l-1 y sembradas en sustrato estéril húmedo, mezcla de vermiculita y arena (1:2). A los 15 días, los brotes enraizados fueron trasladados a macetas de 3 kg con 2.5 kg de suelo. Luego de cinco días de crecimiento en maceta, se procedió con la inoculación. Para este estudio, se regaron las plantas inoculadas con solución nutritiva (Solución hidropónica La Molina A y B).
Se implementaron cuatro tratamientos para evaluar el efecto de las cepas en el crecimiento de L. mutabilis: un testigo, una co-inoculación y dos inoculaciones individuales. El testigo consistió únicamente en la plántula regada por solución nutritiva, la co-inoculación fue una mezcla de la cepa más productora de AIA y la cepa más solubilizadora de fosfato, y las inoculaciones individuales fueron una cepa endofítica y una rizosférica con el mejor resultado simultáneo en las pruebas PGPR, es decir que haya presentado los valores más altos en al menos dos pruebas PGPR. Se agregó a cada maceta 3 ml de inóculo a la concentración de 108 ufc·ml-1. Durante el ensayo se evaluó semanalmente la altura de la planta y a las seis semanas, se evaluó peso fresco y seco de la parte aérea y radicular de la planta.
Para el análisis, se utilizó el Diseño Completo al Azar (DCA) con cuatro tratamientos y 10 repeticiones. Los datos fueron analizados utilizando el análisis de varianza ANOVA y la prueba comparativa Diferencia Mínima Significativa (P ≤ 0.05) empleando el software estadístico R.
Resultados y discusión
Aislamiento de microorganismos
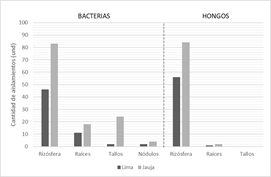
Se obtuvieron en total 333 cultivos puros, de los cuales 190 eran de bacterias y 143 de hongos (Figura 1). La mayoría de los aislados provenían de la rizósfera (80.8%, 269), sólo un 19.2% (64) eran potenciales endófitos de tarwi porque fueron encontrados dentro de tallos, raíces y nódulos. Al ser la rizósfera una zona de interacción con la planta, los microorganismos encuentran en ella un ambiente favorable con nutrientes orgánicos provenientes de las raíces, y esto conlleva a que la concentración de microorganismos en la rizósfera generalmente sea mucho mayor que en el resto del suelo (Glick, 1995). Este resultado es comparable con estudios como el de Angulo et al. (2014) y Ayubb et al. (2017), quienes también obtuvieron mayor cantidad de aislados rizosféricos que de endófitos a partir de Eucalyptus nitens y Oriza sativa, respectivamente.
Por otro lado, los factores ambientales y edáficos influyen sobre la presencia y distribución de microorganismos en el suelo (Dick, 1992). La actividad microbiana está fuertemente determinada por el potencial hídrico, cuando éste alcanza valores muy negativos la actividad microbiana cesa (Jenkinson, 1992); la estructura, porosidad, pH y potencial redox, son factores que en su conjunto actúan de un modo u otro para influir sobre la densidad y actividad microbiana (IGAC, 1993); e incluso las prácticas de cultivo o la edad de la planta ejercen efectos sobre las poblaciones microbiales. En este proyecto, el 64.6% (215) de los aislamientos pertenecía al IRD-Sierra, en Jauja, por lo cual se podría inferir que las condiciones ambientales y edáficas presentes en los campos de Jauja fueron más favorables para la presencia de microorganismos, pero esto requiere un estudio a profundidad para poder confirmarlo.
Antagonismo frente a un patógeno e identificación de cepas
De un total de 143 aislados, sólo 9 inhibieron el crecimiento de Fusarium oxysporum. Los porcentajes de inhibición de estas nueve cepas se observan en la Tabla 1.
Estas cepas antagonistas fueron las únicas que continuaron con el proceso de identificación a nivel de género mediante microscopía. Así se observó que cinco de ellas pertenecían al género Aspergillus, dos a Penicillium y las dos restantes no pudieron ser identificadas por falta de estructuras vegetativas observables. Para cada género, se identificaron hasta nivel de especie las cepas más antagonistas (Tabla 1, Figura 2): N°134 para Aspergillus y N°96 para Penicillium.
Tabla 1. Porcentaje de inhibición de Fusarium oxysporum por las cepas fúngicas.
| N° Cepa | Porcentaje de inhibición | Género más cercano |
|---|---|---|
| 40 | 60.0 | No identificado |
| 43 | 57.1 | No identificado |
| 47 | 46.1 | Aspergillus sp. |
| 96 | 64.0 | Penicillium citrinum |
| 99 | 61.0 | Penicillium sp. |
| 103 | 57.1 | Aspergillus sp. |
| 105 | 38.5 | Aspergillus sp. |
| 131 | 70.0 | Aspergillus sp. |
| 134 | 74.0 | Aspergillus terreus |
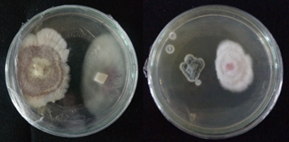
Figura 2 Aspergillus (Izquierda) y Penicillium (Derecha) en interacción dual con Fusarium oxysporum.
Las características macro y microscópicas de la cepa N°134 se muestran en la Figura 3. Se observa que esta cepa es de textura aterciopelada-arenosa, es de color marrón en el centro y blanco en los bordes cuando es joven, pero totalmente marrón cuando crece. Llega a medir 4 cm de diámetro a los seis días de incubación en agar OGY y presenta un ligero pigmento amarillo en el medio. A 100X, se observan un conidióforo con vesícula globosa y cabeza conidial en forma de abanico levemente columnar, conidios globosos, ausencia de ascas y ascosporas. Estas características concuerdan con un estudio de hongos filamentosos en suelo de páramo (Arias & Piñeros, 2008), donde se describen a las colonias de Aspergillus terreus como aterciopeladas, de color rosado parduzco que alcanzan un diámetro de 3.5- 5 cm en 7 días, con exudado incoloro y producción de un pigmento amarillo difusible en el medio. Además, comparando nuestros datos con las claves taxonómicas descritas por Gómez (1951) y Piontelli (2008), se puede afirmar que la cepa Nº 134 es identificada como Aspergillus terreus.
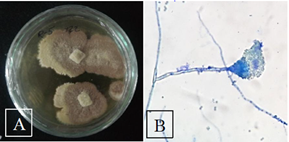
Figura 3 Colonia de cepa N°134 en medio OGY (Izquierda) y estructura vegetativa observada a 100X (Derecha).
Las características macro y microscópicas de la cepa N° 96 se muestran en la Figura 4. Se observan colonias puntiformes de textura aterciopelada, color verde con bordes blancos. A 100X e observaron conidios redondos, unicelulares formando cadenas no ramificadas. Para poder identificarla hasta nivel de especie, esta cepa fue analizada en el departamento de Fitopatología de la UNALM, determinándose que se trataba de la especie Penicillium citrinum.
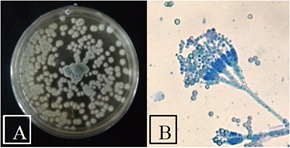
Figura 4 Colonia de cepa N° 96 en medio OGY (Izquierda) y estructura vegetativa observada a 100X (Derecha).
Aspergillus terreus ha sido encontrado como antagonista de Fusarium oxysporum en estudios anteriores: Dawood & Mohamed (2015) estudiaron tres aislamientos de A. terreus como control biológico, los cuales inhibieron hasta un 89% el crecimiento de F. oxysporum por interacción dual; Bashar & Chakma (2014) evaluaron colonias de A. terreus que inhibieron a F. oxysporum mediante interacción dual y producción de metabolitos no volátiles en un 53.33% y 83.33%, respectivamente. Estos trabajos respaldan lo encontrado en nuestra cepa de A. terreus, pues llegó a inhibir el crecimiento del patógeno en 74%, valor similar a lo descrito anteriormente. Quiroz et al. (2008) indica que Aspergillus muestra mecanismos antagónicos de competencia por espacio y producción de sustancias antibióticas denotadas por el cambio de coloración en el medio. Se puede inferir que A. terreus presenta estos dos tipos de acción frente al patógeno.
Penicillium citrinum ha sido catalogado como un patógeno indefinido, es decir, un microorganismo que, sin ser considerado normalmente como fitopatógeno, puede llegar a inhibir el crecimiento de la planta (Hyakumachi & Kubota, 2004). Sin embargo, científicos han evaluado también la capacidad antagónica de este hongo. Alwathnani et al. (2012), evaluaron el potencial de control biológico de algunos antagonistas locales frente a F. oxysporum f. sp. phaseoli y sus resultados del ensayo in vitro revelaron que P. citrinum inhibió el crecimiento del patógeno en un 59%. Junto a otras pruebas, concluyen que este hongo tiene gran potencial de ser usado como agente de control biológico. Dwivedi & Enespa (2013) obtuvieron también resultados similares, a través de la técnica “poisoned food” observaron que P. citrinum inhibió el crecimiento de F. oxysporum f. sp. lycopersici en un 67.54%. Estos resultados respaldan lo observado en nuestro estudio: la inhibición del patógeno en un 64% por parte de P. citrinum.
Es importante conocer que los hongos que presentan actividad antagonista in vitro, pueden no mostrar resultados similares in situ debido a las condiciones ambientales y la competencia con otros microorganismos (Alwathnani et al., 2012).
Capacidades de promoción de crecimiento vegetal e identificación de cepas
La prueba de producción de AIA fue un tamizaje para reducir los 184 aislamientos obtenidos de raíces, tallos y rizósfera. Fueron seleccionados 36, por mostrar resultados positivos en la producción de indol y con estas cepas se realizaron las siguientes pruebas PGPR donde: 8 mostraron capacidad para solubilizar fosfatos tricálcicos, 10 mostraron capacidad antagónica contra Fusarium oxysporum y 35 produjeron sideróforos. Se descartaron aquellas cepas que no solubilizaron fosfato tricálcico ni mostraron antagonismo contra el patógeno y cuya producción de sideróforos (ERP) fue menor a 2. La fijación in vitro de nitrógeno se realizó sólo en las 6 cepas provenientes de nódulos y todas resultaron positivas, con una fijación en el rango de 4.6 - 8.5 mg/l NH4 +. En la Tabla 2, se observan los resultados de las pruebas PGPR de estos aislamientos.
Tabla 2 Capacidades de promoción de crecimiento vegetal en los aislados bacterianos.
| Código | Producción de AIA | Solubilización de fosfato tricálcico (ERS) | Antagonismo contra F. oxysporum (% inhibición) | Producción de sideróforos (ERP) | Fijación de nitrógeno (mg/l NH4+) | Especie más cercana |
|---|---|---|---|---|---|---|
| RB01 | +++ | 4.62 | - | 1.22 | n.d. | Achromobacter marplatensis |
| RB02 | ++ | 3.82 | - | 2.91 | n.d. | Bacillus toyonensis |
| RB03 | ++ | - | 23.24 | 2.27 | n.d. | |
| RB05 | ++ | - | 17.01 | 3.54 | n.d. | |
| RB06 | ++ | - | - | 2.50 | n.d. | |
| RB08 | ++ | 3.47 | 10.79 | 5.00 | n.d. | Lysinibacillus macroides |
| RB10 | ++ | 4.97 | 29.05 | 1.48 | n.d. | Bacillus circulans |
| RB11 | ++ | - | 49.38 | 1.68 | n.d. | |
| RB13 | ++ | - | - | 4.75 | n.d. | |
| RB14 | +++ | 1.72 | - | 3.18 | n.d. | Bacillus cereus |
| RB15 | ++ | - | 28.22 | 1.64 | n.d. | |
| RB16 | ++ | - | - | 2.62 | n.d. | |
| RB18 | ++ | - | - | 2.13 | n.d. | |
| RB19 | ++ | - | - | 2.16 | n.d. | |
| RB21 | ++ | - | - | 2.11 | n.d. | |
| RB22 | ++ | 1.98 | - | 1.76 | n.d. | Paenibacillus polymyxa |
| RB23 | ++ | - | 46.06 | 1.17 | n.d. | |
| RB24 | ++ | - | 48.13 | 1.09 | n.d. | |
| RB26 | ++ | - | 33.2 | 1.20 | n.d. | |
| RAB02 | ++ | 36.51 | - | n.d. | ||
| TB01 | ++ | 2.51 | - | 1.10 | n.d. | Enterococcus mundtii |
| TB04 | ++ | - | - | 4.17 | n.d. | |
| TB05 | +++ | 2.90 | - | 1.39 | n.d. | Lysinibacillus macroides |
| NR01 | n.d. | n.d. | n.d. | n.d. | 8.47 | Bacillus cereus |
| NR02 | n.d. | n.d. | n.d. | n.d. | 5.79 | Bacillus pumilus |
| NR03 | n.d. | n.d. | n.d. | n.d. | 4.60 | Bacillus aryabhattai |
| NR04 | n.d. | n.d. | n.d. | n.d. | 4.57 | Bacillus licheniformis |
| NR05 | n.d. | n.d. | n.d. | n.d. | 6.02 | Bacillus pumilus |
| NR06 | n.d. | n.d. | n.d. | n.d. | 8.04 | Bacillus liqueniformis |
| n.d.: No determinado, referido a una prueba no realizada. |
La identificación molecular se realizó en aquellos aislados que presentaron la producción de AIA y solubilización de fosfatos simultáneamente (8 aislados), y a todas las cepas de nódulos (6 aislados). El análisis comparativo de las secuencias de la región 16S del ADNr obtenidas con aquellas registradas en la base de datos de NCBI mostró que de los 14 aislados, 9 pertenecían al género Bacillus, 2 al género Lysinibacillus, 1 al género Paenibacillus, 1 al género Enterococcus y 1 al género Achromobacter (Tabla 3).
Tabla 3 Identificación molecular de aislados bacterianos.
| Código de la cepa | Especie más cercana | Accesión | Porcentaje de similitud |
|---|---|---|---|
| RB01 | Achromobacte marplatensis CCUG 56371 | NR_118400.1 | 99 |
| RB02 | Bacillus toyonensis BCT 7112 | NR_121761.1 | 99 |
| RB08 | Lysinibacillus macroides LMG 18474 | NR_114920.1 | 99 |
| RB10 | Bacillus circulans NBRC 13626 | NR_112632.1 | 96 |
| RB14 | Bacillus cereus ATCC 14579 | NR_074540.1 | 99 |
| RB22 | Paenibacillus polymyxa IAM 13419 | NR_112117.1 | 99 |
| TB01 | Enterococcus mundtii NBRC 100490 | NR_113906.1 | 98 |
| TB05 | Lysinibacillus macroides LMG 18474 | NR_114920.1 | 100 |
| NR01 | Bacillus cereus ATCC 14579 | NR_074540.1 | 100 |
| NR02 | Bacillus pumilus NBRC 12092 | NR_112637.1 | 98 |
| NR03 | Bacillus aryabhattai B8W22 | NR_118442.1 | 98 |
| NR04 | Bacillus licheniformis DSM 13 | NR_118996.1 | 99 |
| NR05 | Bacillus pumilus NBRC 12092 | NR_112637.1 | 99 |
| NR06 | Bacillus licheniformis DSM 13 | NR_118996.1 | 95 |
La producción de ácido indol acético y sus beneficios en la promoción de crecimiento vegetal ha sido ampliamente reportada. Esta auxina es producida no sólo por plantas superiores, sino también por bacterias y hongos, lo cual posee un rol significativo en la interacción planta-microorganismo pues contribuye en la promoción del crecimiento radicular y foliar de la planta (Vega-Celedón et al., 2016). La solubilización de fosfatos y producción de sideróforos juegan también un rol importante en esta interacción porque hacen disponibles a los nutrientes poco solubles, aumentando la disponibilidad de éstos para que las plantas puedan asimilarlos. La cepa RB10 (Bacillus circulans) mostró la más alta capacidad solubilizadora de fosfato (Figura 5), logrando un 4.96 de eficiencia relativa de solubilización, es decir que la bacteria solubilizó cinco veces más que su crecimiento de colonia. Este valor fue superior a lo reportado por Ortiz (2013) en un estudio de bacterias aisladas de rizósfera de Lepidium meyenii (maca), donde se obtuvo una ERS máxima de 1.28 y en un estudio de rizobacterias de Phaseolus vulgaris (frijol), donde el máximo diámetro de solubilización fue 1 cm (Vílchez, 2017), en comparación a los 2.88 cm que alcanzó RB10. Bacillus circulans, como tal, es reportado también en un estudio de rizósfera de manzana donde mostró capacidades de solubilización de fosfato, producción de AIA, actividad de ACC desaminasa, actividad nitrogenasa, producción de sideróforos, entre otros (Mehta et al., 2015). El aislado RB11 fue el más antagonista frente a Fusarium oxysporum (Figura 5), pero logró sólo un 49.37% de inhibición, valor inferior a estudios de bacterias rizosféricas que lograron un 100% y 96% frente a Fusarium oxysporum en arroz (Trujillo et al., 2007) y vainilla (Adame et al., 2009), respectivamente. El aislado RB08 (Lysinibacillus macroides) fue el que produjo mayor cantidad de sideróforos (Figura 5), alcanzando un 5.00 de eficiencia relativa de producción y un halo de 4 cm, valor superior a lo reportado en un estudio de cepas de Azospirillum en frutilla, donde obtuvieron un máximo diámetro de producción de 2.38 cm (Tortora et al., 2007) y en un estudio bacterias PGPR en tomate, donde el máximo diámetro fue 1.7 cm (Sánchez, 2011). La fijación de nitrógeno más alta se observó para la cepa NR05 (Bacillus pumilus), con una concentración de 6 mg·l-1 de ion amonio, valor relativamente mayor al reportado para Azotobacter y Azospirillum - dos géneros comúnmente conocidos como fijadores de nitrógeno - que obtuvieron 5.2 y 4.6 mg.-1, respectivamente (Lara et al., 2007). La capacidad fijadora de Bacillus pumilus se demostró en un estudio en Eleusine coracana (Dheeman et al., 2019) donde se observó que esta bacteria tuvo un buen crecimiento hasta siete generaciones después en un medio Ashby libre de nitrógeno, lo que significa que utilizó nitrógeno atmosférico como fuente de nitrógeno para crecer.
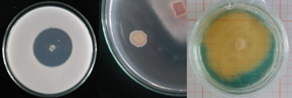
Figura 5 Cepa más solubilizadora de fosfato tricálcico, RB10 (Izquierda); cepa más antagonista de Fusarium oxysporum, RB11 (Centro); y cepa más productora de sideróforos, RB08 (Derecha).
Los géneros encontrados en esta investigación han sido reportados con anterioridad en trabajos como el de Liceta (2015) en sus aislamientos de rizósfera de quinua y por Barquero (2014) en sus aislados de raíces de alubia y pimiento. Especies de Paenibacillus fueron encontradas asociadas a rizósfera de diferentes cultivos (Coelho et al., 2008), Calvo & Zuñiga (2010) aislaron especies de Bacillus en rizósfera de papa y Oliva (2015) logró aislar especies de Bacillus en rizósfera de Lupinus mutabilis para demostrar su potencial como controlador biológico. En nuestros resultados se observó que cepas rizosféricas y endofíticas tuvieron la misma identificación en especie, tal es el caso de RB08 y TB05, identificadas como Lysinibacillus macroides, y RB14 con TB01, identificadas como Bacillus cereus. Esto puede ser debido a que la rizósfera - uno de los ecosistemas más diversos - se ha propuesto como una fuente de adquisición de endófitos para las plantas, ya que las grietas en los tejidos de la planta permiten a las bacterias rizosféricas penetrar y colonizar tejidos internos (Sørensen & Sessitsch, 2007). En este trabajo, todas las cepas aisladas de nódulos se identificaron como Bacillus, la presencia de este género en nódulos se debe a que generalmente Bacillus y Paenibacillus son endofíticos de nódulos (Costa et al., 2013). Por otro lado, si bien el género Bacillus no es reconocido como una bacteria que nodula, sino más como una endofítica de nódulos, algunos trabajos han verificado nodulación para estos géneros (Marra et al., 2012; Jaramillo et al., 2013).
La evaluación de las capacidades PGPR en esta investigación, medidas cualitativamente, nos permitió encontrar microorganismos con potencial uso como bioestimuladores, sin embargo, se necesitan más pruebas in vitro y en campo, para determinar realmente su efectividad.
Inoculación en plántulas de tarwi a nivel de invernadero
La selección de los inoculantes se realizó a partir de los 23 aislados descritos en la Tabla 2. La cepa con mayor producción de AIA fue RB14 y la más solubilizadora de fosfato fue RB10, ambos formaron parte de la co-inoculación. El tercer tratamiento fue RB01 por ser la cepa rizosférica con los valores más altos en solubilización de fosfato y producción de AIA, simultáneamente. Por último, el cuarto tratamiento fue TB05, como la cepa endofítica con más alto valor en solubilización de fosfatos y producción de sideróforos. Las variables evaluadas fueron altura de planta (Tabla 4), longitud de inflorescencia (Tabla 5), peso fresco y seco (Tabla 6).
En la evaluación realizada a los 42 días (p = 0.096), aunque no se observaron diferencias significativas en altura de planta, TB05 mostró valores más altos en comparación a los otros tratamientos, siendo un 11% superior al control. El tamaño de la inflorescencia en las plántulas también fue evaluado en dos ocasiones. A los 24 días (p = 0.218) se observó que los tratamientos mostraron inflorescencias de mayor longitud que el control, pero sólo TB05 logró valores superiores con un promedio de 19.1 mm en comparación al testigo quien obtuvo un promedio de 13.7 mm. A los 35 días (p = 0.334), el inóculo mixto (RB10/14) logró el mayor promedio de longitud (61.8 mm), sin embargo, ninguno de los tratamientos mostró significancia estadística respecto al control, cuyo valor promedio de longitud fue 41.7 mm.
Tabla 4 Evaluación de altura (cm) en plántulas de tarwi. Datos por tratamiento de inoculación.
| DDI | 0 | 10 | 17 | 24 | 35 | 42 |
|---|---|---|---|---|---|---|
| Testigo | 16.55 ± 1.96 a | 26.89 ± 2.38 a | 34.89 ± 2.60 d | 50.56 ± 5.44 b | 69.89 ± 6.92 b | 75.44 ± 6.98 b |
| RB10/14 | 16.20 ± 2.04 a | 27.10 ± 2.56 a | 37.22 ± 1.75 c | 52.56 ± 5.48 ab | 78.33 ± 7.97 a | 82.33 ± 8.59 ab |
| RB01 | 17.10 ± 1.52 a | 28.30 ± 2.06 a | 39.40 ± 1.43 b | 54.40 ± 3.63 ab | 74.00 ± 7.01 ab | 77.70 ± 6.89 ab |
| TB05 | 17.45 ± 3.87 a | 28.60 ± 4.11 a | 41.22 ± 1.62 a | 56.22 ± 4.89 a | 77.22 ± 5.47 a | 83.22 ± 8.52 a |
| DDI: Días después de la inoculación. | abcd: Comparación mediante la prueba de Diferencia Mínima Significativa, por columna. |
Tabla 5 Evaluación de inflorescencia (mm) en plántulas de tarwi. Datos por tratamiento de inoculación.
| DDI | 24 | 35 |
|---|---|---|
| Testigo | 13.75 ± 5.74 b | 41.77 ± 22.70 a |
| RB10/14 | 17.26 ± 5.07 ab | 61.68 ± 19.86 a |
| RB01 | 15.46 ± 4.17 ab | 58.11 ± 31.69 a |
| TB05 | 19.15 ± 6.98 a | 53.49 ± 19.68 a |
| DDI: Días después de la inoculación. | abcd: Comparación mediante la prueba de Diferencia Mínima Significativa, por columna. |
Tabla 6 Evaluación de peso fresco y seco (g) en plántulas de tarwi a los 42 DDI. Datos por tratamiento de inoculación.
| Peso fresco (g) | Peso seco (g) | |
|---|---|---|
| Testigo | 40.45 ± 14.09 a | 6.28 ± 2.34 a |
| RB10/14 | 40.25 ± 13.98 a | 6.41 ± 2.41 a |
| RB01 | 39.88 ± 15.85 a | 6.85 ± 2.33 a |
| TB05 | 51.52 ± 8.28 a | 8.15 ± 1.60 a |
| abcd: Comparación mediante la prueba de Diferencia Mínima Significativa, por columna. |
Al finalizar el ensayo en invernadero, se evaluó el peso fresco y seco de las plántulas de tarwi. En ambos casos, el inóculo TB05 mostró los mayores valores, sin embargo, no se encontró significancia entre los promedios con respecto al control (p = 0.204 para peso fresco y p = 0.275 para peso seco). Se puede resaltar, además, que sólo para el parámetro peso seco, los otros tratamientos mostraron también valores superiores al control. El tratamiento TB05 fue el único que formó frutos (dato no evaluado por ausencia de resultados en los otros tratamientos), y fue un aislado endófito proveniente de los tallos de Lupinus mutabilis. Al respecto, Rosenblueth & Martínez (2006) indican que la capacidad de las bacterias de colonizar tejidos internos de las plantas puede conferirles una ventaja ecológica sobre las bacterias rizosféricas, pues pueden protegerse mejor de los factores bióticos y tensiones abióticas que condicionan a todas las poblaciones bacterianas. Dibut et al. (2009) nos mencionan distintos estudios en Cuba con inoculación del endófito Gluconacetobacter diazotrophicus en cultivos de importancia económica; por ejemplo, en Ipomoea batatas (camote) se logró incrementar el largo de las plantas en un 34%, el número de hojas en un 62%, diámetro del tallo en un 40%, diámetro del tubérculo en un 45% en relación con plantas testigo; en Colocasia esculenta (malanga) se incrementó el rendimiento agrícola en un 38%; en Manihot esculenta (yuca) se incrementó la altura en un 36%, entre otros. Los porcentajes de incremento de la cepa TB05 respecto al control son 10% para la altura de planta, 28% para el tamaño de inflorescencia, 27% para peso fresco y 29% para peso seco, que si bien, no tuvieron significancia estadística, sugiere a TB05 como un tratamiento con potencial a ser utilizado como bioinoculante.
Conclusiones
En este estudio se aislaron bacterias y hongos rizosféricos y endofíticos, los cuales mostraron mecanismos de acción para la promoción de crecimiento de la planta. La mayor parte de los aislamientos bacterianos se identificaron pertenecientes a la familia Bacillaceae. Los géneros encontrados fueron Bacillus, Achromobacter, Lysinibacillus, Paenibacillus y Enterococcus, para bacterias y Aspergillus y Penicillium para hongos. La bacteria endofítica TB05 identificada como Lysinibacillus macroides incrementó en un 10% la altura en plántulas de tarwi en invernadero. Los resultados observados nos permiten sugerir el potencial de uso de estos microorganismos como biofertilizantes para este cultivo. Las evaluaciones futuras in vivo deben apuntar a confirmar el potencial uso de estas cepas como inoculantes, biofertilizantes o agentes de control biológico para reemplazar productos químicos e incrementar la sostenibilidad en la agricultura.












 uBio
uBio