Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.14 n.2 Lima jul./dic 1997
ARTÍCULOS CIENTÍFICOS
Diagnóstico temprano en un brote epidémico del virus Dengue en Piura usando RT-PCR y nested-PCR
Nolasco Oscar 1; Carrillo CarloS 2; Gutierrez Victoria 3; Yabar Carlos 1; Douglas Susan 1, García María 3; Montoya Ysabel 1.
1 División de Biología Molecular, Centro Nacional de Laboratorios en Salud Pública, Instituto Nacional de Salud, Lima - Perú
2 Jefatura, Instituto Nacional de Salud, Lima - Perú
3 División de Virología, Centro Nacional de Laboratorios en Salud Pública, Instituto Nacional de Salud, Lima-Perú.
RESUMEN
Un test de diagnóstico temprano (RT-PCR y Nested-PCR) fue evaluado y comparado con métodos convencionales (cultivo in vitro, IFI y MAC-ELISA). Treinta y cuatro sueros de pacientes correspondientes de un brote epidémico de la costa norte peruana (Mancora, Piura) en mayo de 1997 fueron incluidos en este estudio. Todos los sueros fueron obtenidos de pacientes que presentaron en los primeros cinco días manifestaciones clínicas siendo diagnosticados luego como dengue serotipo 1. Asimismo, RT-PCR permitió diagnosticar 82% de los sueros (28/34), sin embargo Mac-ELISA y cultivo in vitro reconocieron unicamente 41% de los sueros (14/34) y 38% de los sueros (13/34) respectivamente. Por lo tanto, el uso de esta herramienta molecular (RT-PCR y Nested-PCR) permitiró dar un diagnóstico temprano a estos pacientes y actuar inmediatamente ante la presencia de un brote epidémico.
Palabras claves: Dengue, RT-PCR, Nested-PCR, Perú.
ABSTRACT
An early diagnostic test (RT-PCR and Nested-PCR) was assessed and compared with conventional methods (in vitro culture, IFI and MAC-ELISA). Thirty four sera of patients corresponding to a dengue virus outbreak from the Peruvian North Coast (Mancora, Piura) in May 1997 were included in this study. All sera were obtained from patients suffering the first five days of clinical manifestation and were diagnosed as dengue. Likewise, RT-PCR was able to diagnose 82% sera (28/34) however MAC-ELISA and in vitro culture recognised only 41% sera (14/34) and 38% sera (13/34) respectively. Therefore, the use of this molecular tool (RT-PCR and Nested-PCR) will allow to give an early diagnosis to the patients and to act immediately in the presence of an epidemic outbreak.
Key words: Dengue, RT-PCR, Nested-PCR, Peru.
INTRODUCCIÓN
La infección en humanos por el virus dengue está considerada como una enfermedad reemergente en América Central y del Sur 1 por lo que su diagnóstico temprano es importante en salud pública. En el Perú, se han reportado varios brotes epidémicos en ciudades de la Selva amazónica norte y central y en la Costa norte producidos por el virus dengue serotipo 1 desde 19902 . Debido a la aparición de infecciones ocasionados por el virus dengue serotipo 2 en 1995, el Perú se ubicó como un país en riesgo de presentar dengue hemorrágico3. Sin embargo, es de resaltar que hasta la actualidad sólo se han reportado casos de dengue clásico en el Perú.
Generalmente, el diagnóstico serológico de la enfermedad usando MAC-ELISA permite detectar anticuerpos virales a partir del quinto día de las manifestaciones clínicas del paciente 4. Asimismo, la confirmación del agente etiológico se realiza por cultivo celular mediante la visualización del efecto citopático y por evaluacion del IFI. Sin embargo, ambos métodos requieren de tiempos prolongados a fin de obtener un diagnóstico satisfactorio.
Este trabajo de investigación reporta la optimización y validación de las técnicas Transcripción Reversa-Reacción en Cadena de la Polimerasa (RT-PCR) y Nested-Reacción en Cadena de la Polimerasa (Nested-PCR) para el diagnóstico del dengue circulante en nuestro país. De esta manera, el Instituto Nacional de Salud dispone de herramientas moleculares a ser usadas en una intervención temprana ante un brote epidémico del virus dengue.
MATERIALES Y MÉTODOS
SUEROS
Durante el brote de dengue ocurrido en el mes de mayo 1997 en la Costa norte peruana (Mancora, Piura), treinta y cuatro sueros fueron obtenidos provenientes de pacientes con síntomas clínicos compatibles a esta enfermedad. Estas muestras serológicas fueron evaluadas por la técnica de MAC ELISA, IFI y RT-PCR. Asimismo, diez sueros fueron obtenidos de donantes sanos y usados como controles negativos para evaluar las técnicas.
CULTIVOS VIRALES IN VITRO
En el Laboratorio de Arbovirus del Instituto Nacional de Salud los sueros provenientes de casos clínicos sospechosos con infecciones virales de dengue fueron aislados in vitro y tipificados por inmunofluorescencia indirecta (IFI) usando anticuerpos monoclonales antidengue. Asimismo, fueron cultivados in vitro las cepas referenciales del virus dengue-1 (Hawai), dengue-2 (New- Guinea), dengue-3 (H87) y dengue-4 (H 241) proporcionados por el CDC de Atlanta, Georgia, EE.UU. y el virus de fiebre amarilla (F8). El aislamiento del virus dengue se realizó en cultivo celular de mosquito Aedes albopictus clon C6/36 en medio de cultivo MEM suplementado con 5% de suero fetal bovino. Los cultivos se desarrollaron hasta un 75% a 100% de visualización del efecto citopático.
EXTRACCION DE ARN
Para la extracción de ARN del virus dengue a partir de cultivos y sueros fueron empleados dos métodos. El primero se realizó empleando el reactivo TRIZOL (Gibco, BRL). Este método se basa en separar las moléculas de ARN en un sistema de fenol y precipitación con alcohol5.
El segundo método fue realizado usando un kit comercial (QIAamp Qiagen). Para tal fin, la muestra biológica es lisada bajo condiciones desnaturalizantes. Posteriormente, la muestra es colocada en una columna de afinidad a la cual se unirá el ARN mientras que los contaminantes son eliminados mediante lavados. El ARN puro es liberado de la columna con la adición de agua.
RT-PCR
La transcripción reversa (RT) se realizó usando el oligonucleótido D2 específico correspondiente a la región específica que codifica a la proteína preM del virus dengue6. Asimismo, en el sistema de reacción se incluyó a la enzima transcriptasa reversa AMV (Avian Myeloblastosis virus), por un período de 90 minutos a 42ºC.
La Reacción en Cadena de la Polimerasa (PCR) fue realizada usando los oligonucleótidos D2, D1 y la enzima. ADN Polimerasa Taq Gold (Perkin Elmer). La etapa de desnaturalización se realizó a 94ºC durante 50 segundos, la hibridación a 57ºC durante 90 segundos y la extensión durante 72ºC por 120 segundos. PCR se llevó a cabo en 35 ciclos seguidos de una extensión final a 72ºC durante 10 minutos.
NESTED-PCR
El fragmento de ADNc amplificado en RT-PCR diluído 1 /100 fue sometido a una segunda amplificación (Nested-PCR), usándose el oligonucleótido iniciador D1 con oligonucleotidos específicos para cada Serotipo6, Para amplificar el fragmento específico para virus dengue 1 se usaron los oligonucleotidos TSI y D1, para el serotipo dengue 2 se usaron los oligonucleotidos iniciadores TS2 y D1. Asimismo, para dengue 3 se uso TS3 con Dl y para dengue 4 se uso TS4 con D1. Las condiciones de amplificación incluyeron 30 ciclos de desnaturalización 94ºC por 50 segundos, hibridación a 57ºC por 90 segundos y de extensión a 72ºC por 120 segundos seguida de una extensión final de 72ºC por 10 minutos con la enzima ADN Polimerasa Taq Gold. Los productos de amplificación fueron separados por electroforesis en un gel de agarosa al 4% y visualizados con luz ultravioleta previa tinción con bromuro de etidio.
RESULTADOS
EXTRACCIÓN DE ARN VIRAL
Ambos métodos fueron comparados y se determinó que ambos pueden ser usados indistintamente puesto que la pureza y calidad del material biológico es satisfactorio. Sin embargo, se sugiere el uso del kit de Qiagen para la extracción de ARN a partir de sueros por su rapidez, simplicidad y evitar el riesgo que residuos de etanol alteren la actividad de la enzima durante la PCR. Asimismo, para la extracción de ARN a partir de los cultivos virales se sugiere el uso del reactivo Trizol.
DETECC1ÓN Y TIPIFICACIÓN DEL VIRUS DENGUE A PARTIR DE SUEROS Y CULTIVOS IN VITRO
Al usar los oligonucleotidos iniciadores correspondientes a la región Pre-M del virus (D1 y D2) y realizar el RT-PCR se obtuvo exitosamente el producto de amplificación correspondiente a 511 pb. Este producto al ser sometido al Nested PCR para la identificación de los serotipos dengue 1, dengue 2, dengue 3 y dengue 4 se obtuvieron, como se esperaba, fragmentos de 482 pb, 119 pb, 290 y 392 respectivamente (Figura 1). Además, fueron evaluados los ARN de los cultivos virales de Fiebre Amarilla y ARN de cultivos de células de mosquito C6/36 sin infectar, no observándose algún producto de amplificación (Foto 1).
Foto 1: Análisis de los productos de Nestel PCR en gel de agarosa al 4%. Carril 1 RT-PCR de ARN total de cultivo de células de mosquito sin infectar;Carril 2 RT-PCR de ARN total de cultivo viral de fiebre amarilla; Carril 3 RT-PCR de ARN total de cultivo viral de dengue 1 (para todos los serotipos se obtuvo el producto del mismo tamaño); Carril 4 RT-PCR Marcador Ladder 100 pb; Carril 5 Nested PCR para serotipo de dengue 1; Carril 6 Nested PCR para serotipo de dengue 2; Carril 7 Nested PCR para el serotipo de dengue 3; Carril 8 Nested PCR para serotipo de dengue 4.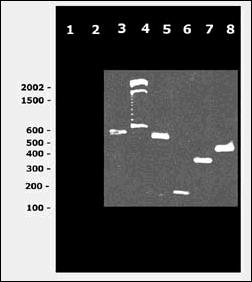
Y CULTIVO IN VITRO
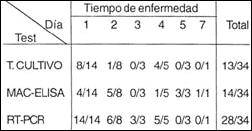
Las muestras de sueros fueron evaluadas por RT-PCR & Nested PCR, así como por MAC ELISA y cultivos virales. La Tabla 1 muestra que los sueros de Piura tipificados por IFI y RT-PCR & Nested-PCR corresponden a dengue serotipo 1.
Asimismo, el Nested-PCR reconoció al 82% de sueros (28/34) mientras que la técnica de MAC ELISA reconoció 41 % (14/34) de los sueros evaluados durante la primera semana de las manifestaciones clínicas. Una similar sensibilidad diagnóstica fue observada a partir de los cultivos virales evaluados (38% = 13/34).
Sin embargo, los sueros que presentan mayor tiempo de enfermedad (>4 días) no pudieron ser diagnosticados por RT-PCR ni ser aislados por cultivos in vitro. Por lo tanto, para evaluar sueros de pacientes con síntomas clínicos mayor a cuatro días se les debe realizar el MAC ELISA (Tabla 1). El serotipo (Dengue 1) fue tipificado en todas las muestras positivas por IFA así como también por Nested -PCR.
Tabla N° 1
Detección del Virus Dengue durante la primera semana de infección
en muestras serológicas provenientes de Máncora (Piura).
DISCUSIÓN
El virus dengue afecta a más de 100 países a nivel mundial y su prevalencia se debe a múltiples factores entre los que se debe considerar las condiciones de pobreza y sanidad principalmente de las ciudades tropicales y subtropicales de América. Por otro lado, la proliferación del vector Aedes albopictus y su rápida dispersión contribuyen a la reemergencia de epidemias que se pensaban estaban bajo control 7,8.
El desarrollo de recientes tecnologías tal como la Reacción en Cadena de la Polimerasa (PCR) permiten disponer de un test de diagnóstico temprano a partir de muestras serológicas desde el primer día de las manifestaciones clínicas del virus dengue en humanos.
Existen múitiples reportes en la literatura sobre secuencias oligonucleotídicas descritos para ser aplicados en el diagnóstico de esta enfermedad sin embargo estas secuencias nucleotídicas inicialmente deben ser validados en el país donde serán usados en el diagnóstico para su correcta aplicación epidemiológica. Nuestros resultados sugieren que los sets de oligonucleótidos reportados por Lanciotti, 1992 pueden ser aplicados satisfactoriamente en Piura. Sin embargo, necesitan ser evaluadas otras áreas epidémicas del virus dengue para poder usar estas secuencias nucleotídicas a nivel nacional.
RT-PCR y Nested-PCR son técnicas comparativamente mas sensibles que el MAC-ELISA usadas en la detección y tipificación del virus dengue. Asimismo, estas herramientas moleculares brindan un diagnóstico temprano de la enfermedad puesto que la muestra serológica debe ser tomada durante los primeros cinco días de manifestaciones clínicas. Es así que podemos disponer de un ensayo de diagnóstico rápido y conocer rápidamente la coexis tencia de los serotipos de virus dengue circulando en territorio peruano para tomar las medidas apropiadas frente al riesgo de presentarse casos de dengue hemorrágico.
Actualmente, los productos de amplificación obtenidos en este trabajo de investigación han sido clonados y su ADN esta siendo secuenciado a fin de determinar las variaciones en la secuencia del virus dengue serotipo 1 circulante en el Perú. Asimismo, la disponibilidad de la secuencia de este serotipo circulante per mitirá desarrollar nuevos oligonucleótidos iniciadores que permitan identificar nuevos serotipos y/o mejorar su identificación en el diagnóstico.
BIBLIOGRAFÍA
1. Gubler,D.J. & Trent D.W. 1994. Emergence of epidemic dengue/dengue hemorragic fever as a public health problem in the Americas. Infectious Agents Diseases. 2:83 -393. [ Links ]
2. García, M., Cabezas, C., Callahan, J.,Yana, B.,Gutierrez, V.,Ortiz, A. & Anaya, E. 1997. Determinacion de IgG y Anticuerpos totales contra el virus dengue, en muestras obtenidas en papel filtro. Revista de Medicina Experimental 14(1) : 45-49. [ Links ]
3. Boletín informativo del Instituto Nacional de Salud.(1996) Año II. Nº 2. Ministerio de Salud. [ Links ]
4. Nogueira, R.M.R., Miagostovich,. M.P., Cavalcunti, S.M.B., Marzochi, K.B.F. and Schatzmays, H.G. 1992. Levels of IgM Antibodies against dengue virus in Río de Janeiro, Brazil. Institut Paster Elsevier. París. [ Links ]
5. Chomczynski,P. & Sacchi N. 1987. Single step method of RNA isolation by acid guanidinium thiocyanate - phenol - chloroform extraction. Anal. Biochem. 162: 156-159. [ Links ]
6. Lanciotti, R., Calisher, Ch., Gubler, D., Ghang, G. and Vorndam, V. 1992. Rapid Detection and Typing of dengue viruses from Clinical samoles by Using Reverse Transcriptase-Polymerase Chain Reaction. Journal of Clinical Microbiology. 30(3): 545-551. [ Links ]
7. World Health Organization (Genova). The World Health Report 1996. Fighting disease fortering development [ Links ]
8. Infecfious Disease - A global Health Threat. Report of the National Science and Technology Council. Committee on International Science, Engineering and Technolog. Working Group on Emerging and Reemerging Infectious Diseases. September 1995. [ Links ]
AGRADECIMIENTOS
Los autores agradecen al CDC de Atlanta, Georgia EEUU por proporcionarnos las cepas referenciales del virus dengue-1 (Hawai), dengue-2 (New Guinea), dengue-3 (H87) y dengue-4 (H 241). Además se agradece a la Dra. Laura Chandler de la Universidad de Texas, Galvestone, por proporcionarnos los sets de oligonucleótido usados en esta investigación.














