Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.15 n.1-2 Lima ene./dic. 1998
ARTÍCULOS CIENTÍFICOS
Identificación de una nueva proteína en Leishmania (Viannia) peruviana
De los Santos, Maxy 1 y Montoya, Ysabel 1
1 División de Biología Molecular; Centro Nacional de Laboratorios de Salud Pública, Instituto Nacional de Salud.
RESUMEN
El análisis de la secuencia nucleotídica y aminoacídica de un clon de la biblioteca de expresión en fago λgt11 de Leishmania (Viannia) peruviana, estableció identidad parcial con los genes de las proteínas acídicas ribosomales P2 de Leishmania (Leishmania) infantum. Este hallazgo unido a ciertos dominios geonómicos conservados, sugeridos de la comparación de 14 secuencias de otras proteínas P1 eucarióticas, confirman que la secuencia del inserto de clon codifica la proteína acídica ribosomal P1 de L. (V.) peruviana denominada LpP1. Este es el primer reporte sobre este tipo de proteína en el género Leishmania.
Palabras claves: Leishmania, leishmaniasis, proteína acídica ribosomal, proteínas-P.
ABSTRACT
Nucleotidic and aminoacidic sequence analysis from a clone of a phage λgt11 obtained from a expression vector library of Leishmania (Viannia) peruviana, established partial identity with the genes of the acidic ribosomal proteins P2 of Leishmania (Leishmania) infantum. These molecular findings along with certain conserved genomic domains, suggested by comparison of 14 sequences of eukaryotic P1 proteins, confirmed that the insert of the clone codes for acidic ribosomal protein P1 of L (V) peruviana refered as LpP1. This is the first report about this type of protein in Leishmania genus.
Key word: Leishmania, leishmaniasis, acidic ribosomal proteins, P-proteins.
INTRODUCCIÓN
Las leishmaniasis son un grupo de infecciones de la piel. membranas mucosas y vísceras que afectan entre 12 a 14 millones de personas en el mundo con una incidencia de dos millones de nuevos casos anuales y 368 millones de personas en riesgo de infección en el mundo 1.
En el Perú la enfermedad presenta dos formas clínicas asociadas a parásitos del complejo braziliensis: la leishmaniasis cutánea andina (LCA) conocida como "uta" causada por L. (V.) peruviana y la leishmaniasis selvática (LS), conocida en su forma mucocutánea corno "espundia" la cual es causada por L. (V.) braziliensis. La "uta" causa lesiones cutáneas benignas, responde al tratamiento clínico o autocura y se presenta en los valles interandinos, mientras que la "espundia" causa lesiones mucocutáneas metastásicas desfigurantes y se distribuye en la selva 2.
El interés y factibilidad de conocer nuevos genes relacionados con la respuesta humoral llevó a la construcción de una biblioteca de expresión en fago 1gt11, a partir de promastigotes en fase logarítmica temprana de L. (V.) peruviana cepa MHOM/PE/84/LC26 lo que permitió clonar, aislar y caracterizar algunos genes aún no identificados en el parásito 3.
Uno de los clones de la biblioteca de cDNA denominado T26-U3, parcialmente secuenciado a nivel de nucleótidos, presentó homología parcial con las secuencias que codifican las proteínas acídicas ribosomales de tipo P2 de L. (L.) infantum y con las de tipo P1 de Mus musculus y Homo sapiens. Nuestro propósito fue realizar el análisis de la secuencia nucleotídica completa y deducir la secuencia aminoacídica para poder establecer el tipo de proteína P codificada.
Las proteínas acídicas ribosomales o proteínas P han sido muy estudiadas genética y serológicamente, son importantes antígenos que generan respuesta humoral en la enfermedad de Chagas 4 la leishmaniasis visceral canina, la leishmaniasis selvática 5, la brucelosis 6, la babesiosis 7 y el lupus eritematoso sistémico (LES) 8.
MATERIAL Y MÉTODOS
Material biológico
Se utilizó el clon T26-U3 que porta el inserto de ADN de Leishmania el cual ha sido clonado en el fago λgt11. La propagación de los fagos fue realizada usando la bacteria E. coli Y1090 mientras que para el mantenimiento y crecimiento de los plásmidos, se utilizó la cepa JM 109.
Se usaron los plásmidos pUC18EcoRI/BAP (Pharmacia) y pCR-Script Amp SK(+) (Stratagene) para el subclonamiento.
Optimización del PCR para el clon T26-U3
El ADNc del clon T26-U3 fue amplificado por PCR utilizando los primers específicos para 1gt11 forward, 5' GGTGGCGACGACTCCTGGA GCCCG 3' y reverse, 5' TTGACACCAGAC CAACTGGTAATG 3' a partir de una placa de lisis del fago crecido en E coli Y1090. El buffer de reacción contuvo 1 U de enzima AmpliTaq DNA Polimerasa y 2 mM de MgCl2. Las condiciones usadas durante la amplificación in vitro fueron 95 ºC por 30 s, 40 ºC por 1,5 min. y 72 ºC por 2 min. + 2s/ciclo durante 30 ciclos. Al producto amplificado se le denominó PCR-U3.
Subclonamiento en pUC18
El producto PCR-U3 fue cortado con 25 U de la endonucleasa EcoRl Para permitir su subclonamiento mediante ligación, en el vector pUC 18 EcoRI/BAP (Pharmacia Biotech), utilizando 2U de T4 DNA ligasa. Las mezclas de ligación fueron precipitadas usando etanol absoluto. El ADN purificado fue usado para transformar E. coli JM 109 por electroporación con un pulso de 1,8 kV.
Las colonias recombinantes fueron seleccionadas para una maxipreparación de ADN plasmídico utilizado en el secuenciamiento temático basado en una modificación del método de terminación de cadena mediado dideoxinucleósidos trifosfatos (ddNTPs)9. Los plásmidos recombinantes fueron denominados pUC18U3.
Secuenciamiento automático de ADN
El ADN fue secuenciado usando el colorante fluorescente cianina (Cy5) (kit Auto Read Pharmacia): Universal 5'-Cy5-d (CGACGTTGTAAAACGACGGCCAGT)-3'y reverso5'- C y 5-d (CAGGAAACAGCTATGAC) -3'. Inicialmente, cinco µg de ADN de pUC18-U3 fueron denaturados con NaOH 0,4 N. Después, el ADN de simple hebra fue precipitado con acetato de Na 0,12M pH 4,8, agua MilliQ y etanol absoluto a -80 ºC. Posteriormente, se centrifugó y el ADN fue centrifugado y lavado con etanol al 70% y resuspendido en agua MilliQ, para agregarle los primers e incubarlo a 65 ºC, 37 ºC y a temperatura ambiente. Finalmente se adicionó DMSO, la enzima T7 DNA Polimerasa, y las mezclas; se dividieron para añadirles los ddNTPs. Antes de colocar las muestras en el gel de secuenciamiento (ReadyMix Gel-Pharmacia), fueron calentadas a 90 ºC. La electroforesis se realizó a 57 ºC por 600 min. Un láser clase 1 de He-Ne excitó el Cy5 a medida que las muestras migraban, la luz emitida fue fotodetectada y digitalizada para su procesamiento por computadora.
Programas de ADN
Las secuencias nucleotídicas obtenidas fueron analizadas utilizando la interfase BLAST. También se usaron los programas DNA Strider 1,0 para hacer mapas de restricción, hallar marcos de lectura e hidrofobicidad y SeqEd 1.0.3 para el alineamiento múltiple de secuencias aminoacídicas.
RESULTADOS
Secuencia parcial de ADN y análisis de restricción del clon T26-U3
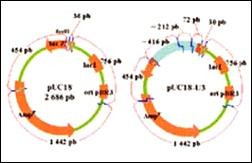
El producto de amplificación de aproximadamente 750 pb referido como PCR-U3 fue cortado con la enzima Eco RI obteniéndose un fragmento de ~700 pb el cual fue subclonado en PUC18 (Fig. 1). La secuencia parcial del recombinante pUC18-U3, asociado al alto contenido de GC, sugirió que contiene 39 nucleótidos que codifican los últimos 13 residuos aminoacídicos del extremo C-terminal de una proteína homóloga a las acídicas ribosomales de tipo P2 de L. (L.) infantum (LiP y LIP')10 y a las acídicas ribosomales de tipo P1 de Cladosporium herbarum, Alternaria alternata, Caenorhabditis elegans, Mus musculus y Homo sapiens. Por lo tanto para completar la secuencia se realizó el mapa de restricción del vector pUC18, del recombinante pUC18-U3, de LiP, de LiP' y de las proteínas P2 de L. (V.) braziliensis y L. (V.) peruviana 11. La enzima de restricción TaqI fue seleccionada por generar el menor número de fragmentos; (Fig. 2), siendo el de ~416 pb subclonado en pCRScript Amp SK (+) a fin de completar la secuencia nucleotídica (Fig. 3).
Fig. 1 El producto de amplificación PCR-U3 (carril 2, ~750 pb, 50 ng)
fue digerido con la endonucleasa EcoRI, para liberar el fragmento
PCR-U3/EcoRI (carril 3, ~700 pb, 80 ng) el cual fue subclonado
en el vector pUC18 (carril 4, 2686 pb, 150 ng) cortado
y desfosforilado (carril 5, pUC18 EcoRI/BAP, 75 ng).
Carril 1: marcador de 100 pb (300 ng);
carril 6: marcador λ/Hind III (250 ng).
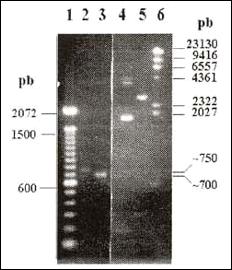
Fig. 2 Cortes y fragmentos obtenidos con el uso de la enzima Taq I.
Los cortes están indicados en azul o celeste, excepto para EcoRI.
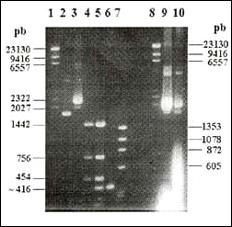
Fig. 3 Fragmentos de pUC18-U3 generados por Taq I (carril 5).
El fragmento de ~416 pb (carril 6), fue subclonado en
pCR-Script Amp SK(+) (carril 9). El nuevo plásmido
pCRS-U3 (carril 10) permitió completar la secuencia
de T26-U3. Carriles 1 y 8, λ/Hind III, carril 2,
pUC18, carril 3, pUC18-U3, carril 4, pUC18/TaqI;
carril 7, ΦX174/ Hae III Gel de agarosa al 1.5%.
La secuencia del clon T26-U3 codifica la proteína acídica ribosomal de tipo P1 de L. (V.) peruviana
La secuencia nucleotídica completa del clon T26-U3 abarca 676 pb, conteniendo un marco de lectura que codifica una proteína de 107 aminoácidos. La región 5' no codificante del clon no posee el miniexón y la región 3' no codificante presenta una cola de poliA.
La secuencia del T26-U3 presenta identidad parcial con los genes de las proteínas LiP' y LiP de L. (L.) infantum y el tramo G-GTG-(GT)11, -A-GG-TG-G-G de T26-U3 tiene identidad parcial con la región 3' no codificante de LiP'.
Cuando se comparó la secuencia aminoacídica del clon T26-U3 mediante BLAST, se confirmó que codifica una proteína homóloga a las proteínas P1 de diversos eucariotes 12. Así se encontró 71,43% de homología total (51,02% identidad, 20,41% cambios conservativos) con la proteína P1 de T. cruzi (TcP1). Por lo tanto T26-U3 contiene la proteína acídica ribosomal de tipo P1 de L. (V.) peruviana a la que se denomino LpP1. La secuencia de LpP1 está disponible en la base de datos GenBank/EMBL/DDBJ con código AF045249 desde febrero de 1998.
DISCUSIÓN
La secuencia nucleotídica descrita, codifica la proteína acídica ribosomal de tipo P1 de L. (V.) peruviana (LpP1), lo cual constituye la primera información sobre la proteína P1 en el género Leishmania.
Hasta el momento los pocos estudios sobre dominios conservados en las proteínas-P de tripanosomátidos, han sido incompletos y contradictorios 4,13 Por ello el análisis comparativo de este gen con otras 14 secuencias aminoacídicas de las proteínas P1 de distintos eucariotes, proporcionó datos confiables de las regiones de LpP1. Las regiones conservadas en LpP1 pueden sostener el hecho que la proteína conservaría su función estructural como parte del pedúnculo ribosomal de L. (V.) peruviana, al lado de otras proteínas P14,15, de las cuales solo se han descrito las de tipo P2 en L. (L.) infantum 10.
Una característica de importancia inmunológica es la región hidrofílica adyacente al extremo C-terminal de la LpP1, que al estar expuesta al reconocimiento de los anticuerpos, podría contener el principal epítope antigénico lineal de LpP1, de modo similar a la proteína P2 de Artemia salina, donde el epítope lineal en la zona hidrofílica es reconocido por los autoanticuerpos del lupus eritematoso sistémico (LES)8. Además la conservación del dominio de máximo valor de hidrofilicidad, podría representar parte del principal epítope lineal antigénico de LpP1. de forma similar a los epítopes de la proteínas P2 de L (L.) infantum que no se ubican en la secuencia consenso de la región C-terminal 16.
En conclusión podemos señalar que la secuencia nucleotídica completa del clon T26-U3 contiene codificada la proteína acídica ribosomal de tipo P1 de L. (V) peruviana (LpP1). Dicha secuencia presenta identidad parcial con las secuencias de los genes de las proteínas LiP' y LiP de L. (L.) infantum. Además el tramo G-GTG-(GT)11-A-GG-TG-G-G de LpP1 tiene identidad parcial con la región 3' no codificante de LiP' de L.(L.) infantum, mientras la secuencia aminoacídica de LpP1 tiene 71.43% de homología con la proteína TcP1 de T. cruzi.
REFERENCIAS BIBLIOGRÁFICAS
1. WHO (1996) The World Health Report. Fighting disease, fostering development. Report of the Director General. WHO Library Cataloguing in Publication Data. [ Links ]
2. Lumbreras H. y Guerra H. (1985) Leishmaniasis in Peru. In Human Parasitic Diseases, Vol 1: Leishmaniasis (Ruitenberg E.J. and Mc Innis A.J. eds.) Elsevier Science Publishers. [ Links ]
3. Montoya Y.C. (1993). Molecular análisis of antigen genes in Peruvian Leishmaniasis. Doctoral Thesis. University of Cambridge, 183pp. [ Links ]
4. Skeiky Y.A., Benson D.R., Guderian J.A., Sleath P.R., Parsons M. and Reed S.G. (1993) Tripanosoma cruzi acidic ribosomal P protein gene family. Novel P proteins encoding unusual cross-reactive epitopes. J. Immunol. 151(10): 5504-15. [ Links ]
5. Soto M., Requena J. M., Quijada L., and Alonso C. (1996) Specific serodiagnosis of human leishmaniasis with recombinant Leishmania P2 acidic ribosomal proteins. Clin. Diagn. Lab. Immunol. 3(4): 387-391. [ Links ]
6. Bachrach G., Banai M., Fishman Y. and Bercovier H. (1997) Delayed type hypersensivity activity of the Brucella L7/L12 rihosomal protein depends oil posttranslational modification. Infect. Immun. 65(1): 267-71. [ Links ]
7. Dalrymple B.P. and Peters J.M. (1992) Identification of L10e/L12e ribosomal protein genes in Babesia bovis. Nucleic Acid Research. 20: 2376. [ Links ]
8. Elkon K., Skelly S.. Parnassa A., Moller W., Danho W., Weissbach H and Brot N. (1986) Identification and chemical synthesis of a ribosomal protein antigenic determinant in systemic lupus erythematosus. Proc. Nat. Acad. Sci. USA 83: 7419-7423. [ Links ]
9. Sanger F. Nicklen S. and Coulsen A.R. (1977) DNA sequencing with Chain terminating inhibitor. Proc. Natl. Acad. Sci. USA 74:5463-7. [ Links ]
10. Soto M., Requena J. M., García M., Gómez L., Navarrete I. and Alonso C. (1993) Genomic organization and expression of two independent gene arrays coding for two antigenic acidic ribosomal proteins of Leishmania. J. Biol. Chem. 268 (29): 21835-43. [ Links ]
11. Panebra A. (1996). Respuesta humoral en leishmaniasis del Perú: Proteínas HSP-70 y Ribosomales P. Tesis Doctoral. Universidad de Buenos Aires, 207pp. [ Links ]
12. Shimmin L. C., Ramirez C., Matheson A. T and Dennis P. (1989) Sequence alignment and evolutionary comparison of the L10 equivalent and L12 equivalent ribosomal proteins from archaebacteria, eubacteria, and eucaryotes. J. Mol. Evol. 29:48-62. [ Links ]
13. Vázquez M. P., Schijman A. G. and Levin M. J. (1992) Nucleotide sequence of a cDNA encoding a Tripanosoma cruzi acidic ribosomal P1 type protein. Nucleic Acid Research. 20(10): 2599. [ Links ]
14. Lin A., Wittman-Liebold B., Mc Nally J. and Wool J.G. (1982) The primary structure of the acidic phosphoprotein P2 from rat liver 60 S ribosomal subunits. J. Biol. Chem. 257 (15): 9189-97. [ Links ]
15. Leijonmarck M., Eriksson S. and Liljas A. (1980) Crystal structure of a ribosomal component at 2.6 A resolution. Nature. 286 (5775): 824-26. [ Links ]
16. Soto M., Requena J. M., Quijada L., Ángel S.O., Gómez L.C., Guzmán F, Patarroyo M.E. and Alonso C. (1995a) During active viscerocutaneous leishmaniasis the anti-P2 humoral response is specifically triggered by the parasite P proteins. Clin. Exp. Immunol. 100: 246-52. [ Links ]














