Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.15 no.1-2 Lima Jan./dic. 1998
ARTÍCULOS CIENTÍFICOS
Estandarización de la PCR para el diagnóstico del virus de la Hepatitis B en el Perú.
Hijar G.1; Carrillo C.3; Padilla C.1; Cabezas C.2; Suárez M.2; Romero G.2 y Montoya Y.1
1 División de Biología Molecular Nacional de Salud, Lima-Perú.
2 División de Virología del Instituto Nacional de Salud, Lima-Perú.
3 Jefe del Instituto Nacional de Salud, Lima-Perú.
RESUMEN
La detección del ADN del virus de la Hepatitis B, (VHB) es el marcador más sensible para determinar la replicación activa e infectividad del virus circulante. Por esta razón la Reacción en Cadena de la Polimerasa (PCR) fue aplicada satisfactoriamente usando primers específicos para gen del antígeno de superficie (HBsAg). Un fragmento de 260 bp fue amplificado in vitro a partir de 200 µl. de sueros de pacientes aplicando PCR.
La tecnología de PCR está siendo aplicada en el diagnóstico de pacientes infectados con el virus de la Hepatitis B pertenecientes a diferentes zonas geográficas.
ABSTRACT
Detection of Hepatitis Viral DNA (HVB) is the most sensitive marker to determinate the active replication and infectivity of the circulating virus. for this reason, the Polymerase Chain Reaction was applied satisfactoriously by using specific primers corresponding to the Hepatitis B surface antigen (HBsAg). A fragment of the 260 bp was in vitro successfully amplified by using 200 µl of sera of patients infected with HVB applying PCR.
The PCR technology is being used in the diagnosis of patients infected with HVB belong to differents geographical areas from Peru.
INTRODUCCIÓN
El Perú un país con más de veintitrés millones de habitantes tiene un promedio de prevalencia entre 1 a 2% para antígeno de superficie (HBsAg) y de 20-30% para anticuerpos contra HBsAg3 del virus de la Hepatitis B (VHB). La prevalencia de la hepatitis B varía de acuerdo a las diferentes regiones geográficas1, localidades, hábitat y los grupos de población distribuidas en las diversas áreas geográficas, presenta zonas hiperendémicas en la región de la selva alta que incluye a 10 departamentos que cuentan con selva alta y áreas rurales de la selva baja6. Además en algunos valles de la vertiente oriental de la cordillera de los Andes como Abancay y Huanta2,7.
El HBsAg es el primer antígeno que aparece en el suero, debido a que ésta proteína es la más inmunogénica y se considera como un marcador temprano de infección por VHB. Cinco a diez por ciento de adultos con infección por VHB, normalmente no se recuperan de la infección y permanecen con seropositividad al HBsAg por largos periodos de tiempo. Estos pacientes se convierten en portadores sanos y/o desarrollan una hepatitis crónica. Se considera que más de dos mil millones de habitantes en el mundo han sido infectados por el virus de la Hepatitis B, de los cuales 280 millones serán portadores crónicos positivos HBsAg.
Se conoce fue el virus se replica activamente en el hígado de los pacientes infectados aumentando la presencia del HBsAg y antígeno de envoltura (HBeAg) en suero, de allí la importancia de poder detectar por la técnica de ELISA a estos dos antígenos. Sin embargo estos antígenos pueden quedar circulantes indefinidamente teniendo una relación directa de la infección y seroconversión del paciente. Por lo que las técnicas moleculares resultan muy útiles para determinar la presencia del ADN del virus tanto en pacientes agudos, crónicos y los que se encuentren en periodo de ventana de la infección.
Hasta la actualidad se cuentas con pruebas inmunológicas que son utilizados para el diagnóstico clínico pero la PCR ayudaría a mejorar el diagnóstico de la enfermedad identificando directamente al virus, lo cual es importante para poder determinar la fase de la enfermedad, pronóstico y tratamiento del paciente.
MATERIALES Y MÉTODOS
Material Biológico
Los seis sueros empleados para este estudio tuvieron como carácter de inclusión la reactividad de al HBsAg y HBeAg del HVB empleando la técnica de ELISA. La confirmación serológica de estos pacientes fue realizada por la División de Virología del INS. Para este sistema es necesario contar con controles positivos y como controles negativos. El control positivo es un suero de paciente positivo a HBsAg y HBeAg, y el control negativo es un suero de paciente que no ha tenido antes la enfermedad o que no se encuentre en áreas endémicas.
Extracción de ADN Viral
El método de extracción de ADN a partir de 200 microlitros de suero, el cual está basado en la lisis de la membrana del virus bajo ciertas condiciones químicas como detergentes, enzimas que permitan lisar la membrana del virus dejando libre el material genético y luego es tratado por una resina de carga catiónica y eluida con agua pura libre de DNAsas y RNAsas.
Luego el ADN es cuantificado por espectrofotometría para conocer la cantidad a utilizar en PCR y poder determinar la sensibilidad.
Reacción en cadena de la Polimerasa (PCR)
La Reacción en Cadena de la Polimerasa (PCR) fue realizada usado oligonucleótidos iniciadores (primers) ya reportados que son específicos para la región del gen del antígeno de superficie (HBsAg) y para todos los subtipos de Hepatitis B11.
Para amplificación se utilizó una enzima ADN Polimerasa, los dNTPs, agua para PCR libre ADNsa y RNAsa, Cloruro de Magnesio, (MgCl2). Los tubos de reacción fueron sometidos a diversas temperaturas: Denaturación del ADN a 95°C por 30 segundos, hibridación de los primers a 55°C por 30 segundos y una extensión a 72°C por 30 segundos. Se repitieron estas 3 temperatura 35 veces y la extensión final a 72°C por 5 minutos.
Se realizó la estandarización de la temperatura de hibridación de los primers, curvas de concentraciones de MgCl2, y curvas de ADN para demostrar la sensibilidad de la técnica. Los productos de amplificación fueron visualizados en geles de poliacrilamida al 12%, coloreados con bromuro de etidio y fotografiadas.
RESULTADOS
El método de extracción fue realizado satisfactoriamente obteniéndose una cantidad de 250 ng. aproximadamente de ADN para cada muestra, así como también la calidad de ADN fue de alta pureza la cual es óptima para ser aplicado a técnicas moleculares como PCR.
En la técnica de PCR se probaron diferentes temperaturas de anillamiento de primers desde 50 a 56°C, siendo la más óptima de 55°C (figura 1).
Teniendo esta temperatura se probaron diferentes concentraciones de MgCl2 desde 1 a 4 mM, siendo la más óptima de 2mM (figura 2).
Para determinar la sensibilidad se probaron diferentes cantidades de ADN desde 100 ng. a 1µg. siendo la sensibilidad de hasta 1 a 10 µg que corresponde de 1 a 10 copias de virus circulante.
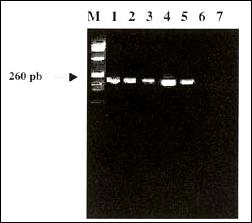
Las condiciones para ser usadas en PCR fueron aplicadas usando la temperatura de anillamiento de los primers fue de 55°C, la concentración de MgCl2 óptima fue de 2 mM. En la estandarización de la técnica se pudo determinar que PCR tiene una sensibilidad alta de aproximadamente detecta hasta 10 partículas virales circulantes y la especificidad fue de un 100%. Además se realizó el PCR de 6 sueros de pacientes infectados, teniendo resultados positivos (figura 3).
Se concluye que esta técnica será de mucha ayuda en el diagnóstico y pronóstico de la enfermedad así como del seguimiento al paciente en caso de ser un portador crónico. Del mismo modo podría emplearse en estudios epidemiológicos de la enfermedad.
Figura 1: Estandarización de la Temperatura de Anillamiento.
M, marcador de peso molecular para fragmentos de ADN (Phi X174/Hae III);
1, control positivo 1; 2, control positivo 2; 3, control negativo
(PCR a suero de persona sana); 4, Control del sistema
(PCR sin muestra sólo con agua tridestilada).
La temperatura empleada es de 55°C.
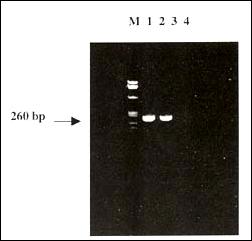
Figura 2. Estandarización de la concentración de MgCl2.
M, marcador de peso molecular para fragmentos de ADN
(Phi X174/Hae III); 1,4 mM de MgCl2; 2,3 mM de
MgCl2; 3,2.5 mM de MgCl2; 4,2 mM de MgCl2;
5,1.5 mM de MgCl2; 6,1 mM de
MgCl2; 7,0 de MgCl2.
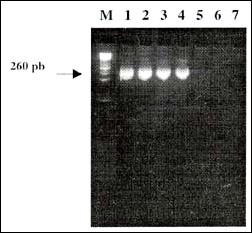
Figura 3. PCR en pacientes confirmados con HVB.
M, marcador de peso molecular para fragmentos de ADN
(Phi X174/Hae III); 1, control positivo; 2-5, pacientes
positivos para HBsAg y HBeAg; 6, control negativo
(PCR a una extracción de suero de persona
sana); 7, control de sistema (PCR sin
muestra solo con agua tridestilada).
DISCUSIÓN
La presencia del ADN del virus de la hepatitis B (HVB) en suero indica la infectividad y la replicación del virus9. El empleo de las últimas técnicas moleculares como Southern o spot-blot hibridación utilizando marcaje radiactivo detectan 0,01-1,0 pg de ADN, que equivale a 3 x 103 a 3 x 10 5 copias de HVB, respectivamente. Sin embargo, métodos más sensibles9 detectan 3 x 106 pg. La PCR es una técnica que nos permite amplificar fragmentos de ADN dando una sensibilidad de 1 a 10 copias y se aumenta la sensibilidad empleando después una hibridación con una sonda marcada.11
En una infección con Hepatitis Viral B (HVB) el marcador más sensible es la detección el ADN del virus en suero ya que indica la replicación y la infectividad del virus10.
En el estado crónico de esta enfermedad la técnica de PCR sería muy importante ya que podría determinar si el paciente está en una fase replicativa, lo cual ayudaría al tratamiento del paciente y al pronóstico de la enfermedad. Con ésta técnica se podrían realizar estudios de monitoreo de la respuesta inmunológica del paciente infectado, así como detectar con la presencia de ADN circulante en casos de reactivación del virus y por su alta sensibilidad determinar los casos donde el paciente se encuentre en el periodo de ventana.
REFERENCIAS BIBLIOGRÁFICAS
1. Cabezas Sánchez C. (1993). Prevalencia de marcadores serológicos para Hepatitis Viral A, B y Delta en escolares aparentemente sanos en Huanta. Tesis para optar el título de Especialista en Enfermedades Infecciosas y Tropicales. Universidad Peruana Cayetano Heredia. [ Links ]
2. Cabezas Sánchez, C. (1997). Epidemiología de las hepatitis virales (HVB) y Delta (HVD) en el Perú. Revista de Medicina Experimental del INS. Segunda época Volumen XIV Nº 1. [ Links ]
3. Cabezas Sánchez C. (1997). Prevalencia de marcadores serológicos para hepatitis viral B y delta en pueblos indígenas de la Amazonía Peruana. Convenio AIDESEP/INS. [ Links ]
4. Carman M. (1990). Genetically defined variants of hepatitis B virus. Reviews in Medical Virology 1, 29-39. [ Links ]
5. Courouce A M, Holland P, Muller J Y and Soulier. (1976). HBs antigen subtypes Karger. Basel. J, p. 158 S. [ Links ]
6. Gastelumendi R., Burstein M., León Barua R. (1960). Contribución al estudio de hepatitis viral endemo-epidémica en Abancay. Ann de la Fac. de Med. XLII: 629-650. [ Links ]
7. Indacochea S. Gotuzzo E., De la Fuente J., Phillips I., Whignal S. (1991). Elevada prevalencia de hepatitis B y Delta en el Valle interandino de Abancay. Revista Medica Herediana 2(4); 168-71. [ Links ]
8. Instituto Nacional de Salud. (1996). Prevalencia de marcadores serológicos para Hepatitis viral B y Delta en Pueblos Indígenas de la Amazonía Peruana. Auspicio: Cooperación Española Cooperación Instituto, Nacional de Salud Amazónica Ejecución: Convenio AIDESEP/INS del 16/09/96. [ Links ]
9. Saiki RK, Scharf S, Faloona F, et al (1985) Enzymatic amplification of b-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia. Science 230: 1350-1354. [ Links ]
10. Scotto J., Hadchouel M, Hery C, et al. (1983). Detection of hepatitis B virus DNA in serum by a simple spot hybridization technique: comparison with results for other viral markers. Hepatology 3,279-284. [ Links ]
11. Yokosuka O, Omata M, Hosoda K, Tada M, Ehata T, and Hosoda O. (1991). Detection and direct secuencing of hepatitis B Virus Genome by DNA Amplification Method Gastroenterology 100(1): 175-181. [ Links ]














