Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.15 n.1-2 Lima ene./dic. 1998
ARTÍCULOS CIENTÍFICOS
Detección de Bartonella bacilliformis usando PCR-RFLP
Padilla C 1; Carrillo C 2; Barbara Ellis 3; Ventura G 4 y Montoya Y 1.
1 División de biología Molecular del Instituto Nacional de Salud, Lima-Perú.
2 Jefe del Instituto Nacional de Salud, Lima-Perú
3 Center for Disease Control and Prevention (CDC), USA.
4 División de Bacteriología Instituto Nacional de Salud, Lima-Perú
RESUMEN
Reportamos la aplicación de las técnicas moleculares PCR-RFLP para la confirmación de infecciones por Bartonella bacilliformis. El método de PCR-RFLP se basa en la amplificación in vitro de un fragmento de 380 pb correspondiente al gen citrato sintetasa a partir de sangre y cultivos in vitro. El análisis del producto de amplificación por cortes con las enzimas restricción Hinfl y Taql permite caracterizar molecularmente que el brote ocurrido en Urubamba, Cuzco fue producido por Bartonella bacilliformis. Este método es aplicado directamente a sangre y a cultivos.
Palabras claves: Bartonella, PCR-RFLP
ABSTRACT
We reported the standarization and aplication of PCR-RFLP for confirmation of patients infected with Bartonella bacilliformis. This method is based in PCR amplification of 380 pb fragment corresponding of citrato sinthetase gene using whole blood and in vitro culture. The analysis of fragment amplification with endonuclease restriction Hinfl and Taql allow tipify when outbreak is caused for Bartonella bacilliformis.
Key word: Bartonella, PCR-RFLP
INTRODUCCIÓN
Barlonella bacilliformis es el agente etiológico de la bartonelosis o enfermedad de Carrión, una enfermedad que se presenta con dos diferentes síndromes: la fiebre de la Oroya y la verruga peruana. Esta enfermedad es transmitida por la picadura del mosquito hematófago del género Lutzomya.
La bartonellosis afecta principalmente los departamentos de Ancash, Cajamarca, Amazonas y La Libertad; en menor incidencia la sierra de Lima, Huancavelica y Huánuco, Piura, Cuzco y Ayacucho1.
A nivel mundial cuatro especies de Bartonella causan enfermedad al hombre: B. bacilliformis, B. quintana, B. henselae y B. elizabethae. Además, existen muchas otras especies no patógenas al hombre como: B. vinsonii, B. vinsonii berkhoffii, B. clarridgeiae, B. talpae, B. peromysci, B. grahamii, B. taylorii y B. doshiae2. En el Perú no existen estudios ni reportes de especies diferentes a Bartonella bacilliformis.
MATERIALES Y MÉTODOS
Cepas bacterianas y muestras sanguíneas El ácido desoxirribonucleico (ADN) obtenidas de cepas patrones Bartonella bacilliformis y Bartonella vinsonii fueron proporcionadas por el Centro para el Control de Enfermedades (CDC), los cuales fueron utilizados para estandarizar el método.
El ADN purificado de Brucella melitensis cepa referencial Rev 1 y ocho aislamientos de Brucella peruanos fueron utilizados como controles de especificidad. Además 4 aislamientos de Bartonella sp y 16 muestras sanguíneas con anticoagulante EDTA fueron recolectadas durante el brote de bartonellosis ocurrido en Urubamba, Cuzco en Mayo de 1998.
Extracción de ADN
Para la extracción de ADN a partir de sangre y aislamientos de cultivo in vitro se utilizó el método fenol/cloroformo6. Brevemente, 200 microlitros de sangre total o suspensión bacteriana fue centrifugada y el sedimento es resuspendido en una solución de lisis conteniendo proteinasa K e incubado a 65°C por 1 hora. Las muestras fueron tratadas con fenol-cloroformo y el ADN del sobrenadante fue precipitado con etanol y resuspendido en agua.
Reacción en cadena de la Pololimerasa (PCR)
Cincuenta nanogramos de ADN purificado es sometido a PCR, utilizando oligonucleótidos dirigidos al gen de la citrato sintasa de Bartonella (proporcionados por la Dra. Barbara Ellis) y la ADN Polimerasa termoestable, bajo las siguientes condiciones: una denaturación inicial de 95°C por 10 minutos; 35 ciclos de 95°C por 30 segundos, 45°C por 45 segundos, 72°C por 45 segundos; y una extensión final de 72°C por 10 minutos. La amplificación es observada en geles de poliacrilamida al 10% y teñidos con bromuro de etidio.
Corte del ADN amplificado
Diez microlitros del producto de amplificación fueron sometidos a digestiones con endonucleasa de restricción de acuerdo a las especificaciones recomendadas por sus fabricantes. Dos enzimas de restricción fueron usadas: TaqI y HinfI. El patrón de cortes es determinado por geles de poliacrilamida al 10%. Los geles fueron teñidos con bromuro de etidio y fotografiados.
RESULTADOS
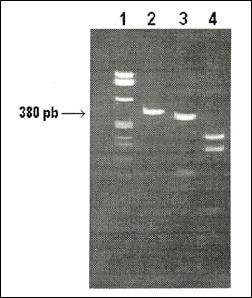
Parte del gen de la citrato sintasa es amplificado por PCR (Figura 1). El tamaño del producto de amplificación es de 380 pb aproximadamente como se esperaba. Es importante mencionar que el sistema es específico porque no se obtiene ningún producto de amplificación con ADN de Brucella. Por otro lado, el análisis del producto de amplificación cortado con las enzimas de restricción indica un patrón análogo con B. bacilliformis (Figura 1).
Cinco muestras sanguíneas de pacientes con síntomas clínicos de bartonellosis presentan el producto de amplificación esperado al aplicar PCR. Asimismo dos casos más fueron confirmados a partir de ADN extraído de aislamiento de cultivo in vitro.
Figura 1: Diagnóstico de Bartonella bacilliformis usando PCR-RFLP.
Carril 1: Marcador de Peso Molecular PhiX174/Hae III;
carril 2: Producto de amplificación del gen citrato
sintasa; carril 3: producto de amplificación
cortado con TaqI; carril 4: producto
de amplificación cortado con HinfI.
DISCUSIÓN
El diagnóstico clínico de bartonellosis en las zonas rurales es difícil e inespecífico. Varios métodos por PCR para el diagnóstico de Bartonella 4,5,6 han sido desarrollados. Sin embargo debe ser destacado que usando PCR-RFLP dirigido al gen citrato sintasa es posible diferenciar las especies de Bartonella. Aplicar este método directamente de muestras sanguíneas permite un diagnóstico rápido (2 días) que durante un brote es fundamental.
También debe ser indicado que este método permite confirmar la identidad de los aislamientos por cultivo in vitro, ya que no es posible esta identificación por bioquímica.
Los productos de amplificación de los aislamientos han sido clonados, secuenciados y su análisis esta siendo realizado exitosamente.
Asimismo, es interés de nuestra institución aplicar la técnica de PCR al estudio de hospederos vertebrados y además intentaremos detectar precozmente a este patógeno en poblaciones de mosquitos.
REFERENCIAS BIBLIOGRÁFICAS
1. MINSA. Agosto de (1998) Doctrina, Normas y Procedimientos para el Control de la Bartonelosis o enfermedad de Carrión en el Perú. [ Links ]
2. Regnery R L, Anderson B E, Clarridge J E, Rodríguez-Barradas M C, Jones D C, Carr J H. (1992). Characterization of novel Rochalimaea species, R henselae sp. nov., isolated from blood of a febrile, human immunodeficiency virus-positive patient. J Clin Microbiol 30: 265-274. [ Links ]
3. Sambrook J, Fritsch E F, Maniatis T. (1989). Molecular Cloning, A Laboratory Manual. Segunda Edición. Cold Spring Harbor Laboratory Press. [ Links ]
4. Anderson B, Sims K, Regnery R, Robinson L, Schmidt M J, Goral S, Hager C and Edwards K. (1994) Detection of Rochalimaea hensenlae DNA in specimens from cat scratch disease patients by PCR. J Clin Microbiol 32(4): 942-948. [ Links ]
5. Matar G M, Swaminathan B, Hunter S, Slater L N, and Welch D F. (1993). Polymerase chain reaction-based Restriction Fragment Lenght Polymorphism Analysis of a fragment the ribosomal operon from Rochalimaea species for subtyping. J. Clin. Microbiol. 31(7): 1730-1734. [ Links ]
6. Regnery R, Spruill C L and Plikaytis B D. (1991). Genotypic identification of rickettsiae and estimation of intraspecies sequence divergence for portions of two rickettsial genes. J. Bacteriol. 173(5): 1576-1589. [ Links ]














