Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.16 n.1-2 Lima 1999
ARTÍCULOS ORIGINALES
Diagnóstico temprano del Virus Dengue 1 usando RT-PCR y perspectivas para la caracterización molecular de Cepas Autóctonas
Yábar C1; Carrillo C3; Nolasco O1; García M2; Montoya Y.1
1 División de Biología Molecular, Centro Nacional de Laboratorios en Salud Pública, Instituto Nacional de Salud.
2 División de Virología, Centro Nacional de Laboratorios en Salud Pública, Instituto Nacional de Salud.
3 Laboratorio de Microbiología, Universidad Peruana Cayetano Heredia.
RESUMEN
Un sistema de diagnóstico para la detección temprana del virus Dengue 1 fue llevado a cabo exitosamente usando la reacción en cadena por polimerasa de transcriptasa reversa (RT-PCR), a través de la amplificación de una porción genómica del gen NS1. Los resultados obtenidos, a partir de muestras clínicas, corroboraron los datos de anteriores trabajos de RT-PCR dirigidos hacia la región estructural del virión. Posteriormente el ADNc del virus Dengue, correspondiente a una de las muestras serológicas, fue clonado y secuenciado. La comparación por análisis de secuencia nucleotídica con otras cepas referenciales determinó que la cepa viral correspondía al serotipo 1.
Palabras clave: Dengue, diagnóstico, reacción en cadena por polimerasa de transcriptasa reversa.
ABSTRACT
A diagnosis system for the early detection of Dengue virus was carried out by amplification of the NS1 gene portion using RT-PCR. Results obtained from clinical samples corresponded to previous RT-PCR works aimed at the structural region of the virion. Dengue cDNA from one of the sera samples was then cloned and sequenced. Comparison by nucleotide sequence analysis with other reference strains determined that this viral strain was Dengue serotype 1.
Key words: Dengue, diagnosis, reverse transcriptase polymerase chain reaction.
INTRODUCCIÓN
El dengue es una enfermedad causada por un virus perteneciente a la familia Flaviviridae, transmitido al hombre a través de la picadura del mosquito Aedes ægypti1. Existen cuatro serotipos antigénicamente distintos denominados DEN1, DEN2, DEN3 y DEN41.
La infección por el virus Dengue afecta a más de 100 países a nivel mundial, por lo que ha sido considerado un problema de salud pública2. Es importante mencionar que la presencia de dos serotipos diferentes de dengue en una misma región pone en riesgo a la población de contraer Fiebre hemorrágica (FHD) o Síndrome del shock (SSD) por Dengue3. En el Perú, la presencia de los serotipos 1 y 2 ha sido determinados recientemente en la selva norte y central, así como también en la costa norte del país4. Por otro lado, es importante señalar que el DEN 1 ha incrementado significantemente su área de infección en nuestro territorio, principalmente en los departamentos de Tumbes, Piura, Lambayeque, Loreto, San Martín, Ucayali y Junín4. Por esta razón, es necesario mantener una constante vigilancia epidemiológica de este serotipo con el fin de evitar algún brote de tipo hemorrágico.
El virus del dengue presenta un genoma de ARN de cadena simple y de sentido positivo, que tiene un tamaño de aproximadamente 11kb, y codifica para una simple poliproteína, que sufre sucesivos cortes para generar proteínas virales individuales5. Las tres proteínas estructurales C, M y E están localizadas en el extremo aminoterminal, mientras que las proteínas no estructurales NS1, NS2A, NS2B, NS3, NS4A, NS4B, NS5, están en el extremo carboxilo de la poliproteína5.
Con respecto a los sistemas de diagnóstico para la detección de dengue en nuestro país, han sido estandarizados el cultivo celular y la prueba serológica de MAC-ELISA. Sin embargo, el desarrollo del efecto citopático, como evidencia de la presencia del virus en el cultivo celular, requiere un tiempo prolongado. Por otro lado, la frecuencia de reacción cruzada en el MAC-ELISA, hace a este diagnóstico no muy específico para efectos de obtener un resultado confirmatorio. Mediante el sistema de RT-PCR se ha podido diagnosticar dengue desde el primer día de enfermedad6.
Este artículo informa sobre la validación de un sistema alternativo de RT-PCR para la detección de dengue tipo 1 a partir de muestras clínicas, dirigiendo la especificidad de los oligonucleótidos hacia la región no estructural del virus (gen de la glicoproteína NS1)7. Mediante este sistema se espera obtener los mismos parámetros de especificidad y sensibilidad de los oligonucleótidos dirigidos hacia la región estructural del virión6 y, de esta manera, contar con otro sistema opcional de RT-PCR para el diagnóstico temprano de dengue.
MATERIALES Y MÉTODOS
MATERIAL SEROLÓGICO
Un grupo de 60 muestras serológicas fueron obtenidas durante un brote epidémico ocurrido en Máncora-Piura. Los sueros fueron clasificados en dos grupos: el primero correspondió a diez controles negativos de personas sanas, mientras que el segundo de cincuenta muestras de sueros positivos de personas con diagnóstico clínico y serológico de dengue. Todas estas muestras fueron evaluadas previamente por MAC-ELISA y RT-PCR y tipificadas como dengue 1 por IFI y Nested PCR6.
CULTIVOS VIRALES
Las cepas referenciales del virus dengue 1 (Hawaii), Dengue 2 (New Guinea), dengue 3 (H-87) y dengue 4 (H- 241) para los controles positivos, y las cepas referenciales de flavivirus, relacionadas con Fiebre amarilla y Virus de West Nile, para el control de especificidad, fueron incubadas en líneas celulares de mosquito Aedes albopictus C6/36 a 28 0C en un medio mínimo esencial (MEM).
RT-PCR
El genoma viral fue extraído y purificado de acuerdo a métodos previamente descritos. Para la reacción de la transcripción reversa se utilizó el oligonucleótido reverso denominado AD37. Asimismo, se incluyó la enzima transcriptasa reversa Rav-2 (Amersham Pharmacia R ) para luego someter la mezcla de reacción a una incubación en baño María por 1h a 42 0C.
La reacción en cadena de la polimerasa se llevó a cabo utilizando los cebadores AD3 y AD47, específico para una región ubicada en la posición 3 400 del gen de la glicoproteína NS1, así como también la enzima Taq Gold ADN Polimerasa (Perkin ElmerR). La reacción se llevó a cabo en un Termociclador Perkin Elmer 9600 y las condiciones de PCR fueron realizadas de acuerdo a Henchal y col7. con algunas modificaciones: Denaturación inicial a 95ºC por 6 minutos, 30 ciclos a 94°C por 1minuto, 45°C por 1 minuto, y 72°C por 1 minuto, para luego concluir la reacción con una extensión final a 72°C por 7 minutos. Posteriormente 5 µl de producto de PCR fueron resueltos en minigeles de agarosa al 1,5% y visualizados en luz UV, después de una previa incubación en Bromuro de Etilio.
CLONACIÓN DE LOS PRODUCTOS DE AMPLIFICACIÓN
Los productos de amplificación fueron purificados, cuantificados y ligados a un vector plasmídico pGEMT- Easy (PromegaR). Una línea celular de bacterias electrocompetentes JM109 (StratageneR) fue transformada con el plásmido recombinante mediante electroporación. Los clones recombinantes fueron seleccionados de acuerdo a un ensayo colorimétrico utilizando X-gal e IPTG, luego fueron incubados en caldo LB ampicilina y el ADN plasmídico fue purificado y cuantificado para ser sometido a la reacción de secuenciamiento.
SECUENCIAMIENTO
Los plásmidos recombinantes, conteniendo el inserto de ADN de interés, fueron sometidos a la reacción de secuenciamiento mediante el método dydeoxi utilizando primers marcados. La reacción se llevó a cabo utilizando el Sistema Autocycle mediante el uso de la enzima Taq Polimerasa. El secuenciamiento de ADNc se realizó en un equipo de secuenciamiento ALF express mediante electroforesis por 10 horas.
RESULTADOS
RT-PCR
El producto esperado correspondió a una banda de ADN de 419 pb, tal como se muestra en la Figura 1.La concentración óptima de MgCl fue de 2 mM, luego de la cual ocurrió una disminución en la intensidad de la banda (ver Figura 2). El ARN viral de la cepa Hawai correspondiente al serotipo 1 fue exitosamente amplificado. Para determinar la efectividad del sistema a partir de suero, una muestra clínica correspondiente a un paciente, con sospecha de dengue 1, fue evaluada obteniéndose satisfactoriamente la banda de diagnóstico esperada. Es importante resaltar que estas muestras de cultivo y de suero de dengue 1 fueron evaluadas antes por Nolasco y col6 obteniéndose en este trabajo los mismos resultados de reactividad.
Asimismo, la ausencia de la banda en el control negativo y cepas referenciales de otros flavivirus relacionados, corrobora la especificidad del sistema.
SECUENCIAMIENTO DEL PLÁSMIDO RECOMBINANTE
Luego de haber clonado el producto de PCR en el plásmido pGEMT-easy, el inserto de ADNc fue sometido a un proceso de secuenciación utilizando un secuenciador automático ALF Express. En ese sentido, una lectura de aproximadamente 100 pares de bases con 6 ambigüedades en la hebra positiva fue obtenida durante esta reacción, mientras que por el sector de la hebra complementaria, una lectura máxima de casi 475 pb con 5 ambigüedades pudo ser determinada.
DISCUSIÓN
El diagnóstico del dengue mediante RT-PCR ha sido desarrollado por diversos autores7-11. Estos estudios demostraron que, mediante esta técnica, el ARN viral es detectado en casi un 90% y que, además, los métodos inmunoenzimáticos alcanzan este porcentaje de sensibilidad6.Con respecto a las pruebas inmunológicas, el desarrollo de un nuevo Kit de diagnóstico conocido como Dip-S-Ticks® ha mejorado el sistema de diagnóstico para la detección de anticuerpos IgM frente al virus dengue. Sin embargo, la especificidad y sensibilidad de cualquier sistema inmunoenzimático no es compararable con el RT-PCR, durante los primeros cinco días de la enfermedad6,12. Empero, el uso combinado de estas dos técnicas facilitaría grandemente la detección temprana de la enfermedad del dengue, disminuyendo el riesgo de obtener resultados indeterminados o reacciones cruzadas.
Por otra parte, el secuenciamiento de ADNc ha permitido corroborar la especificidad y sensibilidad de los oligonucleótidos universales AD3 y AD4, los cuales lograron amplificar una región de 419 pb del gen de la glicoproteína NS1 del virus dengue 1 autóctono de Máncora-Piura (ver Figura 2). Asimismo, muestras de ARN viral extraídas a partir de cultivos celulares de dengue 2, autóctono de la selva norte del país, han sido exitosamente reconocidas por estos cebadores, lográndose obtener la banda esperada de 419 pb (datos no mostrados). La secuencia completa de esta región de NS1 en el virus Dengue 1 fue recientemente registrada en el Banco de Genes13 y en la actualidad está siendo analizada y comparada con otras cepas referenciales de dengue. Las secuencias de nucleótidos y aminoácidos permitirán la realización del análisis molecular de esta región de NS1 y sus implicancias en la virulencia del virus.
Figura 1. Especificidad de los productos de amplificaciónde 419 pb bases correspondiente
al gende la glicoproteína NS1 del virus dengue 1. Carril1: Marcador de peso
molecular ØX174. Carril 2:Control negativo suero normal. Carril 3: Controlpositivo dengue 1 referencial,
cepa Hawaii. Carril4: dengue 1 suero autóctono de Máncora-Perú.Carril 5: Virus de
la fiebre amarilla. Carril 6: Virusde West Nile. Carril 7: Control del Sistema.
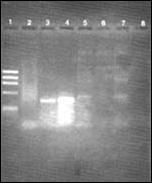
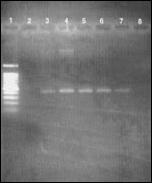
Figura 2. Curva de MgCl 2 de los productos de amplificación de 419 pb bases correspondiente al
gen de la glicoproteína NS1 del virus dengue 1.Carril 1: Marcador
de peso molecular Ladder. Ca-rril2 : 0 mM. Carril 3: 1 mM. Carril 4:
2 mM. Carril5 : 3mM. Carril 6: 4mM. Carril 7: 5mM.
AGRADECIMIENTOS
Agradecemos al CDC de Atlanta, EEUU, por proporcionarnos las cepas referenciales del virus dengue 1 (Hawaii),dengue 2 (New Guinea), dengue 3 (H-87) y dengue 4 (H-241).
A los señores técnicos Juana Choque Portilla y David García Neyra, de la División de Biología Molecular por su entusiasta colaboración.
REFERENCIAS BIBLIOGRÁFICAS
1. Gubler DJ. Dengue and dengue hemorragic fever. Clin Microbiol Rev 1998; 11: 480-6. [ Links ]
2. World Health Organization. The World Health Report 1996. Fighting disease fortering development. Geneva, 1996. [ Links ]
3. Halstead S. Pathogenesis of dengue. Challenge to molecular biology. Science 1988; 239: 76-81. [ Links ]
4. García M, Cabezas C, Callahán J, Yana B, Gutiérrez V, Ortiz A, Anaya E. Determinación de IgG y anticuerpos totales contra el virus dengue, en muestras obtenidas en papel filtro. Rev Med Exp INS 1997; 14 : 45-9. [ Links ]
5. Chambers T, Hahn C, Galler R, Rice C. Flavivirus genome organization, expression, and replication. An Rev Microbiol 1990;. 44: 649-88. [ Links ]
6. Nolasco O, Carrillo C, Gutiérrez V, Yábar C, Douglas S, García M, Montoya Y. Diagnóstico temprano en un brote epidémico del virus dengue en Piura usando RT-PCR y Nested-PCR. Rev Med Exp INS 1997; 14: 13-17. [ Links ]
7. Henchal E, Polo SL, Vorndam V, Yaemsiri C, Innis BL, Hoke CH. Sensitivity and specificity of a universal primers set for the rapid diagnosis of dengue virus infections by polimerase chain reaction and nucleic acid hybridization. Am J Trop Med Hyg 1991; 45: 418-28. [ Links ]
8. Lanciotti R, Calisher CH, Gobler DJ, Chang GJ, Vordman AV. Rapid detection and typing of dengue viruses from clinical samples by using reverse transcriptase-polimerase chain reaction. J Clin Microbiol 1992; 30: 545-51. [ Links ]
9. Puri B, Henchal EA, Burans J, Porter KR, Nelson W, Watts DM, et al. A rapid method for detection and identification of flaviviruses by polimerase chain reaction and nucleic acid hybridization. Arch Virol 1994; 134: 29-37. [ Links ]
10. Pierre V, Drouet MT, Deubel V. Identification of mosquito-borne flavivrus sequences using universal primers and reverse transcription/ polymerase chain reaction. Res Virol 1994; 145: 93-104. [ Links ]
11. Tadeu M, Chelli W, Kashima S, Da Silva E. Identification of brazilian flaviviruses by a simplified reverse transcription-polymerase chain reaction method using flavivirus universal primers. Am J Trop Med Hyg 1998; 59: 357-62. [ Links ]
12. Innis B, Nisalak A, Nimmannitya S, Kusalerdchariya S, Chongswasdi V, Suntayakorn S, et al. An enzyme-linked inmunosorbent assay to characterize dengue infections where dengue and japanese encephalitis co-circulate. Am J Trop Med Hyg 1989; 40: 418-27. [ Links ]
13. Yábar C, Carrillo C, Nolasco O, García M, Montoya Y. Partial genomic analysis of the glycoprotein of the dengue virus type1 from Mancora-Piura, 1998. Access number AFO97020. [ Links ]
Correspondencia: Carlos Yábar. Instituto Nacional de Salud.
Calle Cápac Yupanqui 1400, Lima 11, Perú. Apartado postal 471.
Telf.: (0511) 4719920 - Fax: (0511) 4710179.
Email: biomole@ins.sld.pe














