Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.19 no.1 Lima Jan./mar 2002
TRABAJOS ORIGINALES
Análisis genético del virus peruano de la fiebre amarilla
Carlos Yábar V1; Yván Campos B2; Kelly Quispe T3; Carlos Carrillo P4; Ysabel Montoya P1.
1 División de Biología Molecular, Centro Nacional de Salud Pública, Instituto Nacional de Salud, Lima – Perú.
2 St. Jude Childrens Research Hospital, Memphis – USA.
3 Prince Leopold Institute of Tropical Medicine. Antwerp - Belgium.
4 Universidad Peruana Cayetano Heredia, Lima - Perú.
RESUMEN
Objetivo: Determinar las variantes genéticas de aislamientos del virus peruano de la Fiebre Amarilla (FA). Materiales y métodos: la región carboxiterminal del gen de la envoltura (E) de cinco aislamientos de FA obtenidas de pacientes provenientes de Ayacucho 1978 (PER1), Junín 1995 (PER2), Cerro de Pasco (PER3), Cusco (1998) y San Martín (1999) fue amplificada por PCR, secuenciada y analizada con programas software de ADN. Resultados: el índice de similaridad de la secuencia de nucleótidos entre los cinco aislamientos reveló valores oscilantes entre 94,3% y 99,3%, mientras que la secuencia de aminoácidos presentó valores entre 97,6% y 99,7% de similaridad. El análisis filogenético demostró una distancia genética entre 0,40 y 6,50 mediante la secuencia de nucleótidos y a través de la secuencia de aminoácidos se observó un rango de 0,30 y 4,29. Sin embargo, las secuencias correspondientes a los sitios de glicosilación y a los epítopes de reconocimiento humoral fueron conservadas entre los cinco aislamientos, con excepción de algunos aislamientos de referencia reportados por otros autores. Conclusiones: los virus de FA peruanos forman un grupo filogenético distinto a otros virus de FA sudamericanos, basados en el análisis genéticos del gen E.
Palabras clave: Fiebre amarilla / genética; Filogenia; Perú (fuente: BIREME).
ABSTRACT
Objective: To determine genetic variants among peruvian Yellow Fever (YF) isolates. Materials and methods: Carboxiterminal ends from the envelope protein gene of five YF isolates obtained from patients living in Ayacucho 1978 (PER1), Junin 1995 (PER2), Cerro de Pasco (PER3), Cusco (1998) y San Martín (1999) were amplified using PCR, and they were sequenced and analyzed using DNA software. Results: The identity index using nucleotide sequences among five yellow fever isolates showed values between 94,3% and 99.3%, while amino acid sequences revealed 97,6% to 99,7% similarity. Likewise, the phylogenetic analysis showed that the values for genetic distance among these isolates were between 0,40 y 6,50 using nucleotide sequences; and using aminoacid sequencing, the range was from 0,30 to 4,29. However, important regions, such as glycosilation sites and humoral-recognising sites were preserved among the five isolates excepting some reference isolates previously reported. Conclusions: Peruvian Yellow Fever viruses are grouped in a phylogenetic cluster different to other South American YF viruses, based on the E gene analysis.
Key words: Yellow fever/genetics; Phylogeny; Peru (source: BIREME).
INTRODUCCIÓN
La Fiebre Amarilla (FA) es una enfermedad febril hemorrágica transmitida al hombre a través de la picadura de mosquitos de los géneros Aedes o Haemagogus. Distribuida tanto en África como en Sudamérica, esta enfermedad es causa de muerte de aproximadamente 30 mil personas en más de 34 países1.
En el Perú, los casos de FA han sido confirmados desde los años 70. En 1995, los casos experimentaron un dramático aumento, llegando a reportarse 440 casos con una tasa de mortalidad de 38%2 y seguido de una disminución favorable de casos confirmados en los últimos años.
El agente responsable de la FA es un virus con envoltura perteneciente al grupo de los flavivirus. Presenta un genoma de ARN de aproximadamente 10862 pb, el cual está conformado por diez genes que codifican una poliproteína. Después de un proceso de maduración, la poliproteína da lugar a tres proteínas estructurales y siete no estructurales 3,4.
Una de estas proteínas estructurales es constituyente principal de la envoltura del virus y se conoce como glicoproteína E, primer antígeno reconocido por los anticuerpos del hospedero y responsable del ingreso del virus en la célula hospedera5.
Diferentes estudios usando el gen de la proteína E, así como otros genes que codifican proteínas estructurales y no estructurales han demostrado la existencia de variantes genéticas del virus. Estas variantes han sido denominadas genotipos y están distribuidas por áreas geográficas alrededor del mundo6-10.
Con respecto al virus peruano de FA, Chang et al. (1995), clasificaron dos aislamientos de los años 1977 y 1981, ubicándolos dentro del genotipo IIB. Sin embargo, carecemos de información de los nuevos islamientos virales provenientes de brotes acontecidos durante los últimos años.
En el presente estudio, hemos realizado el análisis genético de cinco aislamientos del virus de la FA circulantes desde el año 1978 hasta 1999. Para tal propósito, hemos seleccionado una región de 1012 pb que codifica la porción carboxiterminal de la proteína E.
MATERIALES Y MÉTODOS
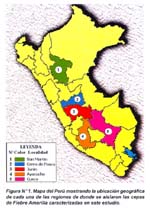
Se trabajó con cinco aislamientos virales proporcionados por la División de Virología del Instituto Nacional de Salud y que fueron obtenidos de sueros de pacientes. Estos aislamientos fueron asignados con códigos de acuerdo a la región geográfica de donde provenían y el año de aislamiento: Cusco 1998 (PER1), Junín 1995 (PER2), Cerro de Pasco (PER3), Ayacucho 1978 (PER4) y San Martín 1999 (PER5) (Figura Nº 1).
SECUENCIAS DE REFERENCIA PARA LA COMPARACIÓN GENÉTICA
Con el fin de realizar la comparación genética entre los aislamientos peruanos y otras cepas sudamericanas de FA se recurrió a la información que brinda el Banco de Genes del Instituto Nacional de Salud de los Estados Unidos, encontrándose dos únicos aislamientos peruanos de FA correspondientes a los años 1977 y 1981, así como también otros aislamientos procedentes de Ecuador, Colombia,
Trinidad y Brasil (Tabla N°1).
PROCEDIMIENTOS
Extracción de ARN
El ARN total fue extraído a partir de cultivos virales en células C6/36 usando el Kit Qiamp viral ARN Kit (QIAGEN ® ) de acuerdo a las recomendaciones del fabricante. En tal sentido, se mezcló 140 µL de muestra con 560 µL buffer de lisis AVL. Posteriormente, esta solución fue mezclada con etanol y purificada en una columna de afinidad. La mezcla total fue centrifugada a 8000 rpm durante un minuto. Las impurezas fueron removidas con sucesivos lavados de etanol. Finalmente, las moléculas de ARN fueron resuspendidas en agua pura de ARNasas y ADNasas para después ser almacenadas en congelamiento a -80°C hasta su posterior análisis.
Transcripción reversa - reacción en cadena de la polimerasa
La reacción de transcripción reversa y la reacción en cadena de la polimerasa (PCR) fueron realizadas mediante el uso de los primeros YF7 y YF2 bajo condiciones experimentales descritas previamente11. El producto final fue analizado por electroforesis y visualizado mediante tinción con bromuro de etidio.
Clonamiento y secuenciamiento
El producto de PCR fue purificado y clonado en un vector plasmídico pGEM-T easy (Promega). Posteriormente, el plásmido conteniendo la secuencia de interés fue introducido en E. coli cepa XL1 Blue por electroporación. Los clones recombinantes fueron purificados y secuenciados de acuerdo al método de Sanger et al., 1977 usando un secuenciador automático ALF Express (Pharmacia).
Análisis con la enzima de restricción Eco RI
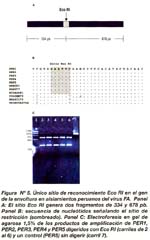
Aproximadamente 250 ng de ADN de producto de amplificación de los aislamientos PER1, PER2, PER3, PER4 y PER5 fueron añadidos en una solución conteniendo 150 mM KOAc, 37,5 mM Tris-acetato (pH 7,6), 0,75 mM β-mercaptoetanol, 15 mM MgOAc, 15 mg de albúmina sérica bovina (BSA) y 5 U de enzima de restricción Eco RI. La mezcla de reacción fue incubada a 37°C por una hora y posteriormente sometida a electroforesis empleando un gel de agarosa al 2%. A continuación, el gel de agarosa conteniendo cada uno de los insertos de ADN fue incubado en bromuro de etidio y luego expuesto a luz ultravioleta para la visualización de los productos digeridos.
Análisis de secuencias y árbol filogenético
Para la alineación de secuencias múltiples se utilizó el método de corrección de dos parámetros de Kimura mediante el programa PileUp del paquete GCG Wiscon-sin, el cual es una combinación de los métodos de Feng and Doolittle12 y Clustal V (http://www-igbmc.u-strasbg.fr/ Computer/ClustalW/clustalv.html)13.
Para el análisis de secuencias de aminoácidos, se realizó la traducción de la secuencia de nucleótidos usando la herramienta Translate del paquete software Expasy (/ /us.expasy.org/tools/dna.html).
Los datos de la alineación de secuencias también fueron utilizados para el cálculo de las distancias genéticas a través de la construcción de matrices. El valor de las distancias fue estimado por el número de sustituciones de cada 100 aminoácidos.
El análisis filogenético fue llevado a cabo mediante el método neighbor-joining14 usando el programa Growtree (Genetics Computer Group).
Todas las secuencias de nucleótidos y aminoácidos de los cinco aislamientos fueron reportados en el banco de genes (Ver Tabla Nº1).
RESULTADOS
Los aislamientos peruanos del virus FA presentaron una alta similaridad entre ellos; sin embargo, presentaron un menor índice frente a cepas sudamericanas de referencia
Los aislamientos peruanos PER1, PER2, PER3, PER4 y PER5 presentaron un índice de similaridad fluctuante entre 97,6% y 99,7%, en tanto que los valores a nivel de aminoácidos estuvieron en el rango de 94,3% y 99,3% (Tabla N°2).
Con respecto a las otras cepas peruanas reportadas anteriormente, observamos que la más alta identidad dentro de la secuencia de nucleótidos fue hallada entre PER4 y PERU77 con un valor de 99,0%; asimismo, la comparación entre las secuencias de aminoácidos reveló una alta similaridad (99,0%) entre los aislamientos PER1 y PERU81.
Al comparar las secuencias de nucleótidos de los aislamientos peruanos con cepas de referencia de otros países de Sudamérica, se observó una variación genética entre 86,0% y 98,0%, en tanto que los aislamientos peruanos PER4 y PERU81 presentaron una mayor identidad con el aislamiento ECUADO81 (98,0% y 99,0%, respectivamente).
De otro lado, el porcentaje de similaridad entre los aislamientos BRASIL79, COLOMB79, TRINIDAD79B y ECUADO81 a nivel de nucleótidos varió entre 89,0% y 97,0%, en tanto que a nivel de aminoácidos estuvo entre 96,0% y 99,0% (Tabla N°2).
El análisis filogenético permitió discriminar dos grupos filogenéticamente diferentes entre los aislamientos sudamericanos
Las secuencias genéticas conteniendo aminoácidos de cada aislamiento fueron alineadas y sometidas al programa Growtree para el cálculo de las distancias genéticas y el árbol filogenético De acuerdo a nuestros resultados, las distancias genéticas entre aislamientos peruanos presentaron valores fluctuantes de 0,40 a 6,50 mientras que entre los aislamientos peruanos y sudamericanos las variaciones fueron mayores (0,40 a 12,44) (Tabla N°3).
De manera interesante COLOMB79, TRINIDAD79B y BRASIL79 presentaron una distancia genética mucho menor (entre 2,10 y 3,64), comparado con los demás aislamientos sudamericanos.
Asimismo, se encontró una estrecha relación filogenética entre PER1, PER2 y PER3 con los aislamientos PERU77, PERU81 y ECUADO81. PER4 permaneció genéticamente más distante de todo ese grupo mientras, que la mayor distancia filogenética entre todos los aislamientos peruanos se observó en PER5, el cual dio origen a una adicional rama filogenética más distante en comparación con los demás aislamientos (Figura N°2).
Con relación a COLOMB79, TRINIDAD79B y BRASIL79, el árbol mostró de manera interesante la formación de un grupo filogenético totalmente separado de los aislamientos peruanos. Las distancias genéticas anteriormente calculadas permitieron deducir con mayor precisión la existencia de una estrecha relación filogenética entre estos aislamientos sudamericanos.
Todos los aislamientos sudamericanos presentaron dos sitios potenciales de glicosilación N-X-S/T con excepción de BRASIL79
El alineamiento de secuencias de aminoácidos de la región en estudio permitió encontrar dos potenciales sitios de glicosilación ligados al aminoácido asparagina (N) del tipo N-X-S/T (Donde s = serina y t = treonina) entre las posiciones 162 al 164 y 202 al 204 (Figura N°3). Es importante resaltar que el primero de ellos es del tipo NGS y estuvo presente en todos los aislamientos sudamericanos, excepto en BRASIL79 debido a un cambio de tipo conservado de N por S (posición 162).
La comparación de dominios y motivos antigénicos reveló un alto grado de conservación entre aislamientos sudamericanos
Los dominios antigénicos identificados en la proteína E del virus proveniente de ácaros (TBEV) conocidos como dominios I, II y III15 y los que fueron descritos más adelante como C, A y B en el virus Dengue16 fueron extrapolados a la secuencia aminoacídica de E en los aislamientos de FA analizados en este estudio. Este procedimiento fue realizado en virtud a la estrecha alta homología existente dentro de la secuencia de aminoácidos de E entre diferentes Flavivirus17 . En ese sentido, el dominio A fue localizado entre las posiciones 21 y 31, el dominio C entre los aminoácidos 22 y 73 y, finalmente, el dominio B entre las posiciones 242 al 256 del total de 337 aminoácidos de E analizados en este estudio (Figura N° 3).
La comparación de aminoácidos entre dichos dominios de los cinco aislamientos reveló un único cambio de tipo conservativo a nivel del dominio B (posición 255) en PER5. Con respecto a los demás aislamientos, PERU77 mostró un cambio no conservado dentro del dominio A (K41N), mientras que BRASIL79, TRINIDAD79B y COLOMB79 presentaron un cambio conservado del aminoácido ácido aspártico (D) por ácido glutámico (E) con respecto a los demás aislamientos (posición 253).
Asimismo, dos aminoácidos (Figura Nº 4) localizados dentro de motivos epitópicos discretos recientemente caracterizados por Ryman18 en el virus FA también fueron analizados en este estudio. De acuerdo a ello, ambos aminoácidos fueron localizados dentro del dominio A (posiciones 48 para D y 51 para T); sin embargo, no se observó ningún cambio a nivel de esta región.
Único sitio de restricción Eco RI en aislamientos peruanos
La secuencia de nucleótidos de todos los aislamientos estudiados nos permitió diseñar un mapa de restricción con todos los sitios de corte para enzimas de restricción. De acuerdo a ello, el mapa reveló un único sitio Eco RI localizado en las posiciones 191-195 y 536-541 en todos los aislamientos peruanos, con excepción de BRASIL79, TRINIDAD79B y COLOMB79. Este sitio teóricamente podría dar lugar a dos fragmentos de 678 y 334 pb cada uno. Para comprobar la existencia de Eco RI (191-195 y 536- 541) en los aislamientos analizados, los fragmentos de PCR de PER1, PER2, PER3, PER4 y PER5 fueron digeridos con la enzima Eco RI (Figura Nº 5).
DISCUSIÓN
El análisis filogenético en cinco aislamientos del virus de FA reveló la existencia de un único grupo filogenético conformado por aislamientos peruanos de FA. Las variantes peruanas, incluyendo las que fueron reportadas previamente, presentaron un rango de similaridad bastante alto, tanto a nivel de nucleótidos como de aminoácidos (más del 97,0%) en reciprocidad con sus distancias genéticas (entre 0,4 y 6,0). Estos índices aumentaron ligeramente al comparar los aislamientos peruanos con COLOMB79, TRINIDAD79B y BRASIL79. Como resultado de esta divergencia, se dio origen a dos filogenéticos distintos, permitiendo clasificar los aislamientos peruanos dentro de un grupo filogenético distinto a los demás sudamericanos.
El aislamiento de ECUADOR81 (Ecuador), el cual se encuentra incluido dentro del grupo peruano, representa ser una importante excepción entre los demás grupos sudamericanos. Según el análisis filogenético, ECUADO81 presentó una estrecha relación filogenética con PERU77 y PERU81. De acuerdo a algunas referencias, PERU81 correspondería a un aislamiento proveniente de Ayacucho19 , mientras que de PERU77 no se tienen mayores datos. Sin embargo, es muy poco razonable que un aislamiento de Ecuador presente una alta relación filogenética con un aislamiento originario del interior del país, debido a que ambas zonas geográficas se encuentran muy distantes. A ello podemos añadir los diferentes factores climáticos y meteorológicos que diferencian una zona de otra y que inciden directamente sobre la tasa de variabilidad de los virus. Prueba de ello, son las múltiples variantes genéticas reportadas entre virus de FA de Asia, África y América6-10. En ese sentido, podríamos afirmar que ECUADO81 es en realidad un virus peruano y no ecuatoriano como lo especifica Chang7.
Es importante señalar que los cinco aislamientos peruanos caracterizados en este estudio presentaron un gradual índice de similitud de acuerdo a su distancia geográfica. Así, tenemos que entre PER1, PER2, PER3 y PER4 (provenientes de regiones limítrofes, ver Figura N°1) existió una alta similaridad de 98,0% y 99,0% a nivel de nucleótidos y aminoácidos, mientras que PER5, localizado a una mayor distancia geográfica y con factores de humedad y clima ligeramente distintos, presentó una menor similaridad frente a ellos (94,0% y 97,0% a nivel de nucleótidos y aminoácidos). Estos datos son concordantes con la distancia geográfica y el grado de divergencia.
Sin embargo, cabe la posibilidad que estos índices de similaridad podrían presentar algún tipo de sesgo debido a que la mayoría de los cinco aislamientos corresponden a diferentes periodos de tiempo (entre 4 y 11 años de diferencia de haber sido aislados). Es importante resaltar que PER4 (Ayacucho 1978) presentó una identidad del 98,0% con PERU81 (Ayacucho 1981). Este dato sugiere que la divergencia genética podría estar más influenciada por la distancia geográfica y no por el tiempo en que aparecen las cepas virales.
De otro lado, el análisis comparativo de secuencias aminoacídicas nos permitió hallar posibles sitios de glicosilación dentro de la zona de estudio. Estos sitios fueron de la forma N-X-S/T y se han visto en algunos Flavivirus, pero en diferentes posiciones dentro de la proteína E20-23.
En el presente estudio hemos hallado dos posibles sitios de glicosilación a nivel de las posiciones 162-164 y 204- 206, que en todos los aislamientos sudamericanos fueron conservados, con excepción de BRASIL79. La relevancia de estas glicosilaciones en el virus FA no están muy bien dilucidadas, sin embargo se ha visto que en el virus Dengue4 la pérdida de un sitio glicosilación por la sustitución de isoleucina por treonina en la posición 155 alteraba la neurovirulencia en ratón20. Asimismo, en el virus de la encefalitis ligada a ácaros (TBEV por sus siglas en inglés), la remoción total de glicanos en la proteína E genera una desestabilización de la proteína, principalmente del dominio C24, mientras que en el virus de la encefalitis de San Luis produce posibles diferencias en los procesos de infección específica21. Para el caso de BRASIL79, no se tienen mayores datos sobre el comportamiento biológico de dicho aislamiento viral, sin embargo la posición 162-164 podría tratarse de un sitio alternativo de glicosilación desde que una variedad de aislamientos de África y Asia han perdido este sitio de glicosilación por cambios de tipos no conservativo (Datos no mostrados).
Del mismo modo, hemos analizado dominios antigénicos previamente definidos por otros autores dentro de la proteína E para determinar la presencia de alguna variación relevante. En ese sentido, la comparación de aminoácidos reveló un cambio de tipo conservativo en PER5(E255D) localizado dentro del dominio B. Asimismo, COLOMB79, TRINIDAD79B, y BRASIL79 presentaron en el mismo dominio un cambio conservativo D253E. Es importante señalar que los cambios de tipo conservativo se producen entre aminoácidos de una misma carga o grupo químico y, por lo tanto, debemos suponer que no generan mayor alteración en el plegamiento de las proteínas. Por el contrario, los cambios de tipo no conservativo generan cambios importantes y, con mayor trascendencia biológica, aparecen dentro de dominios funcionales. Existen reportes de comparación entre cepas atenuadas y virulentas del virus 17D de FA en las que se han observado cambios no conservados en sitios de reconocimiento al receptor del hospedero, relacionándolos con procesos de neurovirulencia25. Otros estudios más recientes demostraron por mutación dirigida que alteraciones dentro del motivo RGD desencadenan una disminución en el título viral en células infectadas26. En el caso del PERU77, hemos observado un único cambio de tipo no conservativo en la posición 41 (K- N). Este cambio se localiza dentro del dominio C, el cual contiene residuos de aminoácidos relacionados al tropismo y virulencia de diferentes flavivirus27. En ese sentido, es probable que esta alteración haya causado algún efecto en la virulencia de PERU77, sin embargo no existe información respecto a la gravedad de la sintomatología que podría haber generado este virus en el paciente afectado.
Finalmente, hemos localizado un sitio de restricción Eco RI el cual corta el gen de la glicoproteína E en dos fragmentos de 678 y 334 pb dentro del grupo de aislamientos peruanos, incluyendo PERU77, PERU81 y ECUADO81. Este sitio podría ser aprovechado para la caracterización de cepas peruanas sin necesidad de recurrir al secuenciamiento directo que resulta ser más caro y necesita el procesamiento de programas software de ADN para la caracterización molecular. Sin embargo, mayores análisis de restricción deberán ser realizados a partir de un mayor número de aislamientos de FA, a fin de comprobar la naturaleza conservada de este sitio de restricción.
En resumen, hemos demostrado la presencia de variantes genéticas del virus peruano de FA a partir de cinco aislamientos virales. Estas variantes se reflejan a partir de los índices de similaridad existentes entre los aislamientos peruanos, los cuáles tienen un rango variable y podrían estar relacionados a la distancia geográfica en que se encuentran separados un aislamiento con otro. Bajo dicho contexto, es necesario continuar con la caracterización de nuevos aislamientos peruanos de otras zonas endémicas del país, con el fin de discriminar otras variantes genéticas que puedan estar llegando desde otras localidades fuera del país, principalmente en zonas fronterizas. Estos sistemas nos permitirán más adelante encontrar mejores vías alternativas de vigilancia epidemiológica de FA en el Perú.
REFERENCIAS
1. World Health Organization. Yellow fever. Geneva: WHO; 1998. [ Links ]
2. Carrillo C. Situación de la FA en el Perú. En: Guía teórica sobre la Reunión de expertos. Estrategias de prevención y control de la FA. Riesgo de urbanización en las américas. Lima: INS; 1998. [ Links ]
3. Rice CM, Lenches E, Eddy SR, Shin SJ, Sheets RL, Strauss JH. Nucleotide sequence of yellow fever virus: implications for flavivirus gene expressions and evolution. Science 1985; 229: 726-33. [ Links ]
4. Chamber T, Hahn C, Galler R, Rice C. Flavivirus genome organization, expression and replication. Ann Rev Microbiol 1990; 44: 649-88. [ Links ]
5. Hung S-L, Lee P-L, Chen H-W, Chen L-K, Kao C-L, Kings C-C. Analysis of the steps involved in Dengue virus entry into host cells. Virology 1999; 257: 156-67. [ Links ]
6. Mutebi JP, Wang H, li li, Bryant J, Barrett, A. Phylogenetic and evolutionary relationships among Yellow fever virus isolates in Africa. J Virol 2001; 75(15): 6999–7008. [ Links ]
7. Chang G, Cropp B, Kinney R, Trent D, Gubler D. Nucleotide sequence variation of the envelope protein gene identifies two distinct genotypes of yellow fever virus. J Virol 1995; 69(9): 5773-80. [ Links ]
8. Lepiniec L, Dalgarno L, Huong VTQ, Monath TP, Digoutte JP, Deubel V. Geographic distribution and evolution of yellow fever viruses based on direct sequencing of genomic cDNA fragments. J Gen Virol 1994; 75: 417-23. [ Links ]
9. Deubel V, Digoutte JP, Monath TP, Girard M. Genetic heterogeneity of yellow fever virus strains from Africa and the Americas. J Gen Virol 1986; 67: 209-13. [ Links ]
10. Deubel V, Pailliez PJ, Cornet M, Schlesinger JJ, Diop M, Diop A, et al. Homogeneity among Senegalese strains of yellow fever virus. Am J Trop Med Hyg 1985; 34(5): 976-83. [ Links ]
11. Yábar C, Montoya Y. Síntesis in vitro de la proteína de la envoltura del virus peruano de la fiebre amarilla. Rev Med Exp 2001, XVIII (1-2): 9-13. [ Links ]
12. Feng DF, Doolittle RF. Progressive sequence alignment as a prerequisite to correct phylogenetic trees. J Mol Evol 1987; 25(4): 351-60. [ Links ]
13. Higgins DG, Sharp PM. CLUSTAL: A package for performing multiple sequence alignments on a microcomputer. Gene 1988; 73: 237-44. [ Links ]
14. Saitou N, Nei M. Neighbor-joining Method. Mol Biol Evol 1987; 4: 406-25. [ Links ]
15. Mandl CW, Guirakhoo F, Holzmann H, Heinz FX, Kunz C. Antigenic structure of the flavivirus envelope protein E at the molecular level, using tick-borne encephalitis virus as a model. J Virol 1989; 63(2): 564-71. [ Links ]
16. Roehrig JT. Immunochemistry of Dengue Virus. In: Gluber & Kuno(Ed). Dengue and dengue hemorrhagic fever. Colorado: Center for Disease Control and Prevention; 1999. p. 199-203. [ Links ]
17. Gritsun TS, Holmes EC, Gould EA. Analysis of flavivirus envelope proteins reveals variable domains that reflect their antigenicity and may determine their pathogenesis. Virus Res 1995; 35(3): 307-21. [ Links ]
18. Ryman KD, Ledger TN, Weir RC, Schlesinger JJ, Barrett AD. Yellow fever virus envelope protein has two discrete type-specific neutralizing epitopes. J Gen Virol 1997; 78(Pt 6): 1353- 6. [ Links ]
19. Ballinger-Cabtree ME, Miller BR. Partial nucleotide sequence of South American yellow fever virus strain 1899/81: structural proteins and NS1. J Gen Virol 1990; 71: 2115-21. [ Links ]
20. Kawano H, Rostapshov V, Rosen L, Lai CJ. Genetic determinants of dengue type 4 virus neurovirulence for mice. J Virol 1993; 67(11): 6567-75. [ Links ]
21. Vorndam V, Mathews JH, Barrett AD, Roehrig JT, Trent DW. Molecular and biological characterization of a non- glycosylated isolate of St Louis encephalitis virus. J Gen Virol 1993; (Pt 12): 2653-60. [ Links ]
22. Nitayaphan S, Grant JA, Chang GJ, Trent DW. Nucleotide sequence of the virulent SA-14 strain of Japanese encephalitis virus and its attenuated vaccine derivative, SA-14-14-2. Virology 1990; 177(2): 541-52. [ Links ]
23. Dalgarno L, Trent DW, Strauss JH, Rice CM. Partial nucleotide sequence of the Murray Valley encephalitis virus. Comparison of the encoded polypeptides with yellow fever virus structural and non-structural proteins J Mol Biol 1986;187(3): 309-23. [ Links ]
24. Guirakhoo F, Heinz F, Kunz C. Epitope model of tick-borne encephalitis virus envelope glycoprotein E: analysis of structural properties, role of carbohydrate side chain, and conformational changes occurring at acidic pH. Virology 1989; 169: 90-9. [ Links ]
25. Hahn CS, Dalrymple JM, Strauss JH, Rice CM. Comparison of the virulent Asibi strain of yellow fever virus with the 17D vaccine strain derived from it. Proc Nat Acad Sci USA 1987; 84(7): 2019-23. [ Links ]
26. Van der Most RG, Corver J, Strauss JH. Mutagenesis of the RGD motif in the yellow fever virus 17D envelope protein. Virology 1999; 265(1): 83-95. [ Links ]
27. Rey FA, Heinz FX, Mandl C, Kunz C, Harrison SC. The envelope glycoprotein from tick-borne encephalitis virus at 2 A resolution. Nature 1995; 375(6529): 291-8. [ Links ]
28. Sanger F, Nicklen S, Coulsen AR. DNA sequencing with chaing terminating inhibitors. Proc Nat Acad Sci USA 1977;74: 5463-7. [ Links ]
Correspondencia: Carlos Yábar Varas. División de Biología Molecular, Instituto Nacional de Salud. Calle Cápac Yupanqui 1400, Lima 11 - Perú.
Apartado postal 471. Telf.: (0511)4719920 - Fax: (0511)4710179.
E-mail: cyabar@ins.gob.pe