Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.19 n.3 Lima jul./set 2002
TRABAJOS ORIGINALES
Caracterización de las mutaciones en el gen rpoβ asociadas a la rifampicina en pacientes con tuberculosis pulmonar*
Juan Agapito1,2; Víctor Neyra1; Juan Castro1; Roberto Accinelli3; Isaías Rodríguez2; José R. Espinoza1.
1 Unidad de Biotecnología Molecular (UBM), Laboratorios de Investigación y Desarrollo de Ciencia y Tecnología (LID), Universidad Peruana Cayetano Heredia. Lima - Perú.
2 Laboratorio de Mycobacterias, Hospital Nacional Cayetano Heredia. Lima – Perú.
3 Laboratorio de Respiración, Instituto de Investigaciones de Altura, Universidad Peruana Cayetano Heredia. Lima – Perú.
RESUMEN
Antecedentes: La resistencia a rifampicina en M. tuberculosis involucra mutaciones en el gen rpoβ que codifica a la subunidad β de la ARN polimerasa. Objetivo: Identificar las mutaciones del gen rpoβ , en cepas de M. tuberculosis asociadas con resistencia a rifampicina aisladas de la Subregión de Salud Lima Norte, Perú. Materiales y métodos: Se cultivó en Lowestein - Jenseen 73 muestras de esputo de pacientes con tuberculosis pulmonar. A 62, con más de 10 colonias por tubo, se les comprobó susceptibilidad a isoniazida, rifampicina, estreptomicina y etambutol. Se realizó la extracción de ADN por PCR, clonación en el vector pGEM-T, transformación, selección de clonas recombinantes y secuenciamiento del ADN plasmídico para la determinación de los polimorfismos del gen rpoβ. Resultados: 52 (83,9%) cepas fueron resistentes a rifampicina (Rif r) y 10 (16,1%) susceptibles (Rif s ). Se encontró alteraciones en el gen rpoβ en 51 de 52 cepas Rif r Se identificaron 20 mutaciones. Las mutaciones más frecuentes fueron encontradas en los codones Ser-531 (62,7%), His-526 (15,7%), Asp-516 (11,8%) y Gln-513 (5,9%). No se observó mutación alguna en las 10 cepas Rif s. 94,2% de nuestras cepas Rif r fueron también resistentes a isoniazida. Conclusiones: Se encontraron mutaciones en el gen rpoB de casi todas las cepas Rif r ; asimismo, casi todas las cepas Rif r fueron también resistentes a isoniazida.
Palabras clave: Mycobacterium tuberculosis / genética; Mycobacterium tuberculosis / efectos de drogas; Tuberculosis resistente a drogas; Rifampicina / uso diagnóstico; Perú (fuente: BIREME).
ABSTRACT
Background: Rifampicin resistance in M. tuberculosis involves mutations in the rpoβ gene, encoding the β subunit of RNA polymerase. Objective: To identify the rpoβ gene mutations in strains of M. tuberculosis isolated from the Health Sub-Region in North Lima, Perú. Materials and methods: 73 sputum samples from patients with pulmonary tuberculosis were cultured on Löwenstein-Jensen media. 62 samples, with more than 10 colonies per tube, were analyzed for susceptibility to isoniazid, streptomycin, rifampicin and ethambutol. DNA was isolated, amplified by PCR, followed by the cloning of the amplified product in the vector pGEM-T, transformation of competent E. coli bacteria, selection of recombinant clones and sequencing of the plasmid DNA for the determination of the polymorphisms in the rpoβ gene. Results: 52 strains out of 73 isolates (83,9%) were resistant to rifampicin (Rif r ), while 10 isolates (16,1%) were found to be rifampicin susceptible (Rif s ). 51 out of 52 Rif r strains contained mutations in the rpoβ gene associated with rifampicin resistance. 20 mutations were identified. The most frequently observed mutations were localized in the codons Ser-531 (62,7 %), His-526 (15,72%), Asp-516 (11,78%) and Gln-513 (5,88 %). No mutations were detected in the 10 Rif s strains. 94,2% of the Rif r strains were also resistant to isoniazid. Conclusions: rpoβ gene mutations associated with rifampicin resistance were present in almost all Rif r strains analyzed. Almost all Rif r strains were also resistant to isoniazid.
Key words: Mycobacterium tuberculosis / genetics; Mycobacterium tuberculosis / drug effects; Tuberculosis, drug resistant; Rifampin / diagnostic use; Peru (fuente: BIREME).
INTRODUCCIÓN
El reciente resurgimiento de la tuberculosis (TB) en países desarrollados se ha acompañado por un incremento en la prevalencia de la resistencia a drogas. De acuerdo con estimados de la Organización Mundial de la Salud (OMS), un tercio de la población mundial está infectada por Mycobacterium tuberculosis. Este importante reservorio de infectados está originando que se produzcan más de 8 millones de casos nuevos cada año en todo el mundo, sumados a los que no se curan y a los que recaen de años previos, hacen estimar una prevalencia actual de 16 millones de enfermos.
Actualmente, el Perú tiene uno de los mejores programas de control de tuberculosis del mundo, donde se ha logrado diagnosticar y tratar a los pacientes con esta enfermedad, obteniéndose para el año 2000 una tasa de morbilidad de 155,6 x 100 000 habitantes, tasa de incidencia de 133,6 x 100 000 habitantes y tasa de incidencia de TB pulmonar frotis positivo nuevos 88 x 100 000 habitantes1. Sin embargo, a pesar de los resultados logrados, se puede observar un incremento de la multidrogorresistencia, de 2,1% en 1995 a 3% en 1999 (p=0,005)2,3.
La aparición de epidemias de TB con cepas multirresistentes en los países desarrollados, sobre todo asociado con la epidemia VIH/SIDA4 es sin duda, uno de los principales riesgos para la salud pública en todo el mundo.
A partir de 1972, la rifampicina (Rif) ha sido utilizada como una droga para el tratamiento de cepas sensibles, así como también para aquellos pacientes que tienen resistencia por lo menos a isoniazida o estreptomicina. Su capacidad bactericida ha hecho posible su utilización en los tratamientos de corta duración para cepas sensibles; sin embargo, su uso es más prolongado en el tratamiento para los pacientes que tienen aislamientos resistentes a isoniazida5. La eficacia de este régimen de tratamiento está cambiando en regiones donde la resistencia inicial a rifampicina es alta, en las cuales la rápida detección de resistencia a rifampicina es una necesidad urgente. De particular importancia, la Rifr ha sido considerada como un marcador de cepas de tuberculosis multidrogoresistentes (TB-MDR), ya que ha sido asociado con brotes de enfermedad en hospitales, prisiones y otras instituciones en el ámbito mundial6.
La Rifr ha sido atribuida a mutaciones puntuales, inserciones y deleciones en una región limitada del gen rpoB que codifica a la subunidad ß de la ARN polimerasa7,8. Por lo menos 95% de los aislamientos para Rifr tienen mutaciones en este gen9,10. Estas mutaciones asociadas con resistencia fenotípica a rifampicina son generalmente localizadas en una región de 81 pb con mutaciones en los codones Ser-531, His-526 y Asp-516 7,10,11. Diferentes variaciones alélicas han sido detectadas en esta región y genotipos específicos del gen rpoB son conocidos por estar asociados con altos índices de Rif r en cepas de M. tuberculosis aisladas de diferentes partes del mundo7.
La aplicación de métodos genéticos para un examen rápido de susceptibilidad a rifampicina tiene importancia para la eficacia del tratamiento y control de TB - MDR. En el presente estudio evaluamos las mutaciones del gen rpoB de cepas resistentes (Rifr ) y susceptibles (Rifs) a rifampicina de M. tuberculosis aisladas de pacientes diagnosticados con TB. El objetivo general de este trabajo fue identificar las mutaciones del gen rpoβ asociadas con Rifr en cepas de M. Tuberculosis aisladas de la Subregión de Salud Lima Norte, Perú.
MATERIALES Y MÉTODOS
MUESTRAS
Las muestras clínicas utilizadas en el presente estudio fueron de pacientes diagnosticados con TB pulmonar provenientes del Hospital Nacional Cayetano Heredia y de los diferentes centros de salud del cono norte de Lima, Perú, durante el año 2001. Se incluyeron 73 muestras de esputo para el aislamiento de M. tuberculosis, de las cuales 62 muestras fueron sometidas a la prueba de susceptibilidad a drogas de primera línea (isoniazida, estreptomicina, rifampicina y etambutol). 11 cultivos no fueron incluidos por presentar aislamiento insuficiente (2 a 4 colonias).
PROCEDIMIENTOS
Baciloscopía y cultivos
Todas las muestras respiratorias fueron examinadas mediante la técnica de examen directo (Baciloscopía) y teñidas con la coloración de Ziehel - Nielsen. Los esputos fueron descontaminados con NaOH al 4% (w/v), de acuerdo al método de Petroff 12 y cultivadas por duplicado sobre el medio de Lowenstein Jensen e incubadas a 37°C. Los cultivos fueron examinados a las 48 y 72 horas de haber sido sembrados para verificar posibles contaminantes por flora secundaria. Revisiones posteriores se realizaron a los 30, 45 y 60 días hasta obtener cultivos positivos.
Susceptibilidad a drogas
La resistencia a rifampicina (Rif r ) fue determinada sobre el medio de Lowenstein - Jensen por el método de las proporciones. Para las cepas Rif r, la concentración mínima inhibitoria (MIC) fue de 40 µg/mL 12. Los aislamientos con crecimiento mayor del 1% en el medio conteniendo rifampicina comparado con un control sin antibiótico fueron considerados como resistentes. Además, a todos los aislamientos se les realizó susceptibilidad para isoniazida (INH), estreptomicina (SM) y etambutol (EMB) empleando el método de proporciones.
Extracción del ADN genómico de cultivos micobacterianos
Cultivos positivos crecidos sobre el medio de Lowestein - Jensen, Rif r y Rif s fueron aislados para su posterior extracción de ADN. La extracción de ADN se realizó de acuerdo a lo descrito por Van Soolingen13.
Diseño de cebadores de extensión (oligonucleótidos)
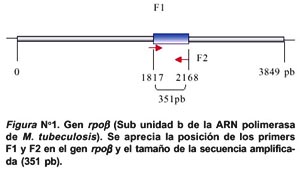
Los cebadores para el PCR fueron diseñados basándose en la secuencia del gen rpoB que codifica a la subunidad β de la ARN polimerasa (Figura N° 1) (Número de acceso a la base de datos en el Gen Bank N° U12205).
Los cebadores fueron diseñados con la ayuda de los programas OLIGO TM versión 4,0 y OLIGO calculator. Se comprobó la especificidad de los oligonucleótidos mediante el uso de los programas BLAST y FASTA (de acceso libre en internet), con el fin de encontrar posibles secuencias homólogas con otros organismos.
Reacción en cadena de la polimerasa (PCR)
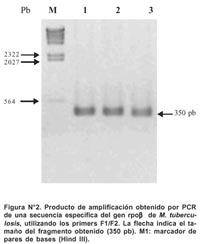
Los oligonucleótidos Forward F1 (5-CAGACGTTGATCA-ACATCCGC-3) y Reverse F2 (5- CAGGTACACGATCTCG-TCGCTAA- 3) fueron sintetizados por Applied Biosystems, Foster City California. Un producto de amplificación de 350 pb del gen rpoB fue obtenido por PCR utilizando los oligonucleótidos descritos anteriormente (Figura Nº 2). Para la reacción se utilizó 50 ng de ADN genómico de M. tuberculosis.
Clonación de los productos de amplificación obtenidos de PCR
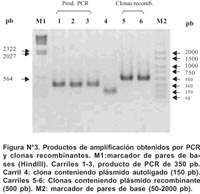
Los productos de amplificación (350 pb) del gen rpoB de cada una de las 62 cepas fueron clonadas en el vector pGEM R -T (PROMEGA).
Las clonas recombinantes (plásmido-inserto) fueron seleccionadas teniendo como agente selectivo a la ampicilina y al sustrato cromogénico IPTG. Las colonias seleccionadas se replicaron en medio LB/ampicilina y el inserto (350 pb del gen rpoB) fue verificado mediante una reacción de PCR, utilizando los primeros T7 (5´ -TAATACGACTCACTATAG- 3´) y SP6 (5´ -AGCTATTTAGGTGACACTATAG- 3´). Estos oligonucleótidos flanquean el sitio de clonación del vector pGEM R -T.
Secuenciamiento de clonas recombinantes
Para purificar los plásmidos se utilizó el Kit WizardR Plus Midipreps DNA purification system (Promega). El secuenciamiento de los plásmidos purificados fueron realizados en el secuenciador automático ALF express (Amershan Pharmacía Biotech) de la División de Biología Molecular del Instituto Nacional de Salud. La reacción de secuenciamiento se realizó con el kit Cys Autocycle TM (Amersham Pharmacia - Biotech), se utilizó 1 mg de ADN plásmidico en la reacción.
Los fragmentos de ADN de diferente tamaño formado por las reacciones, fueron separados por electroforesis en gel de poliacrilamida al 6% en condiciones denaturantes, 7M de urea, durante 12 horas a 1500 v, 60 mA y 25 w.
RESULTADOS
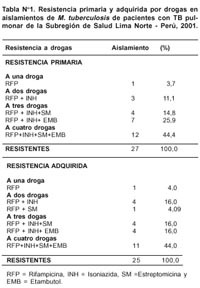
De las 62 cepas de M. tuberculosis utilizadas en este estudio, 52 (84%) fueron Rif r y 10 (16%) Rif s De las cepas Rif r 27 (52%) tuvieron resistencia primaria y 25 (48%) adquirida. La distribución de resistencia por drogas o combinación de drogas por tipo de resistencia se muestra en la Tabla Nº 1. La resistencia simultánea por lo menos a rifampicina e isoniazida (MDR) entre las cepas con resistencia primaria y adquirida fue 41,9% y 37,1% respectivamente.
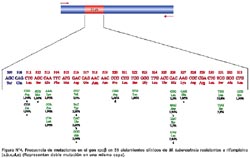
Todos los productos de amplificación obtenidos por PCR mostraron un mismo tamaño (350 pb) para la región del gen rpob en todas las cepas aisladas (Figuras Nº 2 y Nº 3). El secuenciamiento de los productos de amplificación de 62 cepas de M. tuberculosis (52 Rif r y 10 Rif s ) mostraron las siguientes mutaciones en una región corta de 81pb del gen rpoβ: 62,7% de las mutaciones ocurrió en el codón 531(58,8% cambió Ser-Leu y 3,9% de Ser-Trp). El 15,7% de las mutaciones ocurrió en el codón 526 (7,9% cambió His-Leu, 5,9% de His-Tyr y 1,9% cambió His-Asp). 11,8% de las mutaciones ocurrió en el codón 516 (5,9% cambió Asp-Val, 1,9% Asp-Tyr, 1,9% Asp-His y 1,9% cambió Asp-Ala). 5,9% de las mutaciones ocurrió en el codón 513(1,9% cambió Gln-Pro, 1,9% Gln-Lys y 1,9% cambió Gln-Leu). Para los codones 522, 527, 533, 520, 511 y 512 la frecuencia de mutación fue 1,9% respectivamente. (Figura Nº 4)7. De 52 cepas Rif r, 46 aislamientos tuvieron una mutación puntual en un solo codón y 5 presentaron doble mutación en codones separados: (511-Leu y 516-Asp), (516-Asp y 526-His), (516-Asp y 527- Lys), (520-Pro y 526-His) y (511-Leu y 512-Ser). La ausencia de mutación en una cepa resistente no fue observada. No se encontró mutación de 10 cepas Rif s en la secuencia del gen rpoβ analizada en este estudio (Tabla Nº2).
DISCUSIÓN
Éste es el primer trabajo realizado en el país, donde se han caracterizado las mutaciones que ocurren en un gen que confiere resistencia bacteriana en cepas de M. tuberculosis. Hemos podido determinar las mutaciones en el gen rpoβ de M. tuberculosis en las cepas aisladas de 62 pacientes con diagnóstico de TB pulmonar del cono norte de Lima (Figura N° 4).
Hallamos un total de 20 diferentes alteraciones genéticas. En 96,1% de los aislamientos resistentes, las variaciones encontradas estuvieron en los codones 531, 526, 513 y 516 y en 3,9% en los codones 522, 527, 533, 520, 511 y 512. Nuestros hallazgos coinciden con los reportes de estudios realizados en otros países, en donde se halló que más de 96% de cepas de M. tuberculosis RIF r tienen mutaciones en una región hipervariable del gen rpoβ 7, 14 y que 70% de esas mutaciones ocurre en los codones 531 y 526 7,10. Asimismo, estudios realizados por Spindola de Miranda15 en Brasil, han reportado mutaciones similares a las encontradas en este estudio.
Las mutaciones en los codones Ser-531 e His-526 dentro de la región hipervariable de 81 pb del gen rpoβ son las más frecuentes y su frecuencia relativa es muy similar en distintas regiones geográficas 10,15-17. En contraste, la frecuencia relativa de las mutaciones en los codones Asp-516, Gln-513 y Leu-533 varían según las regiones del mundo. En el estudio realizado en Estados Unidos de Norteamérica con cepas de M. tuberculosis de pacientes peruanos residentes en Lima y Cuzco se encontró, al igual que nosotros, que las mutaciones más frecuentes estuvieron en los codones Ser-531 y His-526, pero en las cepas de M. Tuberculosis Rif r la frecuencia de la mutación en His-526 fue la mitad 16. Además, encontramos que 6% de las cepas tuvieron mutaciones en el codón Gln-513, lo que no había sido reportado previamente en cepas de pacientes residentes en el Perú16.
En este estudio encontramos ausencia de mutación en el gen rpoβ en una cepa Rif r. Esto significa que mutaciones en otros genes son responsables de esta resistencia 10. Similarmente, Telenti en 1993 no identificó mutaciones en la región de 305 pb del gen rpoβ en 2 (3%) cepas de 66 aislamientos Rif r 10. A su vez, William en 1994, encontró que 7% de cepas M. tuberculosis Rif r tampoco mostraron mutación en una región de 305 pb del gen rpoβ11.
En 5 cepas RIF r se encontró una doble mutación en codones separados, como resultado de la sustitución de dos aminoácidos para cada cepa (Leu-511 y Asp-516, Asp-516 y His-526, Asp-516 y Lys-527, Pro-520 y His-526 y por último Leu-511 y Ser-512). Mutaciones puntuales en los codones Ser-531, His-526, Gly-513 o Asp516 han sido reportadas por tener altos niveles de resistencia en
E. coli (20 µg/mL) y M. tuberculosis18,19.
La descripción de las bases moleculares de resistencia a rifampicina y el informe de Telenti 10 sobre mutaciones en el gen rpoβ en M. tuberculosis ha servido para el desarrollo de nuevas y rápidas estrategias para la detección de esta resistencia, como la desarrollada en este estudio. La capacidad para determinar la sensibilidad a rifampicina y a otras drogas por métodos microbiológicos implica 4 a 6 semanas y hasta 3 meses, debido al crecimiento lento del bacilo de la tuberculosis, que se duplica cada 18-20 horas20.
Todas nuestras cepas aisladas Rif r, con excepción de 2 (3,9%) tenían resistencia a por lo menos un fármaco más. La MDR entre estas cepas fue de 98% (49/50). Entonces, esto nos hace pensar que el encontrar que una cepa de un paciente de Lima Norte tenga resistencia a rifampicina, indica con una probabilidad cercana a 100%, que lo sea también a isoniazida.
Nuestros resultados demuestran alta asociación entre las mutaciones en el gen rpoβ y el desarrollo de resistencia fenotípica de M. tuberculosis. Significa que podemos utilizar las determinaciones de las mutaciones en el gen rpoβ en cepas de M. tuberculosis para conocer cuáles de ellas son resistentes antes de obtener los resultados de las pruebas de sensibilidad por métodos bacteriológicos.
El secuenciamiento es aplicable para cualquier gen y es rápidamente adaptable para la identificación de mutaciones puntuales asociadas con resistencia a otras drogas antituberculosas, como isoniazida y estreptomicina.
Además, en los estudios de resistencia inicial realizados a nivel nacional reportados por el Instituto Nacional de Salud (INS)2,3 en 1999, dan cuenta de que la resistencia primaria global fue 17,8%, mayor a la registrada en 1995, posiblemente debido a inadecuados esquemas de tratamiento o a mutaciones espontáneas en cepas no expuestas previamente a drogas antituberculosas, tal es el caso de rifampicina (una mutación por cada 10-7 a 10-8 organismos)11. Asimismo se encontró un incremento de la MDR del 2,1% en 1995 al 3% en 19992,3. En la actualidad, la aplicación de técnicas moleculares permite establecer con precisión los mecanismos de resistencia a antibióticos.
Por esta razón, la necesidad de caracterizar cepas resistentes mediante el análisis de secuenciamiento nos ha permitido evidenciar las mutaciones que ocurren en el gen rpoB de M. tuberculosis de un grupo de pacientes con diagnóstico de TB inicial y adquirida del cono corte de Lima.
Asimismo, nuestros resultados confirman que el análisis genético del gen rpoβ basado en el secuenciamiento es aplicable universalmente en distintas regiones geográficas para detectar de manera rápida la resistencia a rifampicina.
AGRADECIMIENTOS
Este trabajo fue realizado en los Laboratorios de Investigación y Desarrollo de Ciencia y Tecnología (LID) Unidad de Biotecnología Molecular (UBM) de la Universidad Peruana Cayetano Heredia; en el Laboratorio de Mycobacterias del Hospital Nacional Cayetano Heredia y en la División de Biología Molecular del Instituto Nacional de Salud (INS).
REFERENCIAS
1. Ministerio de Salud. Informe 2000. Tuberculosis en el Perú. Lima: MINSA; 2001. [ Links ]
2. Ministerio de Salud. Vigilancia de la resistencia a los medicamentos antituberculosos en el Perú: 1995-1996. Lima: MINSA; 1997. [ Links ]
3. Ministerio de Salud. Vigilancia de la resistencia a los medicamentos antituberculosos en el Perú: 1999. Lima: MINSA; 2001. [ Links ]
4. Brudney K, Dobkin J. Resurgent tuberculosis in New York City: human inmunodeficiency virus, homelessness, and the decline of tuberculosis control programs. Am Rev Respir Dis 1991; 144: 745-9. [ Links ]
5. Mitchison DA. Mechanism of drug action in short-course chemotherapy. Bull Int Union Againt Tuberc 1985; 65: 30-7. [ Links ]
6. Bifani PJ, Plikaytis BB, Kapur V. Origin and interstate spread of a New York city multidrug-resistant: Mycobacterium tuberculosis clone family. JAMA 1996; 275: 452-7. [ Links ]
7. Kapur V, Li LL, Iordanescu S, Hamrick Mr, Wagner A, Kreiswith BN, et al. Characterization by automated DNA sequencing of mutations in the gene (rpoβ) encoding the RNA Polymerase B subunit in rifampin-resistant Mycobacterium tuberculosis strains from New York City and Texas. J Clin Microbiol 1994; (32)4: 1095-8.
8. Miller LP, Crawford JT, Shinnick TM. The rpoβ gene of Mycobacterium tuberculosis. Antimicrob Agents Chemother 1994; 38(4): 805-11. [ Links ]
9. Morris S, Bai GH, Suffys P, Portillo-Gómez L, Fair-chox M, Rouse D. Molecular mechanisms of multiple drug resistance in clinical isolates of Mycobacterium tuberculosis. J Infect Dis 1995; 171: 954-60. [ Links ]
10. Telenti A, Imboden P, Marchesi F, Lowrie D, Cole S, Colston JM, et al. Detection of rifampicin-resistance mutatio-ns in Mycobacterium tuberculosis. Lancet 1993; 34: 647-50. [ Links ]
11. Williams DL, Waguespack C, Eisenach K, Crawford JT, Portaels F, Salfinger M, et al. Characterization of rifampin resistance in pathogenic Mycobacteria. Antimicrob Agents Chemother 1994; 38(10): 2380-6. [ Links ]
12. Kantor I. Bacteriología de la Tuberculosis. Buenos Aires: INPAZ; 1988. [ Links ]
13. Van Soolingen D, Hermes PWM, De Hass PWE, Van Embden JDA. Genetic markers for the epidemiology of tuberculosis. Res Microbiol 1992; 143: 385-1. [ Links ]
14. Musser JM. Antimicrobial agent resistance in mycobacteria: molecular genetic insights. Clini Microbiol Rev 1995; 8: 496 514. [ Links ]
15. Spindola de Miranda S, Kritski AL, Fillol I, Habitat C, Panteix G, Drovet E. Mutations in the rpoB gene of rifampicin-resistant Mycobacterium tuberculosis strains isolated in Brazil and France. Mem Inst Oswaldo Cruz 2001;96(2): 247-50. [ Links ]
16. Escalante P, Ramaswamy S, Sanabria H, Soini H, Pan X, Valiente- Castillo O, et al. Genotypic characterization of drug resistant M. tuberculosis isolates in Peru. Tuberc Lung Dis 1998; 79: 111-8. [ Links ]
17. Van Rie A, Warren R, Mshanga I, Jordan AM, Vander Spug GD, Richardson M, et al. Analysis for a limited number of gene codons can predict drug resistance of M. tuberculosis in a high-incidence community. J Clin Microbiol 2001; 39(2): 636-41. [ Links ]
18. Jin DJ, Gross AC. Mapping and secuencing of mutations in the Escherichia coli rpoB gene that lead to rifampicin resistance. J Mol Biol 1988; 202: 45-58. [ Links ]
19. Steiner M, Chávez AD, Lyons HA, Steiner PH, Potugaleza C. Primary drug-resistant tuberculosis: report of and outbreak. N Engl J Med 1970; 283: 1353-8. [ Links ]
20. Vareldzis BP, Grosset I, Kantor J. Drug-resistant tuberculosis: laboratory issues.Tubercle Lung Dis 1994; 75: 1-7. [ Links ]
* Este estudio contó con el apoyo técnico – financiero del Proyecto Vigía. Enfrentando las amenazas de las enfermedades infecciosas emergentes y reemergentes. MINSA – USAID
Correspondencia: Juan Agapito. Unidad de Biotecnología Molecular (UBM),
Laboratorios de Investigación y Desarrollo de Ciencia y Tecnología (LID),
Universidad Peruana Cayetano Heredia.
Dirección: Av. Honorio Delgado 430 Urb. Ingeniería S.M.P. Lima-Perú.
Telf.: (51-1) 319-0000
E-mail: juancagapito@hotmail.com