Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.19 n.3 Lima jul./set 2002
TRABAJOS ORIGINALES
Variabilidad genética de Plasmodium falciparum en pacientes con malaria grave y malaria no complicada en Iquitos - Perú
Gisely Hijar G1; Higinio Quino A2; Carlos Padilla R1; Ysabel Montoya P1
1 División de Biología Molecular, Centro Nacional de Salud Pública, Instituto Nacional de Salud. Lima - Perú.
2 Dirección Regional de Salud Loreto. Loreto - Perú.
RESUMEN
Objetivo: Determinar la diversidad genética del gen que codifica la proteína rica en glutamato (GLURP) de Plasmodium falciparum en pacientes con malaria complicada y no complicada circulante en un área del departamento de Loreto, distrito de Maynas. Materiales y métodos: La diversidad genética fue analizada usando reacción en cadena de la polimerasa (PCR) en 30 muestras sanguíneas de pacientes con malaria no complicada (MNC) y 46 con malaria grave complicada (MGC). Resultados: Ocho genotipos fueron detectados en pacientes con MNC (Genotipo I,II,III, IV,V, VI,VII y VIII ) y cuatro genotipos en los pacientes con MGC (Genotipo V,VI,VII,VIII). Asimismo, en 50% de las muestras con MNC fueron detectadas infecciones múltiples, a diferencia de las muestras de MGC en donde no se detectó infecciones múltiples. Conclusión: Existe una diversidad genética en esta región del gen GLURP de P. falciparum, para esa época (marzo 1998 – abril 1999) y esa área del país. En tal sentido, nuestros resultados podrían servir de base para llevar a cabo estudios epidemiológicos posteriores, ya que permitiría conocer la distribución de las cepas circulantes en nuestro país.
Palabras clave: Malaria; Plasmodium falciparum / genética; Reacción en cadena de la polimerasa; Perú ( fuente: BIREME).
ABSTRACT
Objetive: Determine the genetic variation in the glutamate-rich protein (GLURP) encoding gene of Plasmodium falciparum in patients with severe and Non Complicated Malaria (NCM), Loreto (Maynas). Material y Methods: Genetic diversity was analyzed by using Polymerase Chain Reaction (PCR) in 30 blood samples from patients with Non Complicated Malaria (NCM) and 46 blood samples from patients with severe and complicated malaria (SCM). Results: Eight genotypes were detected in patients infected with NCM (I,II,III, IV,V, VI,VII y VIII genotypes) and four (V,VI,VII,VIII genotypes) in patients with SCM. Moreover, 50% of the samples of NCM patients multiple infections while no other infections were detected in SCM patients. Conclusion: A genetic diversity was found in this region of the GLURP gene of P. falciparum, during this period of time in this region of Peru. Our results could be useful as initial data for future epidemiologic studies, since they snow the distribution of (Plasmodium f. stroins) in our country.
Key words: Malaria; Plasmodium falciparum / genetics; Polymerase chain reaction; Peru (source: BIREME).
INTRODUCCIÓN
La malaria es causante de 1,5 a 2,7 millones de muertes anuales de seres humanos, y más de 2,3 billones de personas viven en áreas de riesgo de la enfermedad1. Su incidencia en el mundo está estimada en un rango de 300 a 500 millones de infecciones al año.
En el Perú, la malaria por Plasmodium falciparum se distribuye en las regiones de la costa norte de clima subtropical y en la selva de clima tropical con excesiva humedad y fuertes precipitaciones; estimándose que más de 8 millones de personas viven en áreas de alto y mediano riesgo de malaria.
La malaria por P. falciparum en el Perú, ha resurgido principalmente en la Cuenca Amazónica2,3 alcanzando sus niveles mas altos en 19964. Estudios clínicos diversos 5 y estudios de cohorte6 indican la aparición de cepas resistentes de P. falciparum a los medicamentos antimaláricos, tanto en la costa norte como en la Cuenca Amazónica.
Datos obtenidos de los años 1996 y 1997 en el Hospital de Apoyo de Iquitos, indican que los pacientes con malaria por P. falciparum presentaron complicaciones, ingresando a las diversas especialidades (544 en medicina, 261 en pediatría y 93 en obstetricia). Del mismo modo, otros informes revelaron que 35% de los casos manifestaron malaria complicada 5 y la tasa de letalidad hospitalaria de malaria por P. falciparum fue 6,68%7,8.
Plasmodium falciparum es el parásito más agresivo de las especies de Plasmodium que afectan al hombre. Este parásito presenta una alta diversidad genética que le permite evadir la respuesta inmunológica y resistir al tratamiento antimalárico. Inicialmente, las investigaciones realizadas a fin de conocer su diversidad genética, estuvieron basadas en el uso de métodos serológicos e isoenzimas; posteriormente en la detección de mutaciones y secuenciamiento de genes9-12. En la actualidad, numerosos marcadores moleculares han sido evaluados para medir la diversidad genética de P. falciparum, entre ellos resaltan genes de enzimas, antígenos de superficie y algunas otras proteínas12. Entre los marcadores moleculares están: el gen que codifica la proteína rica en glutamato de P. falciparum (GLURP)13 , el gen de las proteínas de superficie del merozoito 1 y 2 (MSP 1, MSP 2 )14-17; el gen de la proteína de superficie Pf33218; el gen de la proteína de superficie SPf6619, el gen de la proteína del circunsporozoito (CS)20 y el gen de la proteína anómala relacionada a la trombospondina (TRAP)21 .
Referente a la asociación existente entre la complejidad genética y la severidad de la enfermedad, no se ha encontrado un genotipo específico asociado, sin embargo, en estudios realizados en Senegal se halló una prevalencia del 60% del alelo MSP2 en pacientes con malaria severa22.
Asimismo, recientes estudios indican que un mismo individuo puede simultáneamente presentar múltiples infecciones con distintos genotipos de P. falciparum. La multiplicidad de las infecciones en un huésped puede estar relacionada a la endemicidad de la malaria22, al grado de inmunidad contra P. falciparum y al riesgo clínico de la malaria23,24.
En este estudio, se analizará el polimorfismo genético de la región R2 del gen GLURP de P. falciparum. En el genoma de P. falciparum, el gen GLURP está localizado en el cromosoma 10 en una sola copia, presentando un alto polimorfismo en muestras de otras partes del mundo14. Este gen se expresa en la etapa preeritrocítica y eritrocítica del ciclo de vida del parásito 25. Esta proteína contiene 1271 aminoácidos y está conformada por una región no repetitiva (R0) y dos bloques de secuencias repetitivas (R1 y R2, respectivamente).
El presente estudio se realizó con el objetivo de conocer la diversidad genética de P. falciparum, en pacientes con malaria complicada y no complicada circulante en un área del distrito de Maynas, departamento de Loreto, debido a la alta incidencia y a los casos de resistencia que se presentaron durante los años 1998 y 1999.
MATERIALES Y MÉTODOS
Estudio descriptivo analítico donde se evaluó 30 pacientes con malaria no complicada (MNC) y 46 con malaria grave complicada (MGC).
El lugar de estudio fue el departamento de Loreto, localizado al noreste del Perú cerca al río Amazonas. Los pacientes con MNC procedían de la provincia de Maynas, incluyendo los distritos de Punchana, Iquitos y urbanizaciones periféricas como: Moronacocha, Pampa Grande, Pampa Chica, Palo Seco, Río Itaya, Rumacocha, Río Momón, 9 de Octubre, Distrito de San Pablo Provincia de Mariscal Ramón Castilla y Río Corrientes en la Provincia de Loreto. En tanto que, los pacientes con MGC fueron captados del Hospital de Apoyo de Iquitos.
Se incluyeron febriles (Tº 37,5º C) con gota gruesa positiva para P. falciparum diagnosticados durante marzo de 1998 y abril de 1999 tomándose los criterios recomendados por la OMS en Ginebra para definir los casos MGC 26. Fueron excluidos del estudio los pacientes que presentaron infecciones mixtas de P. falciparum y P.vivax u otras especies de Plasmodium y las gestantes.
De acuerdo con la s características clínicas de los pacientes incluidos en este estudio, se clasificaron en dos grupos:
a. Malaria no complicada (MNC), por fiebre ¡Ý37,5 o C o con antecedentes de fiebre en las últimas 48 horas y parasitemia por encima de 587 a 274 970 parásitos por mL.
b. Malaria grave complicada (MGC), caracterizada por anemia severa con hemoglobina < 5 gm/dL e hiperparasitemia de 208 000 parásitos por mL a 1500 000 parásitos por mL.
MUESTRAS BIOLÓGICAS
El material biológico incluyó sangre total con anticoagulante EDTA (1,5mg/mL) y sangre total impregnada en papel de filtro sin anticoagulante. Dichas muestras sanguíneas fueron recolectadas antes del tratamiento con las drogas antimaláricas. Las muestras de sangre recolectadas en microtubos fueron guardadas a -20°C, mientras que las muestras en papel de filtro fueron conservadas a temperatura ambiente hasta su posterior análisis.
EXTRACCIÓN DE ADN
Extracción de ADN de sangre total
La extracción de ADN se realizó de 200 uL de sangre total con anticoagulante EDTA empleando el kit de purificación de ADN QiAmp Blood (QiaGen TM ). Previamente, los parásitos fueron lisados y el material genético juntamente con las proteínas y otros detritus celulares se liberaron. Las muestras se filtraron a través de columnas con afinidad al ADN. Las columnas retuvieron el ADN de las muestras, el cual fue purificado mediante lavados con el buffer del Kit, seguidamente el ADN fue eluido de las columnas en agua bidestilada libre de ADNasas y finalmente, el ADN fue cuantificado por espectrofotometría y almacenado a 4°C hasta su uso.
Extracción de ADN impregnado en papel filtro
Para purificar el ADN de las muestras sanguíneas impregnadas en papel filtro, se cortó pedazos del papel de 2 a 4 mm de diámetro, luego fueron lavados con la solución de purificación ( FTA Purification Reagent). Posteriormente, el papel se lavó dos veces con 500 uL de PBS (solución bufferada fosfato). El sobrenadante fue retirado y el papel filtro secado a 60°C durante 30 minutos; una vez seco, el papel filtro fue colocado en el tubo de reacción para PCR.
Reacción en cadena de polimerasa
El gen que codifica una proteína rica en glutamato de P. falciparum (GLURP) fue seleccionado como marcador polimórfico genético; para ello, este gen fue amplificado de las muestras usando la técnica de reacción en cadena de la polimerasa (PCR)27, 28 . Asimismo, los oligonucleótidos (GLURP1 y GLURP2) utilizados para esta prueba fueron los reportados por Ross Coppel en la base de datos de secuencia de malaria UNDP/World Bank/WHO-TDR.
Las condiciones de PCR se estandarizaron utilizando ADN de la cepa referencial africana de P. falciparum 3D7. La temperatura óptima de anillamiento fue de 52°C y las concentraciones óptimas de los reactivos fueron 2,5 Mm de MgCl 2 , 200 mM de cada nucleótidos (mezcla de dATP, dCTP, dGTP y dTTP), 1 mM de cada oligonucleótido (GLURP1 y GLURP2) y 0,5 U de la enzima termoestable ADN polimerasa por cada tubo. El ciclaje de amplificación incluyó una denaturación inicial de 95°C durante 10 minutos, luego 30 ciclos de una denaturación de 95°C durante 30 segundos, hibridación de los oligonucleótido a 52°C durante un minuto y la extensión de los iniciadores a 72°C durante 90 segundos; luego una extensión final de 72°C durante 10 minutos. Cada reacción de amplificación fue realizada con su respectivo control positivo ADN de la cepa referencial P. falciparum 3D7, controles negativos y control del sistema. Posteriormente, 5mL del producto de amplificación fueron resueltos mediante electroforesis en geles de agarosa a una concentración al 1,5 %, estos geles fueron teñidos con bromuro de etidio y visualizados a través de un transiluminador de UV modelo TM-20 chromato–vue (UVP Inc.) a la longitud de onda de 365 nm.
Los geles fueron fotografiados empleando una cámara para luz ultravioleta Polaroid MP4+.
RESULTADOS
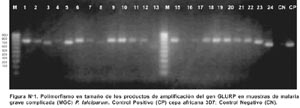
Los productos de amplificación exhibieron los tamaños esperados, obtuviéndose dimensiones entre los rangos de 450 pb y 800 pb, mientras que la cepa 3D7 presentó un producto de amplificación de 900 pb.
DISTRIBUCIÓN ALÉLICA Y POLIMORFISMO EN TAMAÑO
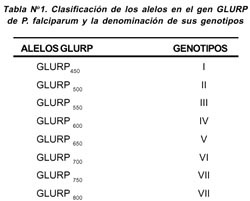
En las 46 muestras incluidas se determinaron ocho diferentes alelos, correspondiendo a tamaños de productos de PCR que migraban entre 450 pb a 800 pb. 80,2% de las muestras analizadas presentaron un sólo alelo. Los alelos más frecuentes fueron el alelo GLURP 800 (genotipo VIII, correspondiente a un tamaño de 800 pb) y el alelo GLURP 450 (genotipo I, correspondiente a un tamaño de 450 pb) de acuerdo al tamaño de las bandas exhibidas, como genotipo I y genotipo VIII respectivamente (Tabla Nº 1 y Figura Nº 1).
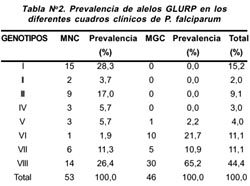
Se encontró también que 19,8% de las muestras correspondieron a infecciones múltiples (más de un alelo de P. Falciparum presente en la muestra), lo cual puede significar que un paciente se encontraba infectado con más de un genotipo de P. falciparum al mismo tiempo. (Tabla Nº 2.)
PREVALENCIA DE LOS GENOTIPOS EN LOS DIFERENTES CUADROS CLÍNICOS
Se observó polimorfismo genético en tamaño del gen GLURP de P. falciparum en pacientes con cuadros clínicos de MNC, obteniéndose fragmentos en el rango de 450 pb a 800 pb. El genotipo I y el genotipo VIII fueron los más prevalentes, encontrándose (15/30) y (14/30) respectivamente (Tabla Nº 2). Asimismo, es importante mencionar que en este grupo de muestras, los pacientes presentaron infecciones múltiples de P. falciparum, encontrándose hasta cuatro genotipos de parásitos en un mismo paciente.
En los cuadros clínicos de MGC, se obtuvieron fragmentos desde 650 pb a 800 pb. (Figura Nº 1). El genotipo VIII de P. falciparum fue el más prevalente (30/46). El genotipo V (1/46) fue encontrado sólo en un paciente. Es importante mencionar que no se detectó infecciones múltiples en los pacientes con MGC.
DISCUSIÓN
Los dos métodos de extracción de ADN, a partir de sangre impregnada en papel de filtro y de sangre periférica, brindaron resultados satisfactorios. El ADN extraído a partir de sangre periférica permitió una recuperación de ADN en un rango de 500 ng a 1 µg, en contraste al método de extracción de ADN a partir de papel de filtro, en el cual no fue posible medir la concentración de ADN en la muestra. Por otro lado, el uso de papel de filtro para la colecta de muestras sanguíneas presenta la ventaja de no requerir transporte especial, haciendo atractivo este método para estudios de campo.
La diversidad genética encontrada en el gen GLURP de P. falciparum en esta área del Perú es similar a la encontrada en otras áreas del mundo. En nuestro caso, esta biodiversidad observada podría estar asociada al comportamiento epidemiológico de la malaria en Loreto como son los cambios climáticos, incremento de la transmisión y el grado de la inmunidad del paciente contra el P. falciparum23.
Asimismo, si analizamos las posibles causas de este polimorfismo podemos mencionar la alta recombinación genética de P. falciparum que ocurre durante la etapa sexual del ciclo de vida del parásito dentro del mosquito Anopheles29.
Nuestros resultados concuerdan con reportes en los cuales se afirma que una alta biodiversidad genética se asocia con áreas de alta transmisión de malaria, sucediendo lo contrario en áreas de baja transmisión de la enfermedad30.
Encontramos también que los genotipos I y III se encuentran siempre acompañados de otros genotipos, de esto se podría sugerir que estos genotipos fueron los antecesores. Si ese fuese el caso, los genotipos II, IV, V, y VII quizás sean los genotipos que se generaron recientemente. Lo mismo se podría sugerir del genotipo VIII el cual por ser el más abundante también podría ser uno de los antecesores de los demás genotipos.
En los dos grupos de cuadros clínicos de malaria no se encontró una asociación directa de un genotipo en especial para ninguno de los cuadros clínicos; sin embargo, es importante mencionar que existe el doble de prevalencia del genotipo VIII en la MGC con respecto a la MNC; además, el genotipo VI se encontró once veces más en la MGC. Asimismo, los genotipos I, II, III y IV se presentaron exclusivamente en los casos MNC, esto podría indicar una posible relación del genotipo VI y VIII con la MGC y de los genotipos I, II, III y IV con MNC.
La prevalencia de los genotipos de P. falciparum encontrados en este estudio son similares a los encontrados en los estudios de Senegal, donde un mayor polimorfismo genético de P. falciparum fue también observado en MNC en relación con MGC21.
Nuestros resultados nos permitirán continuar estudiando la distribución de las cepas resistentes a las drogas antimaláricas en la selva amazónica y en la costa norte de nuestro país. Asimismo, pueden también ser utilizados para investigar la proporción de casos de recrudescencia o reinfección de esta enfermedad.
Adicionalmente, sería importante realizar estudios de la asociación del genotipo humano con los diferentes cuadros clínicos de la malaria, lo cual podría evaluarse estudiando la variabilidad genética en los genes del antígeno leucocitario Humano (HLA), así como también la capacidad de generar interleuquinas y otros receptores humanos involucrados en la citoadherencia del eritrocito infectado.
AGRADECIMIENTOS
Agradecemos al programa de pequeños subsidios de investigación de la Organización Panamericana de la Salud (OPS) por financiar parte de este proyecto, a la Dirección de Salud de Iquitos y al personal del Hospital de Apoyo de Iquitos, quienes ayudaron a llevar acabo este estudio, al Dr. Witzig Richard y al grupo de malaria del Instituto Nacional de Salud: Blg. Nancy Arróspide, Blg. Sonia Gutiérrez y Dr. Wilmer Marquiño.
REFERENCIAS
1. World Health Organization. Weekly Epidemiology. Rec 1997; 72; 269-92. [ Links ]
2. Ministerio de Salud. Malaria en el Perú. Reporte Epidemiológico 1998; Semana Nº 52. Lima: OGE; 1998. [ Links ]
3. Dirección de Salud de Loreto. Boletín de Programa de Control de Malaria 1998. Loreto; MINSA, p. 58-9;1998. [ Links ]
4. Chauca H, Quintana J. Evaluación in vivo de la respuesta de Plamodium falciparum a la Cloroquina en el foco de la carretera Yurimaguas-Tarapoto. Rev Per Epidemiol 1992; 6(2): 15-20. [ Links ]
5. Ministerio de Salud. Evaluación de la nueva estrategia para el control de la Malaria y de la cohorte de tratamiento antimalárico en regiones y subregiones de salud situadas en áreas de alto riesgo de transmisión de malaria. Lima: MINSA; 1995. Informe anual 1994. [ Links ]
6. Gilles HM. Tratamiento del paludismo grave y complicado: guía práctica. Ginebra: Organización Mundial de Salud; 1993: p. 1-56. [ Links ]
7. Barker R, Banchongaksorn T, Courval J, Suwonkerd W, Rimwungtragoon K, Wirth D. A simple method to detect Plasmodium falciparum directly from blood samples using the polymerase chain reaction. Am J Trop Med Hyg 1992; 46(4): 416-26. [ Links ]
8. Viriyakosol S, Siripon N, Petcharapirat C, Petcharapirat P, Jarra W, Thaithong S, et al. Genotyping of Plasmodium falciparum isolates by the polymerase chain reaction and potential uses in epidemiological studies. Bull WHO 1995; 73(1): 85-95. [ Links ]
9. McBride J. S, Walliker D, Morgan G. Antigenic diversity in the human malaria parasite Plasmodium falciparum. Science. 1982; 217: 254–7. [ Links ]
10. Carter R, Voller A. The distribution of enzyme variation in populations of Plasmodium falciparum in Africa. Trans R Soc Trop Med Hyg 1975; 69: 371-6. [ Links ]
11. Kemp DJ, Cowman AF, Walliker D. Genetic diversity in Plasmodium falciparum. Advances in Parasitology 1990; 29: 75-149. [ Links ]
12. Konate L, Zwtyenga J, Rogier C, Bishchoff E, Fontenille D, Tall A. The epidemiology of multiple P. falciparum infec-tions. Trans R Soc Trop Med Hyg 1999; 93(Supp 1): 21-8. [ Links ]
13. Al-Yaman F, Genton B, Reeder JC, Anders RF, Smith T, Alpers MP. Reduced risk of clinical malaria in children infected with multiple clones of Plasmodium falciparum in a highly endemic area: a prospective community study. Trans R Soc Trop Med Hyg 1997; 91: 602-5. [ Links ]
14. Beck H P, Felger I, Huber W, Steiger S, Smith T, Weiss N, et al. Analysis of multiple Plasmodium falciparum infections in Tanzanian children during the phase III trial of the malaria vaccine SPf66. J Infect Dis 1997; 175: 921-6. [ Links ]
15. Quino H, Durand S, Maguiña C. Malaria por P falciparum en Adultos hospitalizados en Iquitos: aspectos clínicos epidemiológicos Acta Médica 2001; 18: 52-7. [ Links ]
16. Snounou G, Bourne T, Jarra W, Viriyakosol S, Wood JC, Brown KN. Assessment of parasite population dynamics in mixed infections of rodent plasmodia. Parasitology 1992; 105: 363-74. [ Links ]
17. Robson KJ, Hall JR, Davies LC, Cristiani A, Hill AV, Wellens TE. Polymorphism of the TRAP gene of Plasmodium falciparum. Proc R Soc Lond Series B:Biological Sciences 1990; 242: 205-16. [ Links ]
18. Quino A, Durand S, Vilca O. Malaria grave y complicada por P. falciparum: estudio clínico descriptivo de 242 casos Bol SPEIT 1997; 6(2): 58. [ Links ]
19. Mercereau-Puijalon O, Fandeur T, Bonnefoy S, Jacquemot C, Sarthou JL. A study of the genomic diversity of Plasmodium falciparum in Senegal. 2. Typing by use of the polymerase chain reaction. Acta Tropica 1991; 49: 293-304. [ Links ]
20. Kain K, Lanar D. Determination of genetic variation within Plasmodium sp. falciparum by enzymatically amplified DNAfrom filter paper disks impregnated with whole blood. J Clin Microbiol 1991; 29(6): 1171-4. [ Links ]
21. Babiker H, Satti G, Walliker D. Genetic changes in the population of Plasmodium falciparum in a Sudanese village over a three-year period. Am J Trop Med Hyg 1995; 53: 7-15. [ Links ]
22. Haddad D, Snounou G, Mattei D, Enamorado I. G, Figueroa J, Stahl S. & Berzins K. Limited genetic diversity of Plasmodium falciparum in field isolates from Honduras. American Journal of Tropical Medicine and Hygiene.1999; 60, 30-4. [ Links ]
23. Su X, Ferdig MT, Huang Y, Huynh CQ, You J, Wootton JC, Wellems TE. A genetic map and recombination parameters of Human malaria parasite Plasmodium falciparum. Science 1999; 286:1351-3. [ Links ]
24. Borre MB, Dziegiel M, Hogh B, Pettersen E, Rieneck K, Riley E, et al. Primary structure and localization of a conserved immunogenic Plasmodium falciparum glutamate rich protein (GLURP) expressed in both the preerythrocytic and erythrocytic stages of the vertebrate life cycle. Mol Biochem Parasitol. 1991; 49: 119-31. [ Links ]
25. Marshall VM, Anthony RL, Bangs MJ, Purnomo RF, Coppel RL. Allelic variants of the Plasmodium falciparum merozoite surface antigen 2 (MSA-2) in a geographically restricted area of Irain Jaya. Mol Biochem Parasitol. 1994; 63:13-21. [ Links ]
26. Robert F, Ntoumi F, Angel G, Candito D, Rogier C, Fandeur T, et al. Extensive genetic diversity of Plasmodium isolates collected from patients with severe malaria in Dakar, Senegal. Trans R Soc Trop Med Hyg 1996; 90(6): 704-11. [ Links ]
27. Aramburú GJ, Ramal AC, Witzig R. Malaria reemergence in the peruvian amazon region. Emerg Infect Dis 1999; 5(2): 209-15. [ Links ]
28. Reeder JC, Marshall VM. A simple method for typing Plasmodium falciparum merozoito surface antigens 1 and 2 (MSA-1 and MSA-2) using a dimorphic-form specific polymerase chain reaction. Mol Biochemi Parasitol 1994; 68: 329-32. [ Links ]
29. Creasey A, Fenton B, Walker A, Thaithong S, Oliveira S, Mutambu S, et al. Genetic diversity of Plasmodium falciparum shows geographical variation Am Trop Med Hyg 1990; 42: 403-13. [ Links ]
30. Contamin H, Fandeur T, Bonnefoy S, Skouri F, Ntoumi F, Mercereau-Puijalon O. PCR typing of field isolates of Plasmodium falciparum. J Clin Microbiol1995; (4): 944-51. [ Links ]
Correspondencia: Gisely Hijar Guerra. División de Biología Molecular, Instituto Nacional de Salud.
Dirección: Calle Cápac Yupanqui 1400, Lima 11-Perú
Telf.: (51-1)471-9920 Fax: (51-1)471-0179
E-mail: ghijar@ins.gob.pe