Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.19 no.4 Lima Oct./dic. 2002
TRABAJOS ORIGINALES
Detección Molecular de Toxinas Termoestable y Termolabil de Escherichia coli mediante Hibridación
Isabel Arias B1; José Carlos Huguet T1
1 División de Bacteriología, Laboratorio de Enteropatógenos, Instituto Nacional de Salud. Lima, Perú.
RESUMEN
Objetivos: Identificar mediante el método de hibridización por colony blot las toxinas de Escherichia coli enterotoxigénica y relacionar los resultados con los serotipos encontrados. Materiales y métodos: se evaluaron todas las cepas de E. Coli recolectadas en el Hospital de Emergencias Pediátricas de Lima durante los meses de diciembre de 1998 - abril 1999. Se usaron dos sendas de ADN que identificaban el gen de la toxina termolábil (LT) y el de la toxina termoestable (ST) Para la detección de los serotipos se usaron 22 antisueros de diferentes categorías de E. coli. Resultados: se encontraron 233 cepas de E. coli, 27,9% de E. coli poseían el gen LT, 3,0% el gen ST y 1,3% tenían ambos. Conclusiones: los serotipos y la presencia de los genes LT y ST no necesariamente tienen relación, demostrándose que la identificación serológica es importante en el estudio epidemiológico de diarreas causadas por E. coli debiéndose confirmar la identificación de las categorías patogénicas mediante la detección de factores de virulencia.
Palabras clave: Escherichia coli/clasificación, Hibridación genética, Toxinas bacterianas, Factores de virulencia (fuente:BIREME).
SUMMARY
Objectives: To identify enterotoxigenic Escherichia coli heat labile and heat stable toxins using the colony blot hibridization technique and match the results with the serotypes found. Materials and Methods: All Escherichia coli strains collected at the Hospital de Emergencias Pediátricas between December 1998 and April 1999 were assessed. Two specific DNA probes that identified the Heat-labil toxin gene (LT) and the Heat-stable toxin gene (ST) were used. 22 types of E. colis antisera were used for detecting the serotypes. Results: 233 E. coli strains were found; 27.9% carried the LT gene, 3,0% the ST gene, while 1,3% of the strains had both genes. Conclusions: The presence of serotypes and of LT and ST genes is not necessarily related, thus demonstrating that serology identification is important in the epidemiological studies of diarrheas caused by E. coli, and the identification of pathogenic categories must be confirmed by the detection of virulence factors.
Key words: Escherichia coli/clasification; Hibridization; Genetic; Bacterial Toxins; Virulence factors. (source: BIREME)
INTRODUCCIÓN
Escherichia coli es uno de los agentes bacterianos que produce diarrea con mayor frecuencia en el Perú y el mundo1-6. Se conocen hasta la fecha seis diferentes categorías que se manifiestan con diferentes cuadros clínicos. Dentro de estas categorías patogénicas, sobresale la E. Coli enterotoxigénica, llamada también ECET o ETEC1. Este serotipo de E. coli es importante en países en desarrollo, pues junto con la E. coli enteropatógena (ECEP) causan la mayoría de los casos de diarreas agudas en la población infantil1-9. La virulencia de ECET está asociada a la presencia principalmente de dos factores patogénicos: la toxina termolábil y la toxina termoestable. La toxina termolábil es una toxina muy parecida a la toxina colérica que provoca un cuadro característico de diarrea acuosa y está compuesta por una subunidad catalítica denominada A y cinco subunidades llamadas B cuya función es la de ligarse a receptores celulares del enterocito. En el caso de la toxina termoestable, ésta es un pequeño péptido que induce la secreción actuando sobre la guanilato ciclasa del enterocito1,10.
En la actualidad, para la detección de ECET se utilizan diversas metodologías. Una de las más conocidas es el ensayo en animales, mediante la inoculación intragástrica en ratones lactantes y la observación in vivo de la acción de las toxinas de ECET10, otro método utilizado es la identificación mediante enzima inmunoensayo (ELISA)11
Una metodología que puede servir para la identificación presuntiva de la categoría ECET es la serotipificación, aprovechando la relación de ciertos serotipos con esta categoría patogénica; sin embargo, esto no es definitivo1,12.
En la actualidad, se considera como pruebas muchos más específicas al cultivo celular y a la detección molecular de los genes LT y ST por hibridación o por reacción en cadena de la polimerasa (PCR)13-15.
Teniendo en cuenta que la categoría patogénica de ECET se caracteriza y se define como aquel grupo de E. coli que posee la toxina termolábil, la termoestable o la combinación de ambas, el presente estudio tuvo la finalidad de evaluar la hibridización por colony blot para la detección de los genes que codifican estas toxinas, así como también relacionar los factores de las toxinas con los serotipos reportados en este estudio.
MATERIALES Y MÉTODOS
Estudio descriptivo analítico, en el que se evaluaron cepas de E. coli, procedentes del Hospital Emergencias Pediátricas (Lima, Perú), durante los meses de diciembre 1998 y abril de 1999. Las muestras obtenidas para el estudio fueron a partir de casos de diarrea que ingresaron a los consultorios y salas de hospitalización. El laboratorio del hospital diagnosticó E. coli en 233 muestras, siendo luego éstas confirmadas mediante pruebas bioquímicas y serológicas en el Instituto Nacional de Salud (Lima – Perú)
Las cepas fueron evaluadas con antisueros para la detección de serotipos: O26, O55, O111, O119, O114, O125, O142, O158, O86, O126, O127, y O128, O28ac, O29, O139, O114, O152, O112ac, O124, O143, O164 y O167. Se utilizaron cepas positivas de E. Coli diarreogénicas referenciales y otras enterobacterias relacionadas como controles negativos.
SONDAS UTILIZADAS
Para la detección del gen de la toxina termolábil (LT) de E. coli enterotoxigénica se utilizó el producto de una reacción en cadena de la polimerasa (PCR) obtenido con los iniciadores LT1: 5 tct cta tat gca cag gga gc 3 y LT2: 5 cca tac tga ttg ccg caa t 316. El PCR se realizó con los siguientes reactivos y concentraciones: Buffer de reacción 1X, Cl2 Mg 2,5 mM; iniciadores 10 pmoles de cada uno; mezcla de nucleótidos 100 mM; enzima amplitaq polimerasa 1 U y ADN 10 ng. Todo diluido en un volumen final de 25 mL. Cada tubo de reacción fue sometido a un ciclaje de temperaturas: 95ºC por 5 minutos, seguidos de 30 ciclos de 95ºC por 1 minuto; 52ºC por 1 minuto y 72ºC por 1.5 minutos. Seguidamente, una etapa de extensión final de 72ºC por 10 minutos. El producto de PCR fue marcado en forma directa con un conjugado de peroxidasa mediante el sistema ECL de Amershan Pharmacia.
Para la detección del gen de la toxina ST de E. Coli enterotoxigénica se empleó un oligonucleótido con la secuencia 5gat tac aac aaa gtt cac agc agt 317. A diferencia de la sonda para LT, este oligonucleótido fue marcado en el extremo 3´ mediante el sistema de UTP fluoresceina ECL de Amershan Pharmacia, siguiendo las especificaciones técnicas del proveedor.
PREPARACIÓN DE LAS MEMBRANAS DE NYLON
Las cepas de E. coli fueron reactivadas en caldo Luria, incubándolas toda la noche a 37ºC. Seguidamente, volúmenes de 5 mL de la suspensión bacteriana fueron sembrados en una membrana nylon sobre una placa petri con agar TSA, incubando toda la noche a 37ºC. Pasado el tiempo de incubación, las membranas fueron tratadas de la siguiente manera: 5 minutos en SDS al 10%, 10 minutos en solución desnaturalizante (NaCl 1,5 M + NaOH 0,5 M) 5 minutos en solución neutralizante (NaCl 1,5 M + Tris 0,5 M) y 5 minutos en solución salina citrada SC 2X (pH 7,0) Finalmente, la membrana fue secada a temperatura ambiente e irradiada con luz UV por 5 minutos.
HIBRIDACIÓN
La hibridación fue realizada utilizando el sistema de quimioluminiscencia ECL de Amershan Pharmacia. En el caso de la sonda marcada con el UTP fluoresceína, se utilizó el sistema ECL. El proceso de incubación de cada sonda duró 3 horas usando un buffer de lavado de alta astringencia con la finalidad de obtener una alta especificidad. Finalmente, la hibridación de cada sonda fue detectada por quimioluminiscencia utilizando un film autoradiográfico.
RESULTADOS
Se distinguió un porcentaje importante de cepas de E. coli con el gen codificante de la toxina termolábil LT 27,9 % (65 cepas), mientras que el gen de la toxina termoestable ST fue detectado en 3,0 % (7 cepas) Sólo 1,3 % (3 cepas) mostró la presencia de los dos genes codificantes de las toxinas LT y ST. 68 % (158) de las cepas no mostraron toxinas.
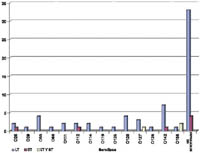
Los serotipos que portaron el gen de la toxina termolábil fueron O55, O111, O126, O127, O26, O142, O128, O114, O129, O112 y O86, mientras un grupo importante de E. Coli (45 cepas) no tipificadas serológicamente con los antisueros utilizados mostraron también el gen de LT. Los serotipos con el gen de la toxina termoestable (ST) fueron: O112, O142, O26. En este caso, se encontraron 2 cepas de E. coli con el gen ST que no pudieron ser tipificadas por serología. En tanto que los serotipos que mostraron el gen tanto de la toxina termoestable y termolábil fueron los siguientes: O158 y O127 (Figura N.º1)
En cuanto a las condiciones técnicas de la prueba, se encontró alguna dificultad al evaluar los filmes autoradiográficos hibridados con la sonda correspondiente al gen de la toxina termolábil LT, debido a que tanto los controles como las cepas problemas presentaron un importante background o señal de fondo, no pudiéndose en algunos casos lograr una buena discriminación entre estos dos grupos. Este problema fue solucionado al tomar como referencia los controles negativos con background y hacer una línea de corte para poder identificar las cepas verdaderas positivas (Figura N.º2) En el caso de los filmes hibridados con la sonda correspondiente al oligonucleótido marcado con fluoresceína, no se observó background importante pudiéndose discriminar con claridad las cepas positivas de las negativas (Figura N.º3)
Figura N° 1. Distribución de toxinas LT o ST en diferentes
serotipos de E. coli.
Figura N° 2. Film autoradiográfico - Colony blot. Detección
de gen LT (toxina termolábil). Sonda: producto de amplificación
por PCR. (1): cepa control negativo a LT, y (2): cepa control
positivo a LT.
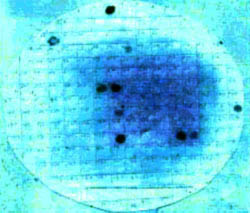
Figura N° 3. Film autoradiográfico - Colony blot. Detección
de gen ST (toxina termoestable). Sonda: oligonucleótido
marcado extremo 3, (1): cepa control negativo a ST, (2): cepa
control positivo sólo a LT, y (3): cepa control Positivo a ST.

DISCUSIÓN
En el presente estudio, se encontró un porcentaje importante de cepas que presentaron la toxina termoestable, termolábil, o ambas, caracterizadas mediante hibridación. Esta identificación de los factores de virulencia es un resultado mucho más fiel para categorizar patogénicamente a una Escherichia coli.
Nuestros resultados muestran un porcentaje mayor de cepas con toxina termolábil que termoestable o cepas con ambas toxinas. Trabajos anteriores muestran diversos resultados indicando en algunos casos frecuencias muy similares de las dos toxinas12 o un mayor porcentaje de E. coli productoras de toxina termolábil18,19. Otros autores indican un distinto perfil epidémico para la diarrea causada por cepas de E. coli LT y ST9.
Se debe mencionar que en muchos de los casos la relación serológica con el factor de virulencia y la categoría patogénica respectiva a los serotipos no concuerdan con la presencia tanto de LT o ST. Es importante indicar, por ejemplo, que serotipos asociados normalmente a la categoría enteropatógena (ECEP) como O111, O119, O126, O127, O142, O125 y O128 como lo describe Nataro1 y los serotipos O112 y O86 asociados a E. Coli enteroinvasiva (EIEC) presentaron en algunos casos el gen LT, ST o ambos. Numerosos autores han reportado la presencia de estos genes en los serotipos indicados anteriormente20-31. En consecuencia, si bien es cierto las pruebas de serotipia son importantes para la identificación de categorías patogénicas, los resultados de estas pruebas serológicas deben ser relacionadas a la clínica y a los factores de virulencia para tener un diagnóstico concluyente. De esta manera, se demuestra que el serotipo no es más una prueba definitiva en la identificación de la categoría patogénica de E. coli.
En nuestro estudio, también se encuentran otros serotipos de los que no se tenía información sobre la presencia de toxinas LT o ST. Tal es el caso del serotipo O55. Sin embargo, cabe mencionar que algunos investigadores reportan un gen muy parecido al LT en el serotipo O55, al cual llaman LT, indicando además una relación antigénica muy fuerte al realizar trabajos de neutralización. Este dato puede ayudar a explicar la presencia de señal de hibridación en cepas de serotipo O55, postulándose el posible apareamiento inespecífico de la sonda LT con el gen LT´ de estas cepas32.
Un importante grupo de cepas que no reaccionaron con los antisueros probados fue positivo a LT, ST o ambos. En este caso, es de suponer que las cepas pertenecen a otros serotipos no evaluados en este estudio, los cuales pueden estar más relacionados a la categoría de E. Coli enterotoxigénica1, 22.
En cuanto al poder de discriminación de la técnica utilizada, se pudo establecer una línea de corte, a pesar de señales de fondo en la hibridación, la cual sirvió para reconocer las cepas positivas tanto a LT como a ST. Es importante mencionar que en muchos de los casos se podría confundir una señal de ruido o background con un resultado positivo. Esta dificultad técnica podría considerarse una desventaja de esta prueba. Sin embargo, la detección de LT o ST mediante hibridación no deja de ser importante ya que se pueden trabajar muchas cepas y es una herramienta eficaz para el tamiz y la identificación primaria de cepas E. Coli enterotoxigénicas. Esta técnica puede ser mejorada con el uso de la reacción en cadena de la polimerasa (PCR) seleccionando las cepas positivas y buscando finalmente los genes LT o ST mediante PCR.
En conclusión, se tiene una herramienta importante para la identificación y caracterización de cepas de E. Coli enterotoxigénica, que servirá para la vigilancia epidemiológica de las diarreas producidas por ECET en el Perú, repotenciando el sistema convencional como la serología.
AGRADECIMIENTO
Agradecemos la colaboración técnica de la Sra. Ana Meza López, Sr. Adrián Gómez y Sr. Gustavo Bellido.
REFERENCIAS
1.Nataro JP, Kaper JB. Diarrhheagenic Escherichia coli. Clin Microbiol Rev 1998; 11(2): 403. [ Links ]
2. Nirdnoy W, Serichantalergs O, Cravioto A, LeBron C, Wolf M, Hoge CW, et al. Distribution of colonization factor antigens among enterotoxigenic Escherichia coli strains isolated from patients with diarrhea in Nepal, Indonesia, Peru and Thailand. J Clin Microbiol 1997; 35: 527-30. [ Links ]
3. Pazzaglia G, Podgore J, Mercado W, Martínez A, Urteaga A, Echevarry E. The etiology of childhood diarrhea in northern coastal Peru: the 1989 Fuerzas Unidas Humanitarian Civic Actiona model for international and interservice cooperation, community service, and scientific opportunity. Mil Med 1991; 156: 402-5. [ Links ]
4. Sarinho SW, da Silva GA, Magalhaes M, Carvalho MR. A study of the importance of the toxigenic E. coli in children with acute diarrhoea in Recife, Brazil. J Trop Pediatr 1993; 39: 304-6. [ Links ]
5. Saelzer E, Munoz P, Pena A, Tellerias L, Fernández A, Giglio M, et al. Bacterial isolation in infants hospitalized for acute diarrhea. Rev Chil Pediatr 1989; 60 (6):328-33. [ Links ]
6. Notario R, Borda N, Gambande T, Sutich E. Species and serovars of enteropathogenic agents associated with acute diarrheal disease in Rosario, Argentina. Rev Inst Med Trop Sao Paulo 1996; 38: 5-7. [ Links ]
7. Vergara M, Quiroga M, Grenon S, Pegels E, Oviedo P,Deschutter J, et al. Prospective study of enteropathogens in two communities of Misiones Argentina. Rev Inst Med Trop Sao Paulo 1996; 38: 337-47. [ Links ]
8. Geyid A, Olsvik O, Ljungh A. Virulence proporties of Escherichia coli isolated form Ethiopian patients with acute or persistent diarrhea. Ethiop Med J 1998; 36: 123-39. [ Links ]
9. Abu-Elyazeed R, Wierzba TF, Mourad AS, Persuki LF, Kay BA, Rao M, et al. Epidemiology of enterotoxigenic Escherichia coli diarrhea in a pediatric cohort in a periurban area of lower Egypt. J Infect Dis 1999; 179: 382-9. [ Links ]
10. Autor del capítulo o de la ponencia. Manual de Procedimientos Parte I: Escherichia coli diarreigénico. Taller Teórico-Práctico. Diagnóstico Molecular de las Enfermedades diarréicas agudas causadas por Vibrio cholerae y Escherichia coli. Buenos Aires 1998. Editores ; año. Pág. inicial y final. [ Links ]
11. Lockwood DE, Robertson DC. Development of a competitive enzyme-linked immunosorbent assay (ELISA) for Escherichia coli heat-stable enterotoxin (STa). J Immunol Methods 1984; 75(31): 295-307. [ Links ]
12. Blanco J, Blanco M, Garabal JI, González EA. Enterotoxins, colonization factors and serotypes of enterotoxigenic Escherichia coli from humans and animals. Microbiología 1991; 7: 57-73. [ Links ]
13. Moseley Sl, Echeverría P, Seriwatana J, Tirapat C, Cahicumpa W, Sakuldaipeara T, et al. Identification of enteritoxigenic Escherichia coli by colony hybridization using three enterotoxin gene probes. J Infect Dis 1982; 145: 863-9. [ Links ]
14. Seriwatana J, Echeverría P, Taylor DN, Sakuldaipeara T, Cahngchawalit S, Chivoratanond O. Identifcation of enterotoxigenic Escherichia coli with syntethic alkaline phosphatse-conjugate oligonucleotide DNA probes. J Clin Microbiol 1987; 25: 1438-41. [ Links ]
15. Schultz C, Pool GJ, Van Ketel R, de Weber B, Speelman P, Dankert J. Detection of enterotoxigenic Escherichia coli in stool samples by using nonradioactively labeled oligonucleotide DNA probes and PCR. J Clin Microbiol 1994; 32: 2393-7. [ Links ]
16. Leong J, Vineland, Dallas W. Nulceotide sequence omparison etween heat-labile toxin B subunit cistron from Escherichia coli of human and porcine oring. Infect Immun 1985; 48: 73 -77. [ Links ]
17. Moseley Sl, Hardy M, Huq I, Echevarría P, Falkow S. Isolation and nucleotide sequence determination of a gene encoding heatstable enterotoxin of Escherichia coli. Infect Immun 1983; 39: 1167-74. [ Links ]
18. Khusmith S, Tharavanij S, Vidulbandhitkit S. Prevalence of enterotoxigenic Escherchia coli in patients with diarrhoea in Bangkok, Southeast Asian. J Trop Med Public Health 1980; 11: 574-5. [ Links ]
19. Jayasheela M, Kumari N, Sandil RK, Saxena SN. Characteristics of Escherichia coli isolates form infantile and childhood diarrhea. Indian J Pediatr. 1989; 56: 87-92. [ Links ]
20. Jung HK. Identification of serotype by use of serologic assay and detection of the enterotoxin gene of Escherichia coli by means of a polymerase chain reaction assay for isolates from pigs, chickens, and cows. Am J Vet Res 1999; 60: 468-72. [ Links ]
21. Begaud E, Mondet D, Germani Y. Molecular characterization of enterotoxigenic Escherichia coli (ETEC) isolated in New Caledonia (value of potential protective antigens in oral vaccine candidates). Res Microbiol 1993; 144: 721-8. [ Links ]
22. Katouli M, Jaafari A, Ketabi GR. The role of diarrheagenic Escherichia coli in acute diarrheal diseases in Bandar-Abbas, Iran. J Med Microbiol 1988; 27: 71-4. [ Links ]
23. Berry RJ, Bettelheim KA, Gracey M. Studies on enterotoxigenic Escherichia coli isolated from persons without diarrhea in Western Australia. J Hyg (Lond) 1983; 90: 99-106. [ Links ]
24. Goldhar J, Gutman R, Peri R. An outbreak (?) caused by Escherichia coli O126:K71:H5 in premature infants. Zentral Bakteriol Mikrobiol Hyg (A) 1981; 250: 470-7. [ Links ]
25. Blanco J, Blanco JE, Blanco M, González EA, Garabal JI, Alonso MP. Toxic and adhesive properties of Escherichia coli strains belonging to classic enteropathogenic serogroups. Acta Microbiol Hung 1993; 40: 335-41. [ Links ]
26. Peruski LF Jr, Kay BA, El-Yazeed RA, El-Etr SH, Cravioto A, Wierzba TF, et al. Phenotypic diversity of enterotoxigenic Escherichia coli strains from a community-based study of pediatric diarrhea in periurban Egypt. J Clin Microbiol 1999; 37: 2974-8. [ Links ]
27. Blanco J, Blanco M, González EA, Blanco JE, Alonso MP, Garabal JI, et al. Serotypes and colonization factors of enterotoxigenic Escherichia coli isolated in various countries. Eur J Epidemiol 1993; 9: 489-96. [ Links ]
28. Thomas LV, Scotland SM, Rowe B. Enterotoxigenic strains of Escherichia coli O128 are not restricted to subgroup ac but also belong to subgroups ab and abc. Infect Immun 1987; 55: 1336-7. [ Links ]
29. Murray BE, Evans DJ Jr, Penaranda ME, Evans DG. CFA/I-ST plasmids: comparison of enterotoxigenic Escherichia coli (ETEC) of serogroups O25, O63, O78, and O128 and mobilization from an R factor-containing epidemic ETEC isolate. J Bacteriol 1983; 153: 566-70. [ Links ]
30. Scotland SM, Day NP, Cravioto A, Thomas LV, Rowe B. Pro-duction of heat-labile or heat-stable enterotoxins by strains of Escherichia coli belonging to serogroups O44, O114, and O128. Infect Immun 1981; 31: 500-3. [ Links ]
31. McConnell MM, Hibberd M, Field AM, Chart H, Rowe B. Characterization of a new putative colonization factor (CS17) from a human enterotoxigenic Escherichia coli of serotype O114:H21 which produces only heat-labile enterotoxin. J Infect Dis 1990; 161: 343-7. [ Links ]
32. Ketyi I, Emody L, Pacsa S, Vertenyi A, Kontrohr T. An altered heart-labile enterotoxin (LT) produced by Escherichia coli serogroup O55 strain. Acta Microbiol Acad Sci Hung 1979; 26: 255-62. [ Links ]
Correspondencia: Isabel Arias Bustamante. Laboratorio de
Enteropatógenos, División de Bacteriología, Instituto Nacional de Salud.
Dirección: Cápac Yupanqui 1400 Jesús María, Lima, Perú.
Telf.: (51–1) 471-9920 Anexo 117. Fax: (51–1) 471-0179.
E-mail: enterop@ins.gob.pe