Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.19 n.4 Lima oct./dic. 2002
COMUNICACIÓN CORTA
Estandarización de la Reacción en cadena de la Polimerasa para la tipificación de Helicobacter pylori
María Zamudio R1; José Huguet T1; Víctor Suárez M1; Cecilia Morón C1; G Vargas C2, César Soriano A3, Oscar Frisancho V3, Alejandro Bussalleu R4.
1 Instituto Nacional de Salud, Lima, Perú.
2 Hospital Arzobispo Loayza – MINSA, Lima, Perú.
3 Hospital Nacional E. Rebagliati – EsSalud, Lima, Perú.
4 Hospital Cayetano Heredia – MINSA, Lima, Perú.
RESUMEN
El presente trabajo presenta avances en la tipificación de aislamientos de Helicobacter pylori mediante la reacción en cadena de la polimerasa (PCR),utilizando como blancos de caracterización al gen de la toxina de vacuolización (Vac) y la isla de patogenicidad (Cag) Los resultados obtenidos a partir de cepas patrones y cepas obtenidas de 17 pacientes con sintomatología asociada a la infección de Helicobacter pylori corroboran los datos de anteriores trabajos al lograrse caracterizar variantes en estas cepas utilizando las regiones de los genes mencionados anteriormente.
Palabras clave: Reacción en cadena de polimerasa; Helicobacter pylori; Técnica de tipificación bacteriana; Perú (fuente: BIREME)
SUMMARY
New achievements related to the typing of Helicobacter pylori through Polymerase Chain Reaction (PCR), using as a target of characterization the toxin associated with vacuolization gene and the pathogenicity island (Cag) are shown in this study. Referential and clinical strains were taken from 17 patients with symptoms of Helicobacter pylori infection and the findings confirmed the results obtained in previous researches, since it was possible to achieve strain typing using these genes.
Key words: Polymerase chain reaction; Helicobacter pylori; Bacterial tyiping techniques; Peru (source: BIREME).
INTRODUCCIÓN
Helicobacter pylori es uno de los agentes infecciosos con mayor prevalencia en el mundo, en donde la mitad de la población se encuentra infectada y actualmente es una de las bacterias más estudiadas por ser el principal agente en el desarrollo de la enfermedad úlcero péptica, gastritis crónica activa y el adenocarcinoma gástrico1.
En el Perú, el cáncer gástrico es la primera causa de mortalidad por neoplasias malignas en el sexo masculino y la tercera causa en mujeres2.
En la actualidad, se ha reportado diversos estudios sobre la tipificación de Helicobacter pylori para demostrar los factores de virulencia. Uno de los métodos más satisfactorios ha sido el análisis molecular de algunos factores: el gen codificante de la toxina de vacuolización VacA y la isla de patogenicidad CagA . Con respecto al gen VacA, se reporta la presencia de polimorfismo en este gen, dividiéndolo en dos secciones: la región señal (s) y la región intermedia (m); ambas regiones poseen variación gen ética, reportándose dos variantes para la región s: 1 y 2 y dos para la m: 1 y 2. Por otro lado, el gen CagA no está presente en todos los H. pylori, generando así variabilidad respecto a su presencia3.
El propósito de este estudio fue validar un sistema de PCR en aislamientos clínicos de H. pylori en el Perú que contribuirá a la tipificación y detección de variantes genéticas de esta bacteria asociadas a la enfermedad gastroduodenal.
MATERIALES Y MÉTODOS
Estudio descriptivo, analítico, que incluyó 17 aislamientos de Helicobacter pylori obtenidos a partir de biopsias gástricas tomadas con endoscopía digestiva en pacientes con gastritis y úlcera péptica en los servicios de gastroenterología de dos hospitales de Lima. Las cepas patrones empleadas como controles fueron: control positivo Helicobacter pylori ATCC 43629 que contiene los genes VacA s1/m2 y CagA negativo 5 y control negativo: Campylobacter jejuni Ga22987.
PREPARACIÓN DEL ADN GENÓMICO DE LOS AISLAMIENTOS DE Helicobacter pylori
Después de realizar los aislamientos primarios de Helicobacter pylori, éste se subcultivó en placas de agar tripticasa soya con 5% de sangre de carnero desfibrinado e incubado por tres a cinco días a 3°C en condiciones de microaerofilia.
Para la extracción del ácido desoxiribonucleíco a partir de los cultivos, se ejecutó el protocolo elaborado según Van Doorn4. Se llevó a cabo la lisis bacteriana mediante la suspensión de los cultivos en buffer tris EDTA y posteriormente incubándose con lisozima 200 mg/mL durante 2 horas a 37°C para romper la pared celular. Seguidamente, se adicionó dodecil sulfato de sodio (SDS) a una concentración final de 0,5% y proteinasa K 200 mg/mL, incubándose a 65°C por dos horas. Con este procedimiento se logra la lisis total de la bacteria. Posteriormente, la solución obtenida fue mezclada con un igual volumen de fenol/cloroformo/alcohol isoamílico para separar las proteínas de los ácidos nucleicos mediante centrifugación a 4500 rpm durante 15 minutos a 4°C. Se recuperó el sobrenadante y finalmente el ADN bacteriano fue precipitado usando alcohol absoluto y resuspendido en agua tridestilada.
REACCIÓN EN CADENA DE LA POLIMERASA PARA LA TIPIFICACIÓN DE LOS GENES VacA y CagA
Los procedimientos para la amplificación de los genes VacA y CagA fueron seguidos según lo indicado en reportes anteriores en los cuales estandarizan y uniformizan los criterios de la reacción en cadena de la polimerasa (PCR)4.
En ese sentido, se realizaron los siguientes pasos: el control de inhibición de muestras de ADN y la amplificación de los genes VacA y CagA.
CONTROL DE INHIBICIÓN DE MUESTRAS DE ADN AMPLIFICACIÓN DEL GEN RIBOSOMAL 16S
Con la finalidad de conocer la condición de la muestra de ADN y no tener falsos negativos al realizar la tipificación de los genes VacA y CagA, se realizó previamente una reacción en cadena de la polimerasa para amplificar in vitro al gen codificante 16S rRNA, cuya secuencia entera de los nucleótidos ya ha sido determinada y es característico de las bacterias.
Los iniciadores utilizados fueron RIBF:
5 TACCTTGTTACG CT T 3 y RIBR 5GGACTAHAGGGTA TCTAA T 3 (H=A, C o T)4,6. El protocolo para la reacción en cadena de la polimerasa fue el siguiente: denaturación inicial a 95°C por 5 minutos, luego 30 ciclos a 95°C por 30 segundos, 33°C por 1,5 minutos y 72°C por 2 minutos. Finalmente, se realizó una extensión final a 72°C por 10 minutos. Toda la reacción se llevó a cabo en un termociclador Genamp 2400.
AMPLIFICACIÓN DE LOS GENES Vac A y Cag A
Para la tipificación de los genes VacA y CagA se utilizaron iniciadores reportados anteriormente4. Para la región señal (s) del gen VacA se utilizaron los iniciadores VA1F: 5ATGGAAATACAACAAACACAC3 y VA1xR: 5CCTGARACCGTTCCTACAGC3. Para la región intermedia (m) del gen VacA se utilizaron los iniciadores HPMGF: 5CACAGCCACTTTCAATAACGA3 y HPMGR : 5CGTC AAAATAATTCCAAGGG3. Para detectar la presencia del gen CagA, se emplearon CagAR: 5CTTCCCTTAATTGCGAGATTCC3 y CagAF: 5TTGACCAACAACCACAAACCG AAG3.
El protocolo de amplificación empleado para la tipificación de los tres marcadores genéticos fue: en un volumen final de 25 μL del tubo de reacción se mezclaron 25 pmol de cada primer específico para cada gen, desoxinucleótidos en concentración final de 200 μM, buffer de reacción (50 Mm KCl, 1,5 mM MgCl 2 , 10 mM Tris-HCl (ph 8,3), 2,5 Mm de MgCl 2 , 1,0 U de AmpliTaq Gold DNA polimerasa (Perkin – Elmer). Para cada muestra se empleó 10 ng de ADN de los cultivos lisados. El progama de temperatura para el PCR comprendió 5 minutos de predenaturación a 94°C, 45 segundos a 50°C y 45 segundos a 72°C y una incubación final a 72°C por 5 minutos. Finalmente, todos los productos de PCR fueron almacenados a 4°C hasta su análisis.
Para la identificación de los productos amplificados, primero 5 μL de la reacción del PCR mezclado con el buffer para muestra con azul de bromo fenol y xilecianol fue analizado por electroforesis de tipo submarino en 1,5% de gel de agarosa y luego se visualizaron mediante la coloración con bromuro de etidio en un transiluminador de luz ultravioleta y fotografiado. El tamaño de los fragmentos de ADN digeridos fue estimado por las distancias de migración del estándar 1 Kb Ladder de peso molecular conocido.
RESULTADOS
El resultado del control de inhibición evidenció la calidad y pureza del ADN de todas las muestras evaluadas que amplificaron el gen universal de las bacterias ARN ribosomal. Todos los aislamientos fueron VacA(+) (Figura Nº01)
La reacción realizada para la fracción del péptido señal (s) dio como resultado un fragmento de 176 pares de bases indicando la variante de tipo s1, que estuvo presente en todos los aislamientos (17/17)
Para la región intermedia m de la toxina Vac se obtuvo fragmentos de 401 y 476 pares de bases para los tipos m1 y m2 encontrándose en igual proporción (50,0%) los genotipos m1 y m2 (Figura Nº.2)
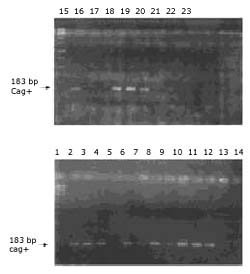
Para el caso de la detección del gen de la isla de patogenicidad cag en cepas de Helicobacter pylori se obtuvo un fragmento de amplificación de aproximadamente 180 bp. En 14/17 de los aislamientos, se detectó el gen CagA y en 3/17 fueron negativos (Figura Nº.3) No se evidenció ningún tipo de amplificación para la cepa de Campylobacter jejuni ni en los controles de sistema con agua como se puede observar en todas las figuras.
Figura N° 1. Detección del gen Vac sección S en Helicobacter pylori.
Carril 1 y 15: marcador de peso molecular 1 kb (VacS1). Carriles 2 al
12 y carriles 16 al 23: VacS1. Carril 24: cepa Helicobacter pylori
ATCC. Carril 13 y 25: cepa ATCC Campylobacter, carril 14 y 26
(control del sistema).
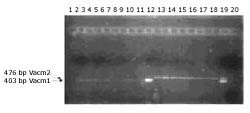
Figura N° 2. Detección del gen Vac región m en Helicobacter pylori.
Carril 1: marcador de peso molecular 1 Kb ladder. Carril 2 – 10: Vac
m tipo 1. Carril 11-17: Vac m tipo 2. Carril 18: Helicobacter pylori
ATCC 43629. Carril 19: variante Vacm1 y Vacm2 en mismo paciente.
Carril 20: cepa ATCC Ga22987 Campylobacter.
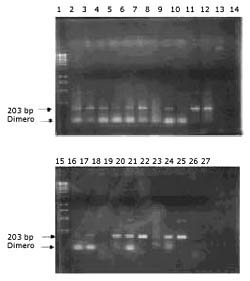
Figura N° 3. Detección del gen Cag en cepas de Helicobacter pylori.
Carril 1 y 15: marcador de peso molecular 1kb. Carriles 2-4, 6-12, 16
y 19-23: Cag (+). Carriles 5, 17 y 18: Cag (-)Carril 24: cepa ATCC de
Helicobacter pylori. Carril 13 y 25: Cepa ATCC de Campylobacter.
Carril 14 y 26: control del sistema.
DISCUSIÓN
En la actualidad, el diagnóstico de Helicobacter pylori puede hacerse mediante una gran variedad de técnicas invasivas y no invasivas, que en general ofrecen un alto grado de sensibilidad y especificidad. El cultivo sigue siendo la prueba de oro, aunque los autores informan que existen algunas factores que limitan el crecimiento de la bacteria 6, 7.
Las técnicas moleculares han causado un gran impacto para el diagnóstico de microorganismos de difícil crecimiento y detección. La reacción en cadena de la polimerasa ha servido para el desarrollo de pruebas específicas. Como era de esperarse, al utilizar esta prueba todos los aislamientos de nuestro estudio fueron VacA(+), confirmándose lo hallado en otros reportes8-11. Asimismo, en todos los aislamientos presentaron la variante s1 al igual que lo publicado en estudios internacionales3, 8,10,12-15. Por otro lado, la obtención del 50% de genotipos m1 y m2 confirma lo encontrado por De Guzmao12, Van Doorn14 y Morales-Espinoza15.
En nuestro país, se han realizado estudios de investigación para detección de este microorganismo con el método Nested -PCR a partir de muestras de placa dental, heces y biopsia gástrica, concluyendo que no se puede emplear como prueba diagnóstica de la infección por Helicobacter pylori 11,17. Ante esta dificultad, se continúan investigando diferentes métodos de identificación y tipificación de Helicobacter pylori usando la tecnología del ADN recombinante. Los resultados encontrados en este estudio de validación concuerdan con los reportes publicados por otros autores referente a las cepas CagA+, que están presentes en poblaciones humanas y circulando en diferentes regiones del mundo9,10,17. El gen cagA es un marcador de la región cag, una isla de patogenicidad que actualmente está en discusión para la producción de enfermedad específica, pero se le reconoce el incremento de virulencia de la bacteria. Con respecto a los genotipos vacA se había planteado hipótesis que tenían implicancias patogénicas. Reportes actuales lo consideran para predecir el status del gen cagA3,9.
La metodología del PCR a partir de cultivos puros de Helicobacter pylori que empleamos nos permite diferenciar y tipificar los diversos genotipos, contribuyendo a la epidemiología molecular de esta bacteria que será de utilidad para el conocimiento de su heterogeneidad.
REFERENCIAS
1. De Idiáquez D, Bussalleau A, Cok J. Nuevos esquemas terapeúticos para el tratamiento de la infección por Helicobacter pylori y evaluación de la reinfección al año postratamiento exitoso. Lima: Universidad Peruana Cayetano Heredia; 2001 p. 98 [ Links ]
2. Rojas A. Susceptibilidad a tres antimicrobianos de cepas de Helicobacter pylori aisladas de pacientes con problemas gástricos provenientes de las Pampas de San Juan de Miraflores, Lima (Tesis para optar el título profesional de Químico Farmaceútico y Bioquímico) Lima: Universidad Inca Garcilaso De la Vega, Facultad de Ciencias Veterinarias y Bioquímica; 1999. [ Links ]
3. Hunt RH, Tyggat GN. Helicobacter pylori basic mechanism to clinical cure. Great Britain: Edit. Kluwer; 2000. p.13-32. [ Links ]
4. Van Doorn L, Figueiredo C, Sanna R, Pena S, Midolo P, Enders K, et al. Expanding allelic diversity of Helicobacter pylori vacA. J Clin Microbiol 1998; 36: 2597-603. [ Links ]
5. Li Ch, Ha T, Chi D, Ferguson D, Jiang Ch, Laffan J. Differentiation of Helicobacter pylori strains directly from gastric biopsy specimens by PCR – based restriction fragment length Polymorphism analysis without culture. J Clin Microbiol 1997; 35: 3021-5. [ Links ]
6. Murray P, Jo B, Pfalles M. Manual of Clinical Microbiology. Sixth Edition; 1995. p. 492-7. [ Links ]
7. Ramírez A. Helicobacter pylori in Perú. Rev Gastroenterol, Peru 1991; 11: 32-38. [ Links ]
8. Gómez J, Elizalde JI, Marco F, Bordas JM, Pique JM, Jiménez de Anta MT. Typification of strains of Helicobacter pylori by the detection of the cagA gene and subtypes of the vacA gene. Enf Infec Microbiol Clin 1999 ; 17: 171-5. [ Links ]
9. Rudi J, Kolb C, Maiwald M, Kuck D, Sieg A, Galle PR, et al. Diversity of Helicobacter pylori vacA and cagA genes and relationship to VacA and CagA protein expression, cytotoxin production, and associated diseases. J Clin Microbiol 1998; 36: 944-8. [ Links ]
10. Alarcón T, Domingo D, Martínez MJ, López-Brea M. CagA gene and vacA alleles in Spanish Helicobacter pylori clinical isolates from patients of different ages. Inmunol Med Microbiol 1999; 24: 215-9. [ Links ]
11. Dunn B, Cohen H, Blaser M. Helicobacter pylori. Clin Microbiol Rev 1997; 10: 720-41. [ Links ]
12. De Gusmao VR, Nogueira E, De Magalhaes DM, Aguiar G, Camargos AM, Ramadan AA, et al. VacA genotypes in Helicobacter pylori strains isolated from chidren with and without duodenal ulcer in Brazil. J Clin Microbiol 2000; 38: 2853-7. [ Links ]
13. Figueiredo C, Van Doorn LJ, Nogueira C, Soares JM, Pinho C, Figueira P, et al. Helicobacter pylori genotypes are associated with clinical outcome in Portuguese patients and show a high prevalence of infections with multiple strains. Scand Gastroenterol 2001; 36: 128-35. [ Links ]
14. Van Doorn LJ, Figueiredo C, Sanna R, Plaisier A, Schneeberger P, de Boer W, et al. Clinical relevance of the cagA, vacA, and iceA status of Helicobacter pylori. Gastroenterology 1998; 115: 58-66. [ Links ]
15. Morales-Espinoza R, Castillo-Rojas G, González-Valencia G, Ponce de León S, Cravioto A, Atherton JC, et al. Colonization of Mexican patients by multiple Helicobacter pylori strains with different vacA and cagA genotypes J Clin Microbiol 1999; 37: 3001-4. [ Links ]
16. Warburton VJ, Everett S, Mapstone NP, Axon AT, Hawkey P, Dixon MF. Clinical and histological associations of cagA and vacA genotypes in Helicobacter pylori gastritis. J Clin Pathol 1998; 51: 55-61. [ Links ]
17. Villaverde I, Gutiérrez R. Helicobacter pylori en muestras de placa dental, biopsia gástrica y heces en pacientes con alteraciones gástricas del Hospital III Félix Torrealva IPSS – Ica (Tesis para optar el título profesional de Biólogo) Ica:Facultad de Ciencias de la Universidad Nacional San Luis Gonzaga; 2000. [ Links ]
Correspondencia: María Zamudio R. División de Bacteriología, Instituto Nacional de Salud.
Dirección: Cápac Yupanqui 1400, Jesús María, Lima 11, Perú.
Telf.: (51–1) 471-9920 Anexo 117. Fax: (51–1) 471-0179.
E-mail: enterop@ins.gob.pe














