Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.20 no.3 Lima July/Sept. 2003
TRABAJOS ORIGINALES
Caracterización molecular de Klebsiella pneumoniae y Enterobacter cloacae productoras de ß-lactamasas de espectro extendido tipo SHV-5 en una unidad de cuidados intensivos neonatal de Lima
Róger Calderón E1; Rosa Sacsaquispe C1; Fernando G. Pasterán2; Marcelo F. Galas2; Javier Soto P3; Juan Riveros Q3; Augusto Valencia R3; Nazario Silva A3; Víctor Suárez M1; Ysabel Montoya P1.
1 Instituto Nacional de Salud. Lima, Perú.
2 Servicio de Antimicrobianos, Instituto de Enfermedades Infecciosas Carlos G Malbrán. Buenos Aires, Argentina.
3 Servicio de Microbiología Clínica HONADOMANI San Bartolomé. Lima, Perú.
RESUMEN
Objetivo: Determinar los mecanismos implicados en la transmisión y resistencia antimicrobiana de aislamientos hospitalarios de Klebsiella pneumoniae y Enterobacter cloacae. Materiales y métodos: Se determinó la diversidad genética de 10 aislamientos bacterianos provenientes de pacientes hospitalizados y muestras ambientales procedentes de una unidad de cuidados intensivos de neonatos de un hospital de Lima utilizando el patrón de banda de ADN ribosomal y plasmídico. Posteriormente, se caracterizó la resistencia antimicrobiana y sus principales factores utilizando electroforesis de punto isoeléctrico, Southern Blotting y PCR. Finalmente se evaluó la capacidad de transferencia de la resistencia mediante ensayos de conjugación bacteriana. Resultados: Todos los aislamientos de K. pneumoniae y E. cloacae presentaron el mismo perfil plasmídico. Los aislamientos de E. cloacae presentaron un mismo patrón genético, por el contrario se encontraron cuatro genotipos distintos de K. pneumoniae altamente relacionados. Todos los aislamientos produjeron ß-lactamasa de especto extendido Tipo SHV-5 transferible a otras especies. Conclusiones: El estudio sugiere que la diseminación de estas bacterias en los neonatos pudo haber sido favorecida por un inadecuado manejo asistencial, una defectuosa conservación de leche para el consumo neonatal y el indiscriminado uso de antibióticos, el cual generó una activa transmisión de genes responsables de la resistencia antimicrobiana. Palabras clave: Klebsiella pneumoniae; Enterobacter cloacae; Resistencia microbiana a las drogas; Unidades de terapia
ABSTRACT
Objective: To elucidate the mechanisms involved in the transmission and antimicrobial resistance of Klebsiella pneumoniae and Enterobacter cloacae nosocomial isolates. Materials and methods: Genetic diversity was determinated for 10 bacterial isolates from hospitalized patients and environmental samples from a neonatal intensive care unit of a Lima hospital, using ribosomal and plasmidic DNA fingerprinting. Afterwards, the antimicrobial resistance was characterized as well as major factors using isofocusing electrophoresis. This was confirmed with Southern Blotting and PCR. Finally the resistance transfer ability was assessed using bacterial conjugation assays. Results: All K. pneumoniae and E. cloacae isolates displayed a unique plasmidic profile. E. cloacae isolates displayed the same genetic profile but, on the contrary, four K. pneumoniae genotypes highly related were found. In addition, it was established that all isolates produced extended spectrum ß-lactamase SHV-5-like transferable to other strains. Conclusions: This study suggests that the bacterial dissemination in neonates can be favored by the inadequate assistance in handling, by defective storage of milk for neonatal use and because of the indiscriminate use of antimicrobial agents, which contributed to the active transmission of genes responsible for antimicrobial resistance. Key words: Klebsiella pneumoniae; Enterobacter cloacae; Microbial drug resistanse; Neonatal intensive care units; nosocomial infections; Hospitals;Perú (source: BIREME)
INTRODUCCIÓN
Klebsiella pneumoniae y Enterobacter cloacae son especies patógenas significativamente asociadas a infecciones intrahospitalarias (IIH) cuyas alternativas terapéuticas cada vez son menores, por ser importantes fuentes de resistencia antibiótica transferible 1,2. En unidades neonatales se han reportado muchos brotes por Klebsiella y están frecuentemente relacionados con colonización extensa en recién nacidos, infecciones sistémicas y muerte 3-6. En tanto, Enterobacter ha cobrado importancia como patógeno en las últimas 3 décadas y se han aislado especies de este género en infecciones del tracto respiratorio, heridas quirúrgicas, tracto urinario, sangre, etc.2
Tanto la dispersión de K. pneumoniae productora de ß-lactamasas de espectro extendido (BLEE) fuera de las unidades de cuidados intensivos (UCI)7 como la diseminación de resistencia plasmídica entre enterobacterias constituyen un gran problema8; por lo que se han ideado estrategias de vigilancia, prevención y control de las infecciones intrahospitalarias.
La producción bacteriana de ß-lactamasas de espectro extendido (BLEEs) es un problema significativo8, dado que afecta la eficacia de los tratamientos antibióticos en los pacientes. La presencia de estas enzimas es la causa más importante de resistencia a los agentes antimicrobianos ß-lactámicos9, las cuales actúan hidrolizando el anillo oxi-imino- aminotiazolil-, inactivando así las penicilinas, monobactams y cefalosporinas de 1º, 2º y 3º generación. Las SHV (SulpHydryl Variable) son ß-lactamasas plasmídicas, aunque en algunas especies son codificadas por genes cromosomales 8-10. Un ejemplo de ello es la enzima SHV1 que es codificada por un gen cromosomal en K. pneumoniae (propia de la especie), pero su espectro de acción es menor, considerada por ello una ß-lactamasa de amplio espectro. La SHV-5, es una enzima plasmídica mutante, que presenta sustituciones en las posiciones 238 Ser ->Gly y 240 Lys ->Glu 8. Tales cambios, así como otros en otras variantes de esta enzima, son responsables del incremento en su espectro de acción, por lo que se consideran a estas enzimas como ß-lactamasas de espectro extendido. La capacidad de transferir la resistencia está asociada a la presencia de plásmidos 11 u otros genes denominados transposones 12 y puede ocurrir entre diversas especies bacterianas. Actualmente en el Perú no tenemos información sobre la real prevalencia de la resistencia antimicrobiana mediada por BLEEs en bacterias gram-negativas, debido a la falta de estudios y la dificultad técnica para su detección. Para ello la investigación epidemiológica es muy importante ya que en el control de las infecciones intrahospitalarias utiliza los estudios microbiológicos permitiendo subtipificar cepas en el reconocimiento de la diversidad clonal de las bacterias patógenas, detectar transmisión cruzada de patógenos intrahospitalarios, determinar las fuentes de infección y reconocer cepas virulentas, entre otras 11,13. La fusión de estrategias epidemiológicas y moleculares contribuyen a la determinación de fuentes infecciosas y la dinámica de transmisión fortaleciendo las medidas de control y prevención. Así, las metodologías moleculares han colaborado desde hace algunos años con el conocimiento de la epidemiología de las infecciones intrahospitalarias 2,11, para lo cual es conveniente combinar la información del perfil de susceptibilidad antimicrobiana, la ribotipificación y el análisis plasmídico como estrategias de investigación. La ribotipificación es una herramienta eficaz que permite una discriminación entre los aislamientos de E. cloacae y K. pneumoniae, dado el nivel de diversidad del locus rrn 14 en estas especies. Por otro lado, el análisis plasmídico es otra herramienta molecular que nos permite determinar, en cierta medida, la existencia de uno o varios factores genéticos que podrían favorecer la diseminación de las especies o variantes circulantes. El objetivo del presente estudio fue determinar la diversidad genética de aislamientos de K. pneumoniae y E. Cloacae productoras de BLEEs aislados de neonatos y recipientes de leche en una UCI neonatal de un hospital de Lima, Perú entre octubre y noviembre del año 2000 y dilucidar el principal mecanismo implicado en la transmisión de la resistencia antimicrobiana. MATERIALES Y MÉTODOS LOCALIZACIÓN DEL ESTUDIO El Hospital Nacional Docente Madre Niño San Bartolomé, está ubicado en Lima Cercado, brinda los servicios de atención médica y quirúrgica dirigido principalmente a pediatría, cirugía y gineco obstetricia, para lo cual cuenta con 251 camas. La Unidad de Cuidados Intensivos (UCI) cuenta con 2 ambientes: UCI Neonatal y UCI infectados, con 4 incubadoras cada una y 3 ventiladores mecánicos. Además, existe una unidad de tratamiento intermedio pediátrica. En la UCI y en la sala de partos se vigila rutinariamente la aparición de infecciones tales como neumonía asociada a ventilación mecánica, endometritis en partos y cesáreas, infecciones de torrente sanguíneo e infección de herida operatoria de cesáreas y hernias. PACIENTES, MUESTRAS CLÍNICAS Y AMBIENTALES Estudio realizado en la UCI del servicio de neonatología del hospital durante los meses de octubre y noviembre del año 2000, en donde ocho (08) neonatos presentaron claros signos de sepsis o neumonía luego de permanecer en el centro obstétrico o en la sala de emergencia del hospital (Tabla 1) y ser trasladados a la UCI. Se realizaron cultivos microbiológicos a cada paciente a partir de sangre y punta de catéter. Como tratamiento empírico inicial fueron administrados aminoglucósidos y ampicilina, y de acuerdo con su evolución se cambió el tratamiento de los pacientes a imipenem y vancomicina. Los pacientes fueron analizados con respecto a su tratamiento antibiótico previo, parámetros clínicos y de laboratorio, coinfecciones y antecedentes de transferencia con otras salas. Además, en las dos salas de UCI se tomaron muestras de preparaciones para alimentación de los neonatos, soluciones antisépticas y de uso parenteral (dextrosa, cloruro de potasio y ephinal) como parte de la investigación epidemiológica en aquellos casos de infecciones intrahospitalarias. Finalmente se usó la siguiente definición de caso: aquella cepa de K. pneumoniae o E. cloacae aislados de los pacientes del servicio de neonatología que tenga un tiempo de hospitalización mayor a 72 horas entre el 15 de octubre y 15 de noviembre del año 2000. AISLAMIENTO, IDENTIFICACIÓN MICROBIOLÓGICA Y SUSCEPTIBILIDAD ANTIMICROBIANA El aislamiento y la identificación inicial de las bacterias fueron realizados con las técnicas bacteriológicas convencionales 15,16 y con el sistema de identificación API 20E (BioMerieux, Francia). El análisis de la susceptibilidad antimicrobiana se realizó mediante las pruebas de sensibilidad por el método de disco de difusión, utilizando los siguientes antibióticos: ampicilina, amoxicilina/ácido clavulánico, piperacilina, piperacilina/tazobactam, cefalotina, cefuroxima, cefoxitina, cefotaxima, cefotaxima/ácido clavulánico, ceftazidima, ceftazidima/ácido clavulánico, aztreonam, cefepime, imipenem, meropenem, amikacina, ciprofloxacina, gentamicina, sulfametoxazol/trimetoprim. La identificación bioquímica de la producción de BLEEs para E. cloacae y K. pneumoniae fue detectada mediante la prueba del sinergismo del doble disco con cefotaxima/ácido clavulánico y ceftazidima/ácido clavulánico 17 según las normas del National Committee for Clinical Laboratory Standards (NCCLS)18. La determinación de las concentraciones inhibitorias mínimas (CIM) a ceftazidima y cefotaxima fue realizada mediante el uso del E-Test (BioMerieux, Francia) evaluando los antibióticos cefotaxima, cefotaxima/ácido clavulánico, ceftazidima, ceftazidima/ácido clavulánico. IDENTIFICACIÓN FENOTÍPICA Y GENOTÍPICA DE ß-LACTAMASAS Las proteínas de membrana externa obtenidas de los lisados crudos de suspensiones bacterianas sometidos a sonicación fueron separadas mediante una electroforesis de isoelectroenfoque en gel de poliacrilamida al 10% (gradiente de pH de 3,5 - 10). Las ß-lactamasas fueron visualizadas mediante su actividad hidrolítica de antibiótico sobre geles de yodo-almidón formando halos incoloros y por comparación con estándares de punto isoeléctrico (pI). Luego, a cada aislamiento se identificó por reacción en cadena de la polimerasa (PCR) específicamente la familia correspondiente a las ß-lactamasas estimadas por PATRÓN DE BANDA PLASMÍDICA Y GENÓMICA El ADN plasmídico fue aislado siguiendo el protocolo descrito por Sambrook y colaboradores 20, en el cual las bacterias fueron sometidas a una lisis con NaOH-SDS y una precipitación con acetato de potasio. Posteriormente, el ADN fue precipitado con etanol y resuspendido en agua estéril. Los plásmidos extraídos fueron evaluados mediante electroforesis en agarosa al 1% y registrados fotográficamente. El análisis de patrón de banda 21 fue realizado por hibridación usando 10 mg de ADN genómico purificado, los cuales fueron digeridos con 50 U de EcoRI. Los fragmentos fueron separados por electroforesis en agarosa al 1%, transferidos a una membrana de nylon (N+) y fijados por acción de luz ultravioleta. La membrana fue incubada con buffer de hibridación (Amersham Pharmacia Biotech) empleando como sonda un fragmento de aproximadamente 1450 pb de ADN ribosomal 16S generado por PCR 14 marcado con peroxidasa usando el sistema quimioluminiscente. Las señales fueron reveladas en una película de autorradiografía y registradas fotográficamente. CONJUGACIÓN BACTERIANA Se realizó un ensayo mediante la mezcla y apareo bacteriano en placas de cultivo usando una cepa sensible de Salmonella sp. como receptora. Como cepas dadoras se seleccionaron al azar 2 aislamientos de K. Pneumonaie y de E. cloacae inoculándolas individualmente en placas de agar tripticasa soya (TSA) e incubadas a 37 ºC durante 18-24 horas. Cada K. pneumoniae y E. cloacae fue mezclada individualmente con Salmonella sp. En cantidades iguales en placas de TSA e incubadas a 37 ºC durante 18-24 horas. Luego de la incubación, las células fueron cosechadas y resuspendidas en agua estéril, haciendo diluciones bacterianas (10 -1 - 10 -6 ). Un volumen de cada dilución fue dispersado en una placa de agar Salmonella-Shigella (SS) conteniendo 4 mg/mL de cefotaxima y luego fueron seleccionadas las colonias de Salmonella sp. transconjugantes en agar SS conteniendo 32 mg/mL de ceftazidima y por la producción de H2S. Para evaluar la transferencia de la resistencia en las bacterias transconjugantes se realizó una prueba de CIM, además de un análisis de hibridación usando una sonda específica para SHV 19, mediante el protocolo de hibridación quimioluminiscente descrito anteriormente. ANÁLISIS DE PERFILES Y CONSTRUCCIÓN DE CLADOGRAMA Los tamaños moleculares de patrones de ADN plasmídico fueron determinados por el uso del software Total Lab 1d from phoretix v.1,0 1996-2000. La relación entre los patrones ribotípicos fue calculada usando el coeficiente de Dice 22 construyendo un árbol cladístico usando el programa Gel Compar II v 2,5 1998, basando el análisis en el algoritmo UPGMA 23, con una tolerancia en la posición de las bandas del 1%. RESULTADOS El primer paciente reportado fue un neonato de 3 días de edad quien presentó una severa dificultad respiratoria, al ingreso a la UCI se le diagnosticó sepsis bacteriana y
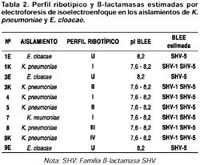
La caracterización microbiana está descrita en la tabla 2. Se identificaron siete cepas de K. pneumoniae y 3 cepas de E. cloacae. Seis aislamientos de K. pneumoniae y dos
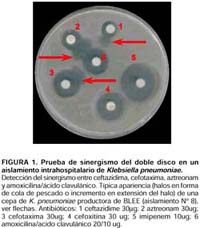
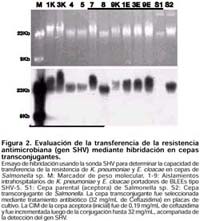
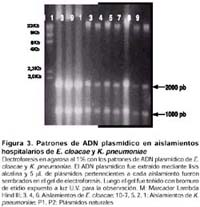
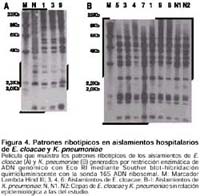
Mediante la prueba de disco difusión, se encontraron que todos los aislamientos de K. pneumoniae y de E. cloacae presentaron patrones similares de susceptibilidad a drogas. La producción de ß-lactamasas de espectro extendido fue determinada en todos los aislamientos mediante el perfil de resistencia a ceftazidima y cefotaxima y la inhibición de estas enzimas mediante la combinación del antibiótico con ácido clavulánico (Figura 1) y confirmada por procedimientos estandarizados. El análisis de isoelectroenfoque demostró que los aislamientos de K. pneumoniae y E. cloacae tuvieron una considerable reactividad ß-lactamasa a pI 8,2, lo cual nos sugiere la posibilidad de encontrarnos frente a una SHV-5. Adicionalmente, sólo los aislamientos de K. Pneumoniae presentaron actividad ß-lactamasa a pI 7.6, la cual corresponde a la ß-lactamasa SHV-1 propia de la especie. La actividad de las ß-lactamasas a pI 8,2 fueron determinadas sobre las cefalosporinas cefotaxima (CTX) y ceftazidima (CAZ) (Tabla 2). Los resultados de actividad a pI 8,2 en E. cloacae fueron confirmados por la reacción en cadena de la polimerada (PCR) para estimar la presencia de la ß-lactamasa SHV específica en los aislamientos. Con el propósito de conocer el nivel de resistencia mediada por ß-lactamasas de espectro extendido se determinó la CIM de CAZ y CTX. En todos los aislamientos hospitalarios el valor para CAZ fue >32mg/mL y para CTX fue >4mg/mL. A través de estos valores se realizaron la selección de las bacterias transconjugantes para determinar la capacidad de transferir la resistencia a CAZ y CTX. Para ello se seleccionaron al azar dos aislamientos de K. pneumoniae (5 y 8) y E. cloacae (1E y 9E) para conjugarse con Salmonella sp. sensible. Sólo se obtuvo un aislamiento de Salmonella transconjugante que recibió el fenotipo resistente a CTX y CAZ, dado que se incrementó el MIC para CTX desde 0,47mg/mL hasta 4mg/mL y de 0,19mg/mL hasta 32mg/mL para CAZ. Para confirmar la transferencia de la resistencia demostrada por la conjugación bacteriana, se ensayó una hibridación del gen SHV usando ADN genómico de la bacteria transconjugante, así como de todos los aislamientos hospitalarios y la bacteria aceptora. Sólo los aislamientos hospitalarios y la Salmonella sp. transconjugante resistente (recibieron los fenotipos resistentes) resultaron positivos (Figura 2). El análisis de patrón de banda plasmídica reveló que todos los aislamientos de K. y Enterobacter presentaron un perfil común, constituido por dos plásmidos de 2000 pb y 1000 pb aproximadamente, como se muestra en la figura 3. No se distinguieron plásmidos de alto peso molecular. La genotipificación de los aislamientos de K. Pneumoniae y E. cloacae se describe en la tabla 1. Los perfiles de ADN ribosomal evaluados en los tres aislamientos de E. cloacae se muestran en la figura 4 A, donde no se observan claras diferencias entre los ribotipos (ribotipo U). Por su parte, los aislamientos de K. Pneumoniae produjeron cuatro patrones ribotípicos distribuidos en los siete aislamientos. El ribotipo I agrupó a los aislamientos tres y cinco. Mientras que el segundo ribotipo (II) agrupó a los aislamientos uno, cuatro y siete. Por último, los aislamientos ocho y nueve presentaron perfiles ribosomales únicos (Figura 4 B). DISCUSIÓN La infección intrahospitalaria por las bacterias entéricas gram-negativas E. cloacae y K. pneumoniae es emergente, particularmente en unidades de cuidados intensivos 1,2. En los últimos años muchos investigadores han reportado muchos brotes homogéneos (uniclonales) y heterogéneos (multiclonales) en los hospitales de todo el mundo, los cuales han sido producidos principalmente por K. pneumoniae 1,7,11,24-29. Mientras tanto, E. cloacae se ha definido como otra de las especies que poco a poco ha cobrado importancia en la epidemiología hospitalaria 2,18,30, lo cual ha sido agravado por la emergencia de la resistencia antimicrobiana y más aún por la posibilidad de su transmisión a otros individuos o especies. Mediante la aplicación de las herramientas moleculares de patrones de banda plasmídico y genómico se determinaron que los aislamientos de E. cloacae de los pacientes 1 y 3 junto al aislamiento 9 procedente de la muestra de leche almacenada en vasos plásticos, son indistinguibles, concordando con la similitud de sus perfiles de sensibilidad antimicrobiana. Sin embargo, la similitud del perfil plasmídico entre E. cloacae y K. Pneumoniae pueden definir a este método como inestable para determinar la asociación entre los aislamientos bacterianos, dada la mayor facilidad de perder o ganar elementos plasmídicos que genómicos 31. El poder discriminativo de la ribotipificación en aislamientos de E. cloacae fue óptimo, al igual que en el trabajo de Hou y colaboradores 31 debido a que los patrones generados con la enzima EcoRI son suficientes para distinguir aislamientos no relacionados epidemiológicamente. De acuerdo al hallazgo se sugiere que una misma cepa de E. cloacae ha sido diseminada en En el caso de K. pneumoniae el análisis genómico mediante el uso de ADN ribosomal tiene un poder discriminativo menor, lo cual pudo ser mejorado utilizando una enzima de restricción adicional 14. En el estudio, se colocaron dos aislamientos no relacionados con el brote resultando diferentes y pudieron funcionar como un control negativo. Tomando en cuenta esto, se encontraron cuatro ribotipos altamente relacionados, distribuidos en las salas de UCI-infectados, Unidad de Tratamiento Intermedio y UCI-neonatal, a pesar que compartían un mismo perfil plasmídico. Esta interrelación entre los aislamientos provenientes de los pacientes se observa de acuerdo con su ligera distancia en la distribución entre sus ribotipos en el árbol cladístico con relación al ribotipo encontrado en los aislamientos no relacionados "N1 y N2". El ribotipo I reúne pacientes UCI-infectados y UCI-neonatología, el cual se muestra altamente relacionado con el ribotipo III perteneciente al aislamiento procedente de la leche de la sala de UCI-infectados. El ribotipo II reúne pacientes de la sala de UCI-infectados y UTI. Un aislamiento con perfil individual perteneció a un único paciente proveniente de la sala UCI-neonatología. Según la aparición de los casos, la transmisión de los microorganismos se inició en la UCI-neonatal; sabiendo que los pacientes de las UCI se encuentran en un elevado riesgo para infecciones intrahospitalarias 11,14,21-24 debido a su estado inmunocomprometido y al continuo uso de procedimientos invasivos 32. Ello tendría concordancia con la aparición del caso de E. cloacae en UCI-neonatología y luego muy probablemente se hayan diseminado hacia la sala de UTI, culminando con la infección de los pacientes en la sala de UCI-infectados junto con la contaminación de los productos alimenticios (leche). La resistencia antimicrobiana caracterizada en estos aislamientos hospitalarios revelaron que en las UCIs de este hospital está circulando un elemento genético de inserción cromosómica (no se han encontrado plásmidos de alto peso molecular) que contendría factores de resistencia a aminoglucósidos y ß-lactámicos. En el análisis de conjugación bacteriana, la resistencia a estos dos tipos de antimicrobianos fue satisfactoriamente transferida a la cepa aceptora. Siguiendo con el análisis se determinó de acuerdo con su punto isoeléctrico que la BLEE codificada por este elemento de inserción cromosómica, puede ser una enzima tipo SHV-5, la que fue encontrada en la cepa aceptora. Esta enzima es altamente prevalente en muchos lugares del mundo, especialmente en Europa y América 33, siendo su transmisión principalmente plasmídica 8,32-33, aunque no se ha descartado su transmisión por otros mecanismos (transposones o integrones). El uso de antimicrobianos de amplio espectro como ß-lactámicos y aminoglucósidos puede controlar la aparición de una infección en un corto tiempo, pero el uso continuo de estos agentes puede favorecer la aparición de pacientes colonizados con bacterias multirresistentes, incrementando los riesgos de adquirir una IIH 32. Por ello la oportuna identificación de las bacterias causantes de las infecciones contribuye esencialmente al control de las infecciones intrahospitalarias. En este caso particular, las infecciones pudieron haber sido favorecidas por un inadecuado manejo asistencial de los pacientes y por la presión selectiva de una inadecuada terapia antimicrobiana, generando la transferencia de elementos genéticos bacterianos que contienen por ejemplo ß-lactamasas de espectro extendido. Se sugiere que la producción de estas enzimas puede ser un factor favorable para la diseminación de las cepas de Klebsiella y Enterobacter procedentes, probablemente, del primer paciente, favorecido también por una incorrecta ventilación mecánica, a pesar de que no todos los pacientes tuvieron acceso a esta última. Las medidas inmediatas tomadas para el control de estas infecciones fueron básicamente el lavado obligatorio de manos antes y después de todo procedimiento asistencial, el uso de vasos descartables con tapa para el almacenamiento de leche, junto con el cambio estricto y periódico de los tubos corrugados del ventilador. Gracias a ello, pudo reducirse la tasa de ataque de infección intrahospitalaria de 36,0% en el mes de noviembre a 0,0% en el mes de diciembre. Royle y colaboradores 34 aseguran que mediante el uso de estrategias simples tales como lavado de manos y capacitación al personal de enfermería se prevendrá eficientemente la aparición de infecciones de bacterias productoras de BLEEs, se demuestra así que un control óptimo de un brote no necesita medidas sofisticadas o costosas. Por lo encontrado se sugiere la posibilidad de un único mecanismo de resistencia en todos los aislamientos, el cual puede estar constituido por la transferencia de genes de integración al genoma. Esta semejanza puede explicarse por la presión selectiva de los antibióticos en las UCIs hospitalarias, que favorece la colonización y la infección 27 produciéndose luego la transmisión de genes y plásmidos que confieren resistencia antibiótica en muchos organismos, y en especial entre enterobacterias. Debido al incremento de la resistencia antimicrobiana relacionado al inadecuado uso de antibióticos de amplio espectro deberían considerarse nuevas políticas de control de antibióticos 30. Muchos métodos prácticos en el control de antibióticos incluyen restricción al uso de agentes particulares, definiendo indicaciones para el uso cíclico de ciertos antibióticos con el fin de limitar la presión selectiva en la flora intrahospitalaria 28,29. Finalmente este estudio muestra la existencia de una población heterogénea de cepas de K. pneumoniae y E. cloacae productoras de la ß-lactamasas de espectro extendido tipo SHV-5, diseminadas en algunos ambientes de un hospital de la ciudad de Lima. Su diseminación puede estar relacionada con la expresión de factores genéticos de resistencia de inserción genómica y favorecida por un tratamiento subóptimo 30. Esto puede ser controlado por métodos estrictos y sencillos tales como el lavado de manos del personal asistencial, el óptimo uso de material descartable, el continuo reemplazo de los tubos corrugados de ventilación y el uso racional de antibióticos. REFERENCIAS 1. Podschun R, Ullmann U. Klebsiella spp. as nosocomial pathogens: epidemiology, taxonomy, typing methods, and pathogenicity factors. Clin Microbiol Rev 1998; 11(4): 589-603. [ Links ] 2. Sanders WE, Sanders CC. Enterobacter spp.: pathogens poised to flourish at the turn of the century. Clin Microbiol Rev 1997; 10(2): 220-41. [ Links ] 3. Hable KA, Matsen JM, Wheeler DJ, Hunt CE, Quie PG. Klebsiella type 33 septicemia in an infant intensive care unit. J Pediatr 1972; 80(6): 920-4. [ Links ] 4. Kayyali MZ, Nicholson DP, Smith IM. A Klebsiela outbreak in a pediatric nursery: emergency action and preventive surveillance. Clin Pediatr 1972; 11(7): 422-6. [ Links ] 5. Hill HR, Hunt CE, Matsen JM. Nosocomial colonization with Klebsiella, type 26, in a neonatal intensive-care unit associated with an outbreak of sepsis, meningitis, and necrotizing enterocolitis. J Pediatr 1974; 85(3): 415-9. [ Links ] 6. Eisenach KD, Reber RM, Eitzman DV, Baer H. Nosocomial infections due to kanamycin-resistant, (R)-factor carrying enteric organisms in an intensive care nursery. Pediatrics 1972; 50(3): 395-402. [ Links ] 7. Rice LB, Willey SH, Papanicolaou GA, Medeiros AA, Eliopoulos GM, Moellering RC, et al. Outbreak of ceftazidime resistance caused by extended-spectrum ß-lactamases at a Massachusetts chronic-care facility. Antimicrob Agents Chemother 1990; 34(11): 2193-9. [ Links ] 8. Bradford PA. Extended-spectrum ß-lactamases in the 21st century: characterization, epidemiology, and detection of this important resistance threat. Clin Microb Rev 2001; 14(4): 933-51. [ Links ] 9. Livermore DM. ß- Lactamases in laboratory and clinical resistance. Clin Microbiol Rev 1995; 8(4): 557-84. [ Links ] 10. Bush K, Jacoby GA y Madeiros A. A functional classification scheme for ß-lactamases and its correlation with molecular structure. Antimicrob Agents Chemother 1995; 39(6): 1211-33. [ Links ] 11. Rutala WA, Weber DJ. Nosocomial infections. Infec Dis Clin North Am 1997; 11: 245-78. [ Links ] 12. Heritage J, Hawkey PM, Tood N, Lewis IJ. Transposition of the gene encoding a TEM-12 extended-spectrum ß-lactamase. Antimicrob Agents Chemother 1992; 36(9): 1981-6. [ Links ] 13. Olive DM, Bean P. Principles and applications of methods for DNA-based typing of microbial organisms J Clin Microbiol 1999; 37(6): 1661-9. [ Links ] 14. Bingen EH, Denamur E, Elion J. Use of ribotyping in epidemiological surveillance of nosocomial outbreaks. Clin Microbiol Rev 1994; 7(3): 311-27. [ Links ] 15. Instituto Nacional de Salud. Manual de procedimientos bacteriológicos en infecciones intrahospitalarias. Lima: INS; 2001. Serie de Normas Técnicas 28. [ Links ] 16. Farmer J. Enterobacteriaceae: Introduction and Identification. En: Murray PR, Baron EJ, Pfaller MA, Tenover FC, Yolken RH. Manual of clinical microbiology, 7th ed. Washington DC: American Society for Microbiology; 1999. p. 442-58. [ Links ] 17. Jarlier V, Nicolas MH, Fournier G, Philippon A. Extended broad-spectrum ß lactamases conferring transferable resistance to newer ß-lactam agents in Enterobacteriaceae: hospital prevalence and susceptibility patterns. Rev Infect Dis 1988; 10(4): 867-878. [ Links ] 18. Ferraro MJ, Craig WA, Dudley MN, Eliopoulus GM, Hecht DW, Hindler J, et al. Performance standards for antimicrobial disk susceptibility test; Approved Standard. 7th ed. National Committee for Clinical Laboratory Standards. 2000 Documents M2-A7 and Supplemental Tables M100-S10 (M2). [ Links ] 19. Arlet G, Philippon A. Construction by polymerase chain reaction and use of intragenic DNA probes for three main types of transferable ß-lactamases (TEM, SHV, CARB). FEMS Microbiol Lett 1991, 66(1): 19-25 Erratum in: FEMS Microbiol Lett 1991; 68(1): 125. [ Links ] 20. Sambrook J, Fritsch EF, Maniatis T. Molecular cloning. A laboratory manual. 2nd ed. New York: Cold Spring Harbor Laboratory Press; 1989. [ Links ] 21. Popovic T, Bopp C, Olsvik O, Kiehbaluch J. Ribotyping in molecular epidemiology, En: Persing DH, Smith TF, Tenover FC, White TJ, editors. Diagnostic molecular microbiology: principles and applications. Washington DC: American Society for Microbiology, 1993. p. 573. [ Links ] 22. Dice LR. Measures of the amount of ecologic association between species. Ecology 1945; 26(3): 297-302. [ Links ] 23. Leitner T, Escanilla D, Franzén, Uhlén M, Albert J. Accurate reconstruction of a known HIV-1 transmission history by phylogenetic tree analysis. Proc Natl Acad Sci USA 1996; 93(20): 10864-9. [ Links ] 24. Arlet G, Sanson-Le Pors MJ, Rouveau M, Fournier G, Marie O, Schlemmer B, et al. Outbreak of nosocomial infections due to Klebsiella pneumoniae producing SHV-4 ß-lactamase. Eur J Clin Microbiol Infect Dis 1990; 9(11): 797-803. [ Links ] 25. Eisen D, Russell EG, Tymms M, Roper EJ, Grayson ML, Turnidge J. Random amplified polymorphic DNA and plasmid analyses used in investigation of an outbreak of multiresistant Klebsiella pneumoniae. J Clin Microbiol 1995; 33(3): 713-7. [ Links ] 26. Rice LB, Eckstein EC, De Vente J, Shlaes DM. Ceftazidime-resistant Klebsiella pneumoniae isolates recovered at the Cleveland Department of Veterans Affairs Medical Center. Clin Infect Dis1996; 23(1): 118-24. [ Links ] 27. Dorsey G, Borneo HT, Sun SJ, Wells J, Steele L, Howland K, et al. A heterogeneous outbreak of Enterobacter cloacae and Serratia marcescens infections in a surgical intensive care unit. Infect Control Hosp Epidemiol 2000; 21(7): 465-9. [ Links ] 28. Sanders WE, Sanders CC. Cycling of antibiotics: an approach to circumvent resistance in specialized units of the hospital. Clin Microbiol Infect 1996; 1(4): 223-5. [ Links ] 29. McGowan J. Do intensive hospital antibiotic control programs prevent the spread of antibiotic resistance? Infect Control Hosp Epidemiol 1994; 15(7): 478-83. [ Links ] 30. Adams BG, Marrie TJ. Hand carriage of aerobic gram-negative rods may not be transient. J Hyg 1982; 89(1): 33-46. [ Links ] 31. Hob ST, Wang CC, Chu ML. Ribotyping and random amplification of polymorphic DNA for nosocomial Enterobacter cloacae isolates in a pediatric intensive-care unit. Infect Control Hosp Epidemiol 1997; 18(11): 769-71. [ Links ] 32. Asensio A, Oliver A, Gonzalez-Diego P, Baquero F, Perez-Diaz JC, Ros P. et al. Outbreak of a multiresistant Klebsiella pneumoniae strain in an intensive care unit: antibiotic use as risk factor for colonization and infection. Clin Infect Dis 2000; 30(1): 55-60. 33. Kim J, Shin HS, Seol SY, Cho DT. Relationship between blaSHV-12 and blaSHV-2a in Korea. J Antimicrob Chemother 2002; 49(2): 261-7. [ Links ] 34. Royle J, Halasz S, Eagles G, Gilbert G, Dalton D, Jelfs P, et al. Outbreak of extended spectrum ß-lactamase producing Klebsiella pneumoniae in a neonatal unit. Arch Dis Child Fetal Neonatal Ed 1999; 80(1): F64-8. [ Links ]