Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.20 no.3 Lima July/Sept. 2003
TRABAJOS ORIGINALES
Genotipificación de aislamientos de Bartonella bacilliformis por amplificación de elementos repetitivos mediante el uso de REP-PCR y ERIC-PCR
Carlos Padilla R 1; Gladis Ventura E 2
1 División de Biología Molecular. Instituto Nacional de Salud, Lima, Perú
2 División de Bacteriología. Instituto Nacional de Salud, Lima, Perú
RESUMEN
Objetivos: Genotipificar los aislamientos de Bartonella bacilliformis a través de la amplificación de elementos repetitivos mediante el uso de ERIC-PCR y REP-PCR, y determinar si existe variabilidad genética entre aislamientos de varias zonas endémicas. Materiales y Métodos: Se evaluaron mediante el uso del ERIC-PCR y REP-PCR 17 aislamientos de B. bacilliformis de Lima, Cusco y Ancash. Los aislamientos fueron realizados durante los años 1998 y 1999. Para el análisis de los patrones de bandas se usó el software GelCompar 4,0. Resultados: Fueron identificados en los 17 aislamientos 10 genotipos. Los genotipos D, E y H fueron detectados en Cusco; mientras que los genotipos B, C, G, J e I en Lima; y el genotipo F en Ancash. Conclusiones: Nuestros resultados sugieren que REP-PCR y ERIC-PCR son métodos útiles para genotipificar aislamientos de B. bacilliformis. La variabilidad genética debe ser tomada en cuenta en estudios epidemiológicos y clínicos de Bartonelosis; así como el desarrollo de nuevas técnicas diagnósticas y de vacunas.
Palabras clave: Bartonella bacilliformis; Reacción en Cadena de la Polimerasa; Infecciones por Bartonella; Genotipo. (fuente: BIREME)
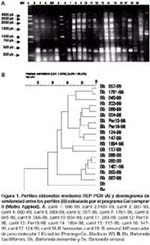
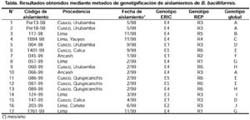
ABSTRACT Objetive: To genotype Bartonella bacilliformis isolates by repeated elements amplification using ERIC-PCR and REP-PCR, and to determine if there is any genetic variability among isolates from several endemic areas. Materials y Methods: 17 B. bacilliformis isolates from Lima, Cusco and Ancash were assessed using ERIC-PCR and REP-PCR. Fingerprintings of the samples were obtained, and they were analyzed using GelCompar 4,0 software. Results: 10 genotypes were identified. Genotypes D, E and H were detected in Cusco; while genotypes B, C, G, J and I were detected in Lima; and genotype F was found in Ancash. Conclusions: Our results indicate that REP-PCR and ERIC-PCR are useful methods for genotyping B. bacilliformis isolates. Genetic polymorphism should be taken into account in epidemiological and clinical studies, as well as for the development of new vaccines and diagnostic methods. Key words: Bartonella bacilliformis, Polymerase Chain Reaction; Bartonella infections; Genotype. (source: BIREME) INTRODUCCIÓN La Bartonelosis es una enfermedad causada por el bacilo gram-negativo Bartonella bacilliformis y transmitida por la picadura del mosquito flebótomo del género Lutzomya 1,2 esendémica en el Perú, Ecuador y Colombia 1-6. En el Perú, los departamentos endémicos para esta enfermedad son: Ancash, Lima, Piura, Cusco, Cajamarca, Amazonas, La Libertad, Huancavelica, Huánuco y Ayacucho 2. Existen dos fases clínicas bien diferenciadas de esta enfermedad: la fase aguda caracterizada por anemia hemolítica y la fase crónica caracterizada por la presencia de verrugas, la primera puede ser mortal si no es tratada a tiempo 1-3. Las herramientas moleculares para la tipificación de aislamientos han demostrado ser muy importantes en el estudio de la epidemiología de las enfermedades infecciosas. Sin embargo, hay pocas metodologías descritas para caracterizar aislamientos de B. bacilliformis. Un reciente informe realizado por Birtles y colaboradores 7 describe el uso del método de polimorfismo de fragmentos amplificados (amplified fragment length polymorphism o AFLP) y secuenciamiento de genes para la identificación de genotipos de B. bacilliformis; sin embargo, estos métodos requieren varias etapas en su procesamiento. La amplificación de elementos genéticos repetitivos usando los sistemas ERIC-PCR (amplificación de secuencias intergénicas de consenso repetitivas de enterobacterias) y REP-PCR (amplificación de elementos palindrómicos extragénicos repetitivos) también han sido descritas para la caracterización de aislamientos del género Bartonella 8-11. Probablemente, al igual que para B. hensenlae; estas técnicas también pueden ser útiles para genotipificar aislamientos de B. bacilliformis 9-11. El presente trabajo tuvo por objetivo genotipificar los aislamientos de B. bacilliformis a través de la amplificación de elementos repetitivos mediante el uso de la ERIC-PCR y REP-PCR y determinar si existe variabilidad genética entre las cepas aisladas de varias zonas endémicas de Bartonelosis en el Perú. MATERIALES Y MÉTODOS AISLAMIENTOS Se evaluaron 17 muestras de B. bacilliformis de Ancash (2), Lima (6) y Cusco (9), las cuales fueron aisladas entre los años 1998 a 1999, como parte del programa de vigilancia de la Bartonelosis (Tabla). Los aislamientos fueron clasificados como B. bacilliformis usando secuenciamiento del gen gltA de la enzima citrato sintetasa según Norman y colaboradores 12. EXTRACCIÓN DE ADN Para la extracción de ADN genómico se usó 200 mL de cultivo bacteriano usando el kit comercial Qiamp Tissue Kit (QIAGEN Inc. Valencia, California.) de acuerdo con las recomendaciones de los fabricantes. Se usó 400 mg de proteinasa K (Sigma Inc. St. Louis, MO) y la solución para lisar los cultivos (ATL) de este kit. Las muestras fueron ERIC-PCR Y REP-PCR En la genotipificación de los aislamientos de B. Bacilliformis se utilizaron las técnicas de REP-PCR y ERIC-PCR. Los oligonucleótidos usados para realizar ERIC-PCR (ERIC1R y ERIC2) y para REP-PCR (Rep1R-Dt y Rep2-D) fueron los mismos que los reportados por Versalovic 13. El volumen final de las mezclas de reacción fue 25 mL, con 200 mM de cada nucleótido trifosfato (A, C, G y T), 2 U de ADN polimerasa termoestable Amplitaq (PE Applied Biosystems, Foster City, California.), 2,5 mM de MgCl 2 y 1 mM de cada oligonucleótido. Se usó 50 ng de ADN genómico purificado Los ciclos de amplificación usados para REP-PCR fueron: una denaturación inicial de 95 ºC por 7 minutos; seguidos de 30 ciclos de 90 ºC por 30 segundos, 43 ºC por 1 minuto, y 65 ºC por 8 minutos, y una extensión final de 65 ºC por 16 minutos. Los ciclos de amplificación usados para ERIC-PCR fueron: una denaturación inicial de 95 ºC por 7 minutos, seguido de 30 ciclos de denaturación a 94 ºC por 1 minuto, temperatura de hibridación de 40 ºC por 1 minuto, y una extensión de 65 ºC por 8 minutos, con una extensión final de 65 °C por 16 minutos. Los productos de amplificación se separaron por electroforesis en geles de agarosa al 1,5%, las condiciones de la electroforesis fueron 75 V por 2 horas en solución TAE 1X, los geles se tiñeron con bromuro de etidio, visualizados en una cámara de luz ultravioleta y fotografiados en una cámara automática MP4+ Polaroid (Polaroid Co. Waltham, MA). Además, para cada experimento se usó como control el ADN genómico purificado de B. hensenlae y B. vinsonii. ANÁLISIS DE LOS PATRONES DE BANDAS Para el análisis de los patrones de bandas obtenidas se usó el software GelCompar 4,0 (Applied Maths, Kortrijk, Belgium). Fueron considerados como un genotipo aquellos aislamientos cuyos perfiles se agruparon en un cluster que presentó una similitud superior a 75%. RESULTADOS Se obtuvieron perfiles o patrones de banda (fingerprints) usando REP-PCR y ERIC-PCR de los 17 aislamientos de B. bacilliformis. Cada perfil presentó múltiples bandas de amplificación. Usando REP-PCR se obtuvieron bandas con tamaños entre 500 pb y 3 500 pb, mientras que usando ERIC-PCR las bandas presentaron un tamaño entre 200 Ambos métodos presentaron alto grado de resolución entre los aislamientos. Los aislamientos de B. bacilliformis fueron agrupados en grupos o clusters importantes y las cepas dentro de estos clusters presentaban un índice de similitud mayor a 75%. Las cepas de B. hensenlae y B. vinsonii que fueron usadas como controles se ubicaron fuera del grupo de cepas de B. bacilliformis (Figuras 1 y 2). Para clasificar mejor los aislamientos, los resultados tanto del ERIC-PCR como del REP-PCR se combinaron de la misma manera como se ha reportado para aislamientos
DISCUSIÓN El análisis de variabilidad genética usando los marcadores REP-PCR y ERIC-PCR han sido útiles para genotipificar varios géneros de bacterias 14-21, siendo aplicados con éxito para caracterizar aislamientos de B. hensenlae 8-10. Los patrones de bandas obtenidos con REP-PCR presentaron mayor resolución (generaron más bandas) que aquellos obtenidos con ERIC-PCR, esto también fue Se pudo observar una gran variedad genética entre aislamientos de B. bacilliformis, pudiendo definirse 10 genotipos, los cuales se distribuyen en diversas áreas endémicas de Bartonelosis: los genotipos A, D, E y H en Cusco; los genotipos B, C, G, J y I en Lima; y los genotipos A y F en Ancash. Sin embargo, debido al pequeño número de aislamientos analizados, no es posible determinar que sólo existan 10 genotipos de B. bacilliformis en el Perú, ni medir asociaciones entre los aislamientos y los datos epidemiológicos. Pero sí es posible determinar que durante el brote de Urubamba de 1998 circuló el genotipo A; mientras que los genotipos D, E y H estuvieron presentes en Cusco en 1999. Aunque se ha observado diferencias en las manifestaciones clínicas de los casos en Cusco con respecto a otras zonas endémicas como Ancash o Lima 6, no existe evidencia suficiente de que este fenómeno sea causado por diferencias en los aislamientos analizados de estas zonas, probablemente, estas manifestaciones clínicas sean explicadas por múltiples factores como el polimorfismo de los aislamientos, la variabilidad genética del vector y estado inmunológico de los pobladores peruanos. La variabilidad genética de aislamientos de B. Bacilliformis debe ser tomada en cuenta en futuros trabajos clínicos y epidemiológicos, en el desarrollo de nuevas técnicas
REFERENCIAS 1. Maguiña C, Gotuzzo E. Bartonellosis. New and Old. Infect Dis Clin North Am 2000; 14(1): 1-22. [ Links ] 2. Ministerio de Salud. Doctrina, normas y procedimientos para el control de la Bartonelosis o enfermedad de Carrión en el Perú. Lima: MINSA; 1998. [ Links ] 3. Maguiña C, Garcia PJ, Gotuzzo E, Cordero L, Spach DH. Bartonellosis (Carrion's disease) in the modern era. Clin Infect Dis 2001; 33(6): 772-9. [ Links ] 4. Cooper P, Guderian R, Paredes W, Daniels R, Perera D, Espinel M, et al. Bartonellosis in Zamora Chinchipe province in Ecuador. Trans R Soc Trop Med Hyg 1996; 90(3): 241-3. [ Links ] 5. Alexander B. A review of bartonellosis in Ecuador and Colombia. Am J Trop Med Hyg 1995; 52(4): 354-9. [ Links ] 6. Ellis BA, Rotz LD, Leake JA, Samalvides F, Bernable J, Ventura G, et al. An outbreak of acute bartonellosis (Oroya fever) in the Urubamba region of Peru, 1998. Am J Trop Med Hyg 1999; 61(2): 344-9. [ Links ] 7. Birtles RJ, Fry NK, Ventosilla P, Caceres AG, Sanchez E, Vizcarra H, et al. Identification of Bartonella bacilliformis genotypes and their relevance to epidemiological investigations of human bartonellosis. J Clin Microbiol 2002; 40(10): 3606-12. [ Links ] 8. Dehio C, Lanz C, Pohl R, Behrens P, Bermond D, Piemont Y, et al. Bartonella schoenbuchii sp. nov., isolated from the blood of wild roedeer. Int J Syst Evol Microbiol 2001; 51(Pt 4): 1557-65. [ Links ] 9. Sander A, Ruess M, Bereswill S, Schuppler M, Steinbrueckner B. Comparison of different DNA fingerprinting techniques for molecular typing of Bartonella hensenlae isolates. J Clin Microbiol 1998; 36 (10): 2973-81. [ Links ] 10. Sander A., Bühler C, Pelz K, von Cramm E, Bredt W. Detection and isolation of two Bartonella hensenlae variants in domestic cats in Germany. J Clin Microbiol 1997; 35(3): 584-7. [ Links ] 11. Rodriguez-Barradas MC, Hamill RJ, Houston ED, Georghiou PR, Clarridge JE, Regnery RL, et al. Genomic fingerpriting of Bartonella species by repetitive element PCR for distinguishing species and isolates. J Clin Microbiol 1995; 33(5): 1089-93. [ Links ] 12. Norman AF, Regnery R, Jamenson P, Greene C, Krause DC. Differentiation of Bartonella-like isolates at the species level by PCR-restriction fragment length polymorphism in the citrate synthase gene. J Clin Microbiol 1995; 33(7): 1797-803. [ Links ] 13. Versalovic J, Koeuth T, Lupski JR. Distribution of repetitive DNA sequences in eubacteria and application to fingerprinting of bacterial genomes. Nucleic Acids Res 1991; 19(24): 6823-31. [ Links ] 14. Wong HC, Lin CH. Evaluation of typing of Vibrio parahaemolyticus by three PCR methods using specific primers. J Clin Microbiol 2001; 39(12):4233-40. [ Links ] 15. Herman L, Heyndrickx M. The presence of intragenically located REP-like elements in Bacillus sporothermodurans is sufficient for REP-PCR typing. Res Microbiol 2000; 151(4):255-61. [ Links ] 16. Wieser M, Busse HJ. Rapid identification of Staphylococcus epidermidis. Int J Syst Evol Microbiol 2000; 50 Pt 3: 1087-93. [ Links ] 17. De la Puente-Redondo VA, del Blanco NG, Gutierrez-Martin CB, Garcia-Pena FJ, Rodriguez-Ferri EF. Comparison of different PCR approaches for typing of Francisella tularensis strains. J Clin Microbiol 2000; 38(3): 1016-22. [ Links ] 18. Jersek B, Gilot P, Gubina M, Klun N, Mehle J, Tcherneva E, et al. Typing of Listeria monocytogenes strains by repetitive element sequence-based PCR. J Clin Microbiol 1999; 37(1):103-9. [ Links ] 19. Appuhamy S, Coote JG, Low JC, Parton R. PCR methods for rapid identification and characterization of Actinobacillus seminis strains. J Clin Microbiol 1998; 36(3): 814-7. [ Links ] 20. Appuhamy S, Parton R, Coote JG, Gibbs HA. Genomic fingerprinting of Haemophilus somnus by a combination of PCR methods. J Clin Microbiol 1997; 35(1): 288-91. [ Links ] 21. Tcherneva E, Rijpens N, Naydensky C, Herman L. Repetitive element sequence based polymerase chain reaction for typing of Brucella strains. Vet Microbiol 1996; 51(1-2): 169-78. [ Links ] AGRADECIMIENTOS Agradecemos la ayuda y cooperación del Dr. José Bernable, de la Bióloga Karina Jaramillo y de la Bióloga Antonia Calvo por la obtención de las muestras para este estudio y también al Dr. Angel Rosas Aguirre.