Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.21 no.2 Lima Apr./jun. 2004
TRABAJOS ORIGINALES
Variabilidad genética en cepas de Sporothrix schenckii aisladas en Abancay, Perú Susan Holechek 1; José Casquero C 2; Susana Zurita M 2; Jorge Guevara C 3; Ysabel Montoya P 1,4 1 Laboratorio de Biología Molecular, Centro Nacional de Salud Pública, Instituto Nacional de Salud. Lima, Perú. RESUMEN Objetivo: Identificar los genotipos de S. schenckii que circulan en 2 distritos de la provincia de Abancay, Perú.
2 Laboratorio de Micología, Centro Nacional de Salud Pública, Instituto Nacional de Salud. Lima, Perú.
3 Oficina de Epidemiología, DISA Abancay. Abancay, Perú.
4 Instituto Peruano de Energía Nuclear. Lima, Perú.
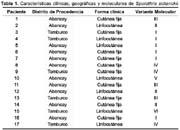
Palabras clave: Sporothrix; Genotipo; Técnica del ADN Polimorfo Amplificado Aleatorio; Peru. (fuente: BIREME). ABSTRACT Objective: To identify S. schenckii genotypes circulating in 2 districts from the Abancay province in Peru. Key words: Sporothrix; Genotype; Random Amplified Polymorphic DNA Technique; Peru (source: BIREME). INTRODUCCIÓN La esporotricosis es una infección subcutánea causada por el hongo dimórfico Sporothrix schenckii que usualmente existe como saprofito en asociación con material orgánico como la vegetación descompuesta, el suelo o el heno. La piel del hospedero que sufre un trauma puede permitir el ingreso de las conidias del hongo, que habitan en el ambiente, y luego recorrer ascendentemente los canales linfáticos, empezando a desarrollar lesiones granulomatosas a lo largo de este canal, las cuales, luego, se ulceran y drenan, siendo la principal manifestación el tipo linfocutáneo 1-3. Mediante técnicas moleculares se ha demostrado que otras formas de inocular el micelio son por picadura de mosquitos, y probablemente, por arañazo de gato 4. La provincia de Abancay, valle altoandino ubicado al centro sur del Perú (Figura 1), tiene una altura de 2750 msnm y es considerada como hiperendémica, y donde la población más afectada es la de niños de 0 a 15 años, con una incidencia anual de 156 casos/100 000 personas 5. La información nacional se refiere a reporte de casos, tratamiento y epidemiología de la enfermedad 2, 5-11, careciendo de datos acerca de los genotipos de S. schenckii que circulan en áreas adyacentes a la provincia de Abancay. Los métodos convencionales identifican limitadamente a los hongos en base a caracteres macroscópicos, microscópicos, bioquímicos y serológicos, lo cual no permite distinguir diferencias entre las cepas 12, 13. Para ello, las herramientas moleculares surgen como una opción para identificar estas diferencias, con implicancias diagnósticas y epidemiológicas 22. La literatura internacional indica que la tipificación de cepas de S. schenckii de diferentes zonas geográficas se basa en el empleo de Restriction Fragment Length Polymorphism (RFLP) en ADN mitocondrial (ADNmt), Random Amplified of Polymorphic DNA (RAPD) y Cariotipificación 13 - 21. El presente trabajo es el primer estudio que tiene como finalidad identificar los genotipos de S. schenckii que circulan en 2 distritos de la provincia de Abancay, mediante la tipificación del ADN genómico. MATERIAL Y MÉTODOS Se evaluaron 17 cepas halladas en pacientes con esporotricosis linfocutánea y cutánea fija diagnosticados entre los meses de junio y septiembre de 1998, procedentes de los distritos de Abancay y Tamburco (provincia de Abancay, departamento de Apurímac). Los aislamientos se realizaron en los centros de salud de Santa Teresa, Tamburco y Hospital Regional de Abancay, como parte de un estudio de factores de riesgo para adquirir esta enfermedad. Posteriormente éstas fueron remitidas al Laboratorio de Micología del Instituto Nacional de Salud, donde fueron mantenidas en caldo infusión cerebro corazón con glicerina al 30% y almacenadas a 20°C. Las cepas fueron identificadas evaluando las características macromorfológicas y microscópicas, posteriormente llevadas al subcultivo sobre agar papa dextrosa e incubación a 28°C por 15 días, para, finalmente, demostrar el dimorfismo térmico a 37°C y temperatura ambiente por 3 y 7 días, respectivamente. Luego, las cepas fueron incubadas en caldo cerebro corazón con agitación por 72 horas a 35°C para obtener concentraciones < 1 x 10 6 células/mL. El ADN genómico fue extraído empleando zimolasa (ICN) a una concentración final de 1000 U/mL y purificado utilizando el kit Genomic - tip 20/G (QIAGEN ®), según las recomendaciones proporcionadas por el fabricante 23. La técnica de RAPD - PCR fue realizada en un volumen final de 25 L, la mezcla contenía 10 ng de la muestra de ADN requerida, 2 mM MgCl 2, 200 M de cada dATP, dTTP, dCTP, y dGTP (Perkin Elmer ®); 0.2 M del oligonucleótido GTG 5 (GTG GTG GTG GTG GTG), agua destilada y esterilizada para completar el volumen final y 1 U de la enzima Taq Gold Polimerasa (Perkin Elmer ®). Las condiciones de amplificación óptima fueron hot start (94°C por 5 minutos), desnaturalización (94°C por 20 segundos), hibridación (50°C por 50 segundos), extensión (72°C por 20 segundos) y, extensión final (72°C por 6 minutos). Las reacciones de PCR fueron mantenidas a 4°C. Las bandas de amplificación fueron observadas mediante electroforesis en un gel de agarosa 1,5%, teñidas con bromuro de etidio y visualizadas mediante rayos ultravioletas. El RAPD-PCR fue repetido tres veces independientemente y los patrones fueron detectados consistentemente en cada vez. Los genotipos fueron asignados basados en la presencia y número de bandas observadas en el gel de agarosa. RESULTADOS De las 17 cepas estudiadas, 7 correspondieron a lesiones linfocutáneas (41,2%) y 10 a aislamientos de la forma cutánea fija (58,8%) (Tabla 1). Se identificaron seis genotipos y se denominaron como I, II, III, IV, V y VI. El genotipo I fue el predominante en 6 aislamientos (pacientes 3 al 7) mostrando 4 bandas de ADN con un tamaño que oscila entre los 4 400 pb y 10 000 pb. Los genotipos II (pacientes 2, 12, 14), III (pacientes 1, 11, 13) y IV (pacientes 8, 9, 17) generaron 5, 2 y 3 bandas que oscilaron entre los 2 300 pb y 10 000 pb. Los genotipos V (paciente 10) y VI (paciente 15) presentaron 5 y 3 bandas entre los 2 200 pb y 10 000 pb, respectivamente. Los genotipos II y V fueron los que presentaron mayor cantidad de bandas, mientras que el genotipo III generó el menor número de bandas. Existen fragmentos de ADN conservados en todas las variantes cuyos rangos oscilan entre los 6500 pb y 4 400 pb (Figura 2). No se logró asociar los genotipos obtenidos con caracteres clínicos y geográficos (Tabla 1). DISCUSIÓN Son muy escasas las investigaciones en el país sobre la epidemiología molecular de S. schenckii. Los resultados encontrados en este estudio demuestran la presencia de seis variantes genéticas en el valle de Abancay mediante la técnica de RAPD-PCR. Diversos investigadores han genotipificado S. schenckii basados en el análisis del perfil de restricción del ADNmt distinguiendo 2 grandes grupos filogenéticos a partir del análisis de 30 tipos de ADNmt (1 - 30) sugiriendo que las cepas procedentes de América Central, Sudáfrica, Norte y Sur América pertenecen al grupo A y las que provienen de Australia, China, Japón corresponden al grupo B 14-19. Otros autores han empleado el RAPD y Cariotipificación para relacionar diferentes tipos clínicos y áreas geográficas con genotipos 13, 20, 21. Nuestros hallazgos indican que existe variabilidad genética en cepas de S. schenckii aisladas en 2 distritos colindantes de la zona hiperendémica de Abancay. Recientemente Neyra et al.24 estudiaron 25 cepas de S. schenckii procedentes de Amazonas (2), Apurímac (9), Ayacucho (1), Cajamarca (3), La Libertad (1), Lima (6), Loreto (2) y Puno (1) señalando que existen 2 grandes grupos genéticos, además que las cepas de Apurímac no predominan en ningún grupo, por lo que en ambas investigaciones se coincide en la biodiversidad genética de este hongo y la dificultad de correlacionar los genotipos obtenidos con los orígenes geográficos de los aislamientos y formas clínicas. Sin embargo, Mesa-Arango et al.13 asocian caracteres fenotípicos con diferentes orígenes geográficos de los aislamientos de S. schenckii, además, indican que esta variabilidad genética puede ser ocasionada por un tipo de recombinación no meiótica en un ciclo parasexual o por la presencia de mutaciones. A partir de este estudio se podrán realizar futuras investigaciones con un mayor número de cepas para conocer la dinámica poblacional y la tasa de variabilidad de S. schenckii. Los nuevos conocimientos acerca del genoma de este hongo permitirán diseñar oligonucleótidos con fines diagnósticos. REFERENCIAS BIBLIOGRÁFICAS 1. Rippon JW. Micología Médica. Hongos y Actinomicetos Patógenos. 3ª ed. México: Editorial Interamericana.1990. [ Links ] 2. Bustamante B, Campos PE. Endemic Sporotrichosis. Curr Opin Infect. Dis 2001; 14(2): 145-49. [ Links ] 3. Kotton, C. Esporotricosis. MedlinePlus Bethesda: Enciclopedia Médica. VeriMed Healthcare Network [Homepage en Internet]. [Actualizado 9 Mar 2001; fecha de acceso: 5 Ago. 2003], aprox. 1 pantalla. Disponible en: http://www.nlm.nih.gov/medlineplus/spanish/ency/article/001338.htm [ Links ] 4. Reiss RS, Schubach TMP, Guimaraes AJ, Monteiro PCF, Zancopé Oliveira RM. Molecular typing of Sporothrix schenckii strains isolated from clinical specimens in Rio de Janeiro, Brazil (Abstract). En: 14 th Congress International Society Human and Animal Mycology. Helsinki: ISHAM; 2000. p.133. [ Links ] 5. Lyon GM, Zurita S, Casquero J, Holgado W, Guevara J, Brandt ME, et al. Population-Based surveillance and a case-control study of risk factors for endemic lymphocutaneous sporotrichosis in Peru. Clin Infect Dis 2003; 36(1): 34-39. [ Links ] 6. Cabezas C, Bustamante B, Holgado W, Begue R. Treatment of cutaneous sporotrichosis with one daily dose of potassium iodide. Pediatr Infect Dis J. 1996; 15(4): 352 - 54. [ Links ] 7. Pappas PG, Tellez I, Deep AE, Nolasco D, Holgado W, Bustamante B. Sporotrichosis in Peru: description of an area of hyperendemicity. Clin Infect Dis 2000; 30(1): 65 - 70. [ Links ] 8. Zurita S, Loayza I, Casquero J. Valle endémico de Esporotricosis. Abancay, Perú. Libro de resumen del V Congreso Latinoamericano de Enfermedades Tropicales. La Habana, Cuba. 1997. [ Links ] 9. Burstein Z. Contribución al estudio de las micosis profundas en el Perú; acerca de tres casos de Sporotrichosis. Jornada de Microbiología y Parasitología. Rev Soc Per Derma 1967; 1: 61 - 77. [ Links ] 10. Burstein Z. Nuevas contribuciones al conocimiento de la Sporotrichosis en el Perú; formas clínicas poco frecuentes. Rev Soc Per Derma 191; 145-55. [ Links ] 11. Burstein Z. Aporte al diagnóstico de las micosis humanas en el Perú. [Tesis doctoral]. Lima: Universidad Nacional Mayor de San Marcos; 1970. [ Links ] 12. McEwen JG, Taylor JW, Carter D, Xu J, Felipe MS, Vilgalys R, et al. Molecular typing of pathogenic fungi. Med Mycol 2000; 38 (Suppl): 189-97. [ Links ] 13. Mesa-Arango AC, Del Rocío Reyes-Montes M, Pérez-Mejía A, Navarro-Barranco H, Souza V, Zúñiga G, et al. Phenotyping and genotyping of Sporothrix schenckii isolates according to geographic origin and clinical form of sporotrichosis. J Clin Microbiol 2002; 40(8): 3004 - 11. [ Links ] 14. Mora-Cabrera M, Alonso RA, Ulloa-Arvizu R, Torres Guerrero H. Analysis of restriction profiles of mitochondrial DNA from Sporothrix schenckii. Med Mycol 2001; 39(5): 439 - 44. [ Links ] 15. Ishizaki H, Kawasaki M, Aoki M, Vismer H, Muir D. Mitochondrial DNA analysis of Sporothrix schenckii in South Africa and Australia. Med Mycol 2000; 38(6): 433 - 36. [ Links ] 16. Ishizaki H, Kawasaki M, Aoki M, Matsumoto T, Padhye AA, Mendoza M, Negroni R. Mitochondrial DNA analysis of Sporothrix schenckii in North and South America. Mycopathologia 1998; 142 (3): 115-18. [ Links ] 17. Ishizaki H, Kawasaki M. Molecular epidemiology of Sporothrix schenckii. Nippon Ishinkin Gakkai Zasshi 2000; 41(4): 245 - 49. [ Links ] 18. Lin J, Kawasaki M, Aoki M, Ishizaki H, You G, Li R. Mitocondrial DNA analysis of Sporothrix schenckii clinical isolates from China. Mycopathologia 1999; 148 (2): 69-72. [ Links ] 19. Ishizaki H, Kawasaki M, Aoki M, Miyaji M, Nishimura K, García Fernández J. A. Mitochondrial DNA analysis of Sporothrix schenckii in Costa Rica. J Med Vet Mycol 1996; 34(1): 71-73. [ Links ] 20. Liu X, Lian C, Jin L, An L, Yang G, Lin X. Characterization of Sporothrix schenckii by random amplification of polymorphic DNA assay. Chin Med J (Engl) 2003; 116(2): 239-42. [ Links ] 21. Tateishi T, Murayama SY, Otsuka F, Yamaguchi H. Kariotyping by PFGE of clinical isolates of Sporothrix schenckii. FEMS Immunol Med Microbiol 1996; 13(2): 147-54. [ Links ] 22. Cooper CR Jr, Breslin BJ, Dixon DM, Salkin IF. DNA Typing of Isolates Associated with the 1988 sporotrichosis epidemic. J Clin Microbiol 1992; 30(7): 1631 - 35. [ Links ] 23. QIAGEN GmbH. Genomic DNA Handbook for blood, cultured cells, tissue, mouse tails, yeast, bacteria (gram - and some Gram +). Germany: Qiagen GmbH, Hilden; 1999. [ Links ] 24. Neyra ER, Velando R, Bustamante B. Patrones genéticos de aislamientos peruanos de Sporothrix schenckii. IV Congreso Virtual de Micología "Hongos Patógenos en América Latina" http://congresomicoloqia.ucv.ve Junio 27 - Julio 4, 2003 [ Links ] Correspondencia: José Casquero. Laboratorio de Micología, Centro Nacional de Salud Pública. Instituto Nacional de Salud. Lima, Perú.
Dirección: Cápac Yupanqui 1400. Jesús Maria. Lima, Perú.
Teléfono: (511) 471-9920 anexo 140.
Correo electrónico: micolog@ins.gob.pe